El microARN-18a de los macrófagos M2 inhibe TGFBR3 para promover la progresión del carcinoma nasofaríngeo y el crecimiento tumoral a través de la vía de señalización de TGF-β
Resumen
Objetivos
El carcinoma nasofaríngeo (CPN) es un tipo de enfermedad nasofaríngea con altas propiedades de metástasis e invasión. Se ha demostrado que los macrófagos activados alternativamente asociados a tumores (M2) se conectan con NPC. Con base en esto, este estudio tiene como objetivo explorar el mecanismo y la participación del microARN-18a (miR-18a) de los macrófagos M2 en NPC.
Métodos
Las células mononucleares de sangre periférica se diferenciaron en macrófagos y los macrófagos se polarizaron al tipo M2 mediante interleucina-4. Las células SUNE-1 y CNE2 se transfectaron con miR-18a restaurado o empobrecido o con el receptor del factor de crecimiento transformante-beta III (TGFBR3) para explorar sus funciones en la progresión de NPC con la participación de la vía de señalización del TGF-β. A continuación, las células SUNE-1 y CNE2 se cultivaron conjuntamente con macrófagos M2 que habían sido tratados con miR-18a o TGFBR3 restaurado o empobrecido para comprender sus funciones combinadas en NPC con la participación de la vía de señalización TGF-β.
Resultados
MiR-18a se expresó altamente y TGFBR3 se expresó escasamente en células NPC. La restauración de MiR-18a, la eliminación de TGFBR3 o el cocultivo con imitadores de miR-18a o los macrófagos M2 transfectados con si-TGFBR3 promovieron la progresión de las células SUNE-1, el crecimiento tumoral en ratones, la disminución de p-Smad1 / t-Smad1 y la elevación de p- Smad3 / t-Smad3. La regulación a la baja de miR-18a, la sobreexpresión de TGFBR3 o el cultivo conjunto con inhibidores de miR-18a o macrófagos M2 transfectados con OE-TGFBR3 deprimieron la progresión de las células CNE2, el crecimiento tumoral en ratones, el aumento de p-Smad1 / t-Smad1 y la disminución de p-Smad3 / t-Smad3.
Conclusión
Nuestro estudio aclara que el miR-18a de los macrófagos M2 da como resultado la progresión de las células NPC y el crecimiento tumoral promovidos en ratones desnudos mediante la represión de TGFBR3, junto con la inactivación de Smad1 y la activación de Smad3.
Introducción
El carcinoma nasofaríngeo (CPN) es un tumor maligno epitelial que tiende a la infiltración local y la metástasis temprana a distancia [1]. Los pacientes con NPC a menudo se quejan de parálisis del VI par y síndrome de Horner [2]. En la actualidad, el tratamiento aplicado consiste predominantemente en radioterapia y radioterapia y quimioterapia integradas [3]. Desafortunadamente, la radioterapia y la quimioterapia se acompañan inesperadamente de complicaciones, y la resistencia adquirida a la radioterapia dificulta los resultados de la NPC [3]. Dado que la tarea de profundizar en posibles terapias dirigidas es una prioridad.
Se ha documentado que los microARN desregulados (miARN) están implicados en la tumorigénesis, metástasis, invasión y resistencia a la radioterapia y la quimioterapia de las NPC [1]. Como una subfamilia de miARN, miR-18a facilita la progresión de NPC a través del supresor de la morfogénesis en la inhibición de los genitales 1 y la activación de la vía mTOR [4]. Además de eso, se verifica además que miR-18a activa la proliferación de células NPC y la metástasis a través de la regulación DICER1 [5]. Además, se ha demostrado que la progresión de las células NPC está impulsada por miR-18a a través del deterioro de la biogénesis del miARN [6]. Además, se ha documentado que miR-18a funciona de manera crítica en la metástasis de NPC [7]. Los macrófagos activados alternativos (M2) son componentes importantes de las neoplasias malignas sólidas y hematológicas y están relacionados con la progresión, la metástasis y la resistencia a la terapia [8]. Los macrófagos asociados a tumores polarizados M2 se asocian con un pronóstico precario de NPC [9]. Curiosamente se registran las diferencias de macrófagos M2 en NPC negativo al virus de Epstein-Barr y positivo al virus de Epstein-Barr [10]. Hay un estudio que destaca que miR-18a inhibe la metástasis hepática de las células de cáncer de colon al inducir macrófagos M1 [11]. El receptor del factor de crecimiento transformante beta III (TGFBR3) es un correceptor de TGF-β que proporciona el ligando del receptor de TGF-β tipo II para estimular la señalización y el equilibrio de la superficie celular, y el TGFBR3 soluble es un regulador durante la progresión del cáncer [12]. Se informa que TGFBR3 de baja expresión induce un microambiente tumoral inmunotolerante [13]. Por el contrario, la sobreexpresión transitoria de TGFBR3 induce la apoptosis en células NPC humanas [14]. Hasta donde sabemos, la polarización de macrófagos M2 inducida por miR-181a avanza la metástasis de células tumorales mediada por macrófagos M2 a través del factor 6 similar a Kruppel y el eje α de la proteína de unión CCAAT / potenciador [15].
En conjunto, aunque muchos estudios han descubierto el papel independiente de los macrófagos miR-18a, TGFBR3 y M2 en NPC, las interacciones combinadas entre estos tres factores siguen siendo esquivas. Considerando que este estudio se lanza para descifrar los mecanismos y participación de estos factores en NPC.
Materiales y métodos
Declaración de ética
El experimento fue aprobado por el comité de ética del Third Xiangya Hospital, Central South University y cumplió con los estándares de ética médica. Este estudio se realizó con el consentimiento por escrito de todos los donantes. Los experimentos con animales cumplieron con los requisitos de las regulaciones nacionales de manejo y uso de animales de laboratorio.
Recolección de células mononucleares de sangre periférica
Los monocitos se obtuvieron de sangre periférica de donantes sanos mediante un método adherente. Se obtuvieron muestras de sangre periférica de donantes sanos del Departamento de Hematología del Third Xiangya Hospital, Central South University. Los macrófagos derivados de monocitos se obtuvieron mediante la adherencia plástica de células mononucleares de sangre periférica (PBMC) previamente aisladas mediante centrifugación en gradiente de densidad (Ficoll-Paque, GE Healthcare) a partir de preparaciones de capa leucocitaria de sangre de donantes sanos. Entonces, 2.0 × 10 6 Se cultivaron PBMC en medio Eagle modificado de Dulbecco (DMEM) suplementado con suero humano al 10% (Millipore, Bedford, MA, EE. UU.) Y penicilina / estreptomicina en placas de 12 pocillos (NalgeNunc, NY, EE. UU.). Cuando las PBMC se adhirieron a la pared durante 2-3 h, el sobrenadante y las PBMC suspendidas se eliminaron repetidamente para obtener monocitos adherentes.
Polarización de macrófagos
Se indujo a los monocitos a diferenciarse en macrófagos mediante el factor estimulante de colonias de macrófagos humanos (M-CSF) y se polarizaron en macrófagos M2 mediante interleucina (IL) -4.
Inducción de macrófagos
Los monocitos se cultivaron en suero bovino fetal (FBS) al 20% -DMEM y se añadieron con M-CSF (100 µg, Peprotech, NJ, EE. UU.) Hasta la concentración final de 100 ng / ml. El medio se renovó por la mitad cada 3 días y luego se complementó con 100 ng / mL de M-CSF. Cultivadas hasta el día 7-8, se recolectaron parte de las células y los marcadores de superficie de macrófagos CD68 [16], CD163 [17] y CD206 [18] se probaron mediante un ensayo de inmunofluorescencia [19] para identificar macrófagos.
Polarización de macrófagos
Los macrófagos se polarizaron a macrófagos M2 suplementando 20 ng / ml de IL-4 (Peprotech) al medio de diferenciación durante otras 24 h. Una parte de las muestras de macrófagos M2 se utilizó para la detección de citometría de flujo. Las muestras se dividieron en 3 tubos:el tubo 1 era el mismo lote de macrófagos adherentes sin estimulación con IL-4; los tubos 2 y 3 eran los macrófagos adherentes estimulados por IL-4. Las muestras se diluyeron hasta aproximadamente 10.000 células cuando se cargaron y se agregaron con inmunoglobulina G no específica para bloquear el receptor Fc. Luego, las muestras se agregaron con anticuerpo de isotipo no específico, anticuerpo CD68 marcado con PE y anticuerpo CD163 marcado con PE (ambos de Biolegend, CA, EE. UU.). Se incubaron durante 30 min y se enjuagaron con albúmina de suero bovino al 0,5% en solución salina tamponada con fosfato (PBS), las muestras se centrifugaron y se convirtieron en una suspensión celular con 500 μL de PBS para su detección.
Reacción en cadena de la polimerasa cuantitativa con transcripción inversa
Se empleó la reacción en cadena de la polimerasa cuantitativa con transcripción inversa (RT-qPCR) para detectar miR-18a, CCL22, receptor activado por proliferador de peroxisomas γ (PPAR-γ) y expresión de ARNm de TGFBR3 en las células recolectadas.
El ARN total se extrajo de las células mediante Trizol (Invitrogen, Carlsbad, CA, EE. UU.) Y se transcribió a la inversa en ARN complementario mediante el kit Mir-X miRNA First Strand Synthesis (Clontech, Mountain View, CA, EE. UU.) Para miR-18a y PrimeScriptTM RT Kit de mezcla maestra (Takara, Dalian, China) para CCL22, PPAR-γ y TGFBR3. U6 y gliceraldehído-3-fosfato deshidrogenasa (GAPDH) fueron controles de carga para miR-18a, CCL22, PPAR-γ y TGFBR3. SYBR @ En la PCR se utilizó Premix Ex Taq ™ II (Perfect Real Time) (Takara) en un sistema LightCycler 480 II (Roche Diagnostics, Indiana, EE.UU.). El cálculo de datos fue evaluado por 2 - △△ CT método. Los cebadores de PCR se presentaron en la Tabla 1.
Ensayo de transferencia Western
El ensayo de transferencia de Western se aplicó a la detección de TGFBR3, proteína (t) -Smad1 total, fosforilada (p) -Smad1, t-Smad3 y p-Smad3 en las células recolectadas.
Se extrajo la proteína total de las células y se determinó la concentración de proteína basándose en el kit de ácido bicinconínico. La muestra de proteína se cargó en los pocillos en electroforesis en gel de poliacrilamida con dodecilsulfato de sodio y se transfirió a una membrana de fluoruro de polivinilideno (PVDF). La membrana de PVDF se bloqueó con leche descremada y se incubó con anticuerpos primarios TGFBR3 (1:2000, R&D Systems, Minneapolis, MN, EE. UU.), T-Smad1 (1:1000), p-Smad1 (1:1000, Santa Cruz Biotechnology) , t-Smad3 (1:1000), p-Smad3 (1:1000) y GAPDH (1:1000, todos de Abcam, Cambridge, MA, Reino Unido) que fue seguido de incubación con el anticuerpo secundario marcado con peroxidasa de rábano picante ( 1:500, Jackson ImmunoResearch Laboratories, PA, EE. UU.). Lavada 3 veces con solución salina tamponada con tris con Tween 20, la membrana se desarrolló mediante quimioluminiscencia mejorada. La cuantificación de las señales fue completada por los Institutos Nacionales de Salud ImageJ Imaging. Software de análisis de procesamiento con intensidad de señalización normalizada a GAPDH.
Cultivo y detección celular
Las líneas de células NPC humanas CNE2, TW03, C666-1 y SUNE-1 y la línea celular nasofaríngea humana normal NP96 (Institutos de Ciencias Biológicas de Shanghai, Academia de Ciencias de China, Shanghai, China) se cultivaron en el Instituto Conmemorativo de Roswell Park (RPMI) - Medio 1640 (Gibco, CA, EE. UU.) Que contiene FBS (Gibco), 100 μg / ml de penicilina y 100 μg / ml de estreptomicina y se pasó a una confluencia del 80%. Se utilizó RT-qPCR para detectar la expresión de miR-18a. Entre esas líneas celulares NPC, las células CNE2 y SUNE-1 mostraron la diferencia más grande y más pequeña en la expresión de miR-18a de las células NP96, por lo que se seleccionaron para ensayos de regulación negativa o positiva de miR-18a.
Agrupación y tratamiento de células
Entre todas las líneas celulares NPC, se seleccionaron las células SUNE-1 con la diferencia más pequeña de las células NP96 en la expresión de miR-18a. Guiados por las especificaciones de Lipofectamine 2000 (Invitrogen), las células SUNE-1 se transfectaron con imitadores de miR-18a, control negativo de imitaciones de miR-18a (NC), si-TGFBR3 o si-TGFBR3 NC.
Entre todas las líneas celulares NPC, las células CNE2 con la mayor diferencia de las células NP96 en la expresión de miR-18a fueron seleccionadas y transfectadas con inhibidores de miR-18a, inhibidores de miR-18a NC, sobreexpresión (OE) -TGFBR3 o OE-TGFBR3 NC por Lipofectamine 2000 (Invitrogen).
Guiados por las especificaciones de Lipofectamine 2000 (Invitrogen), se transfectaron macrófagos M2 con imitadores de miR-18a, imitadores de miR-18a NC, si-TGFBR3, si-TGFBR3 NC, inhibidores de miR-18a, inhibidores de miR-18a NC, OE-TGFBR3, o OE-TGFBR3 NC.
Cocultivo de macrófagos M2 y células NPC
Se adoptó el cocultivo celular en la cámara Transwell para explorar los efectos del miARN del macrófago M2 en las células NPC. La cámara superior se llenó con macrófagos M2 con un tamaño de poro de 0,4 μm, que solo impidió que pasaran las células de la cámara superior, pero no las pequeñas moléculas secretadas por las células, como vesículas, factores de crecimiento, nutrientes, etc. se propagó con células NPC.
Se incubaron SUNE-1 y CNE2 en FBS normal (Gibco). Las células en la fase de crecimiento logarítmico se adoptaron para experimentos.
Las células SUNE-1 y CNE2 se cultivaron conjuntamente con macrófagos M2 en medio FBS-RPMI-1640 al 10% (ambos de Gibco) en una placa de cultivo celular con inserto Transwell (Coring, Corning, NY, EE. UU.) Con un tamaño de poro de 0,4 μm .
Las células SUNE-1 no se cultivaron conjuntamente con macrófagos M2, ni se cultivaron conjuntamente con macrófagos M2, miR-18a imita macrófagos M2 transfectados, miR-18a imita macrófagos M2 transfectados con NC, macrófagos M2 transfectados con si-TGFBR3 o si -TGFBR3 Macrófagos M2 transfectados con NC.
Las células CNE2 no se cultivaron conjuntamente con macrófagos M2, ni se cultivaron conjuntamente con macrófagos M2, macrófagos M2 transfectados con inhibidores de miR-18a, macrófagos M2 transfectados con inhibidores de miR-18a, macrófagos M2 transfectados con OE-TGFBR3 o OE-TGFBR3 Macrófagos M2 transfectados con NC.
Ensayo de bromuro de 3- (4, 5-dimetiltiazol-2-il) -2, 5-difeniltetrazolio
La viabilidad celular se probó mediante el ensayo de bromuro de 3- (4, 5-dimetiltiazol-2-il) -2,5-difeniltetrazolio (MTT), que fue un ensayo colorimétrico aplicado para determinar la actividad de la deshidrogenasa mitocondrial, que redujo el MTT a formazán.
Tripsinizado y sembrado en placas de 96 pocillos a 4 × 10 4 células / pocillo, las células se privaron de medio de cultivo en las horas 0, 12, 24, 36 y 48, respectivamente, y se suplementaron con solución de MTT (500 μL, 0,5 g / L). Se incubaron durante 4 h, se descartó el sobrenadante y las células se incubaron con 200 μL de solución de dimetilsulfóxido. Los valores de densidad óptica (DO, 490 nm) se midieron en un lector de microplacas (ELX808IU, BioTek, VT, EE. UU.). Cada grupo se configuró con 6 pozos paralelos.
Ensayo de formación de colonias
La capacidad de formación de colonias de las células NPC se evaluó mediante un ensayo de formación de colonias, que reflejó la dependencia de la población celular y la proliferación clonal celular.
Se cultivaron durante 24 hy se separaron con tripsina al 0,25%, se sembraron 300 células en una placa de 35 mm con 3 pocillos paralelos en cada grupo. Con medio de cultivo renovado cada 3 días, las células se cultivaron durante una semana y se fijaron con 5 mL de paraformaldehído al 4%. Después de eso, las células se tiñeron con una solución de tinción de violeta cristal y se secaron al aire. Se invirtió la placa sobre la que se superpuso una película transparente de rejillas, y se contó el número de colonias (más de 50 células) bajo un microscopio (Olympus, Tokio, Japón).
Prueba de Scratch
La migración celular se evaluó mediante una prueba de raspado. Las células se tripsinizaron, se sembraron en placas de 6 pocillos con 3 pocillos paralelos para cada grupo y se cultivaron hasta una confluencia del 90%. Luego, las células se incubaron en el medio con FBS al 2% y se dibujaron rayas verticales con una punta de 100 μL. Las células se fotografiaron a las 0 y 24 h bajo un microscopio invertido para medir la distancia de migración celular.
Ensayo Transwell
La invasión y migración celular se ensayaron mediante el ensayo Transwell. La cámara superior de la cámara Transwell se sumergió previamente y se añadió con 100 μL de Matrigel (Coring) que se había diluido con medio RPMI 1640 sin suero a 1:100. Las cámaras superior e inferior se agregaron con 200 μL y 600 μL de medio RPMI 1640 sin suero por separado. Posteriormente, se añadió a la cámara inferior 600 μL de medio RPMI 1640 que contenía FBS al 10%, mientras que a la cámara superior se le añadieron 200 μL de suspensión celular (12,5 × 10 4 células / mL). Se incubaron durante 40 h, las células se tiñeron con una solución de tinción de violeta cristal y se limpiaron con un hisopo de algodón para contar las células que pasan a través del Matrigel bajo un microscopio.
Citometría de flujo
La apoptosis celular y la distribución del ciclo celular se determinaron mediante citometría de flujo.
La distribución del ciclo celular se evaluó mediante tinción con yoduro de propidio (PI). Las células se sembraron en placas de 6 pocillos a 4 x 10 5 células / pocillo, y se cultivaron hasta una confluencia del 70 al 80%. Se fijaron en etanol al 70% preenfriado durante la noche, se centrifugaron las células (se desechó el sobrenadante), se les añadió ARNasa (1 g / L, 200 μL) y Triton X-100 (2 μL), y se tiñeron con solución de tinción PI para 30 minutos. Después de eso, la distribución del ciclo celular se detectó mediante un citómetro de flujo (BD Bioscience, NJ, EE. UU.) A 488 nm de acuerdo con las diferentes intensidades de fluorescencia celular en cada frase (frase G0 / G1, frase S y frase G2 / M).
La apoptosis celular se midió mediante anexina V-isotiocianato de fluoresceína (FITC) y tinción doble de PI. Las células se resuspendieron en 500 μL de tampón de unión y se tiñeron con 5 μL de solución de tinción de Anexina V-FITC y 10 μL de solución de PI. La apoptosis celular también se probó mediante un citómetro de flujo (BD Bioscience) en 30 minutos sin exposición a la luz. En el gráfico de dispersión, las células vivas en el cuadrante inferior izquierdo (Q4) eran FITC - / PI - , las células apoptóticas en la etapa temprana en el cuadrante inferior derecho (Q3) eran FITC + / PI - , y las células necróticas y apoptóticas en la etapa tardía en el cuadrante superior derecho (Q2) fueron FITC + / PI + . Tasa de apoptosis =porcentaje de apoptosis temprana (Q3) + porcentaje de apoptosis tardía (Q2).
Xenoinjertos de tumores en ratones desnudos
Se observó el crecimiento tumoral estableciendo un modelo de NPC en ratones desnudos. Separadas por tripsina al 0,25%, las células SUNE-1 y CNE2 en la fase logarítmica se configuraron en una suspensión de células individuales a 5 × 10 7 células / mL. La suspensión celular (0,2 ml) se inyectó en la axila derecha de los ratones mediante un microinyector para establecer modelos de ratón. Los ratones modelados se criaron en un entorno libre de patógenos específicos. A partir del 4º día se observó crecimiento tumoral y se pesaron los ratones cada 4 días. Se sacrificaron ratones desnudos el día 20 después de la inyección y se resecaron los tumores, se pesaron con una balanza electrónica y se fotografiaron.
Ensayo génico indicador de luciferasa dual
Se adoptó el sistema de genes indicadores de luciferasa dual para confirmar los sitios de unión de miR-18a y la región 3 'no traducida (UTR) del ARNm de TGFBR3. Se utilizó un sitio web de predicción biológica (http://www.microrna.org/microrna/home.do) para analizar el gen diana de miR-18a y descubrir la existencia del sitio de unión complementario de miR-18a en el 3 ′ UTR de TGFBR3. Se empleó el ensayo del gen indicador de luciferasa dual para verificar adicionalmente si el TGFBR3 estaba directamente dirigido por miR-18a. Se construyó un sitio de unión pmirGLO-TGFBR3 de tipo salvaje (WT) y un pmirG-LO-TGFBR3 de tipo mutante (MUT) de TGFBR3 3'UTR. TGFBR3-WT o TGFBR3-MUT y miR-18a imita o imita NC se cotransfectaron en células SUNE-1 y CNE2 mediante Lipofectamine 2000 (Invitrogen) y se incubaron durante 48 h. Se utilizó un kit de ensayo de luciferasa (Promega, Madison, WI, EE. UU.) Para analizar las células.
Análisis estadístico
Para el análisis se utilizó el software estadístico SPSS21.0 (IBM Corp. Armonk, NY, EE. UU.). Los datos se expresaron como media ± desviación estándar. t analizó las diferencias entre dos grupos pruebe las diferencias entre varios grupos mediante el análisis de varianza unidireccional (ANOVA), seguido de la prueba post hoc de Tukey. Se consideró una diferencia significativa en P <0.05.
Resultados
Identificación de macrófagos M2
Los monocitos enriquecidos por el método adherente y los monocitos inducidos por M-CSF se recolectaron de sangre periférica de donantes sanos. La detección por inmunofluorescencia de CD68, CD206 y CD163 confirmó que las PBMC inducidas por M-CSF in vitro se convirtieron en macrófagos con características moleculares típicas, que cumplieron con nuestros requisitos (Fig. 1a).
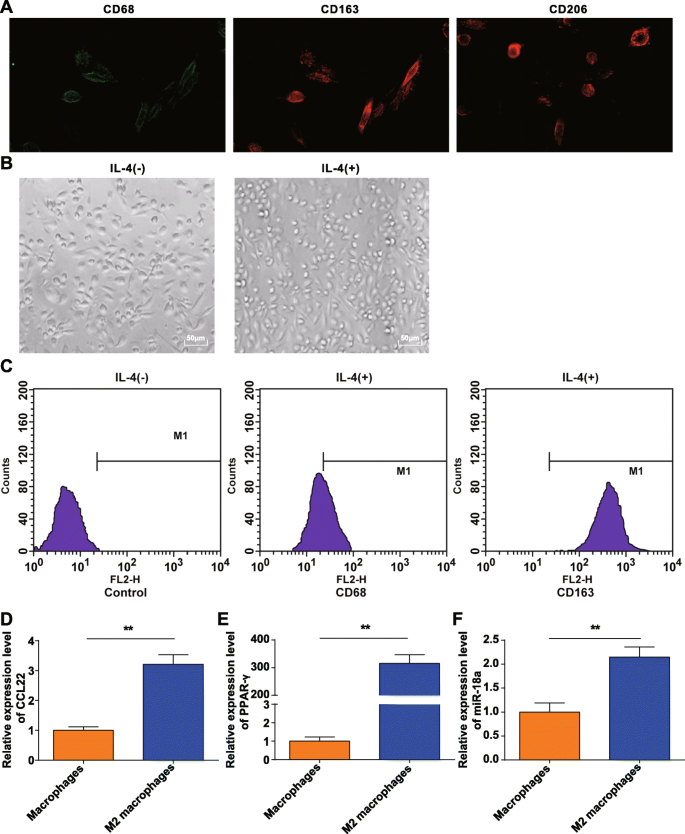
Identificación de macrófagos M2. un . CD68, CD206 y CD163 se expresaron en la superficie de macrófagos obtenidos después de la inducción de monocitos in vitro. b Los macrófagos polarizados por IL-4 fueron macrófagos M2. c CD163 se expresó mucho y CD68 se expresó escasamente en macrófagos M2 polarizados por IL-4. d CCL22 se expresó en gran medida en macrófagos M2. e PPAR-γ se expresó en gran medida en macrófagos M2. f miR-18a se expresó altamente en macrófagos M2; * P <0,05; ** P <0,01. Los datos de medición se expresaron como media ± desviación estándar, N =3. t analizó las comparaciones entre dos grupos prueba
Los macrófagos obtenidos se polarizaron con IL-4 y se observaron al microscopio para determinar su morfología. Se manifestó que los macrófagos (M0) sin estimulación de IL-4 eran diversos e irregulares, mostrando una forma redonda, ovalada o fusiforme. Estimulados por IL-4, los macrófagos M2 se volvieron más grandes y predominantemente volvieron a una forma redonda, que estaban en conformidad con las características morfológicas de los macrófagos M2 como se describió anteriormente [20] (Fig. 1b).
La citometría de flujo probó los antígenos de superficie de las células adherentes estimuladas por 20 ng / mL de IL-4 durante 24 h y encontró que CD68 se expresaba al 21,16% mientras que CD163 estaba al 98,69% del número total de células (Fig. 1c), lo que confirma las células adherentes son macrófagos M2. RT-qPCR reveló que con las células M0, por el contrario, CCL22 y PPAR-γ (moléculas polarizantes típicas) aumentaron en los macrófagos M2 (Fig. 1d, e), lo que indica la inducción exitosa de macrófagos M2.
RT-qPCR también manifestó que la expresión de miR-18a aumentó en los macrófagos M2 en contraste con los macrófagos M0 ( P ˂ 0.05) (Figura 1f).
MiR-18a se expresa mucho y TGFBR3 se expresa mal en las células NPC
El sitio web de bioinformática (miRanda) predijo los objetivos potenciales de miR-18a y TGFBR3 se consideró como un objetivo de miR-18a (Fig. 2a). Se implementó el ensayo del gen indicador de luciferasa dual para verificar que el miR-18a se dirigía a 3'UTR de TGFBR3. Se clonaron TGFBR3-WT o TGFBR3-MUT en el vector pmirGLO y se cotransfectaron con imitadores de miR-18a o NC en células SUNE-1 y CNE2. Los imitadores de miR-18a no tuvieron influencia sobre la actividad luciferasa de TGFBR3 3′UTR-MUT pero alteraron la de TGFBR3 3′UTR-WT en células SUNE-1 y CNE2, lo que sugiere que TGFBR3 era un gen diana regulado por miR-18a (Fig. . 2b).
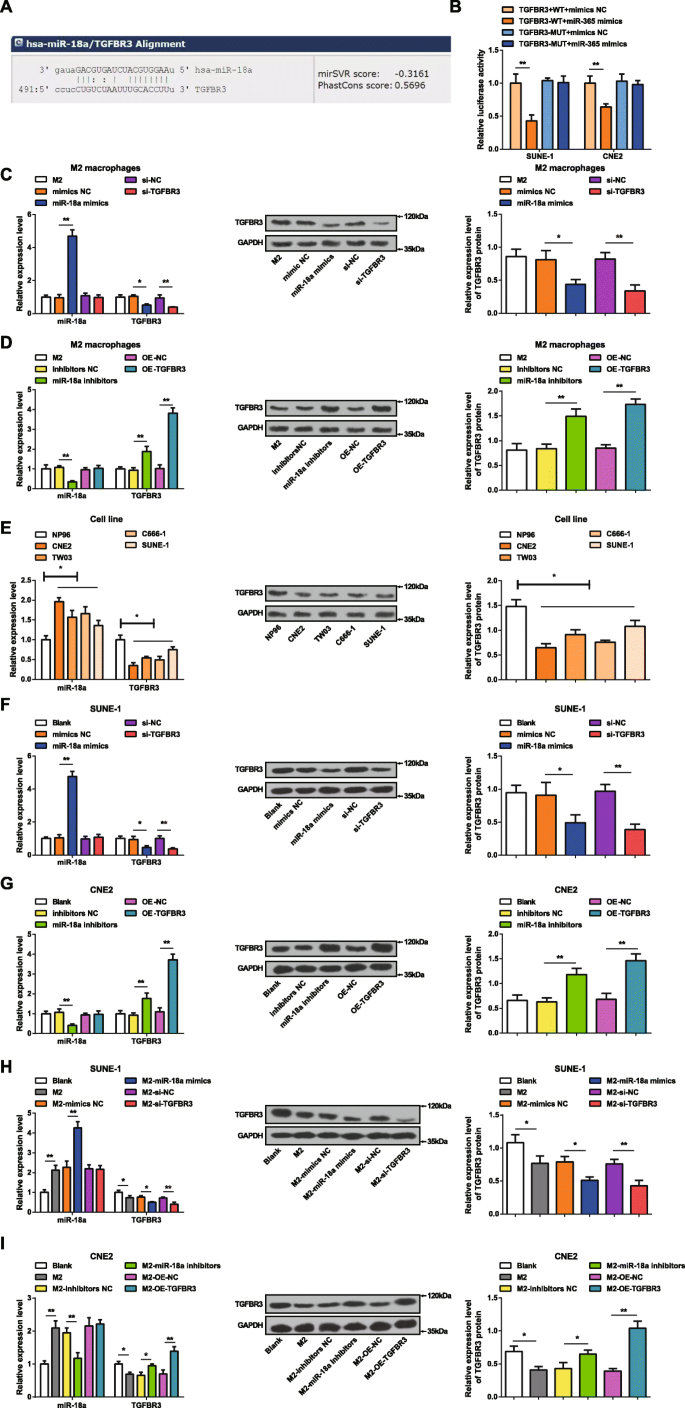
MiR-18a está altamente expresado y TGFBR3 se expresa pobremente en células NPC. un miRanda predijo que miR-18a se dirigía a TGFBR3. b Ensayo de gen indicador de luciferasa dual verificado miR-18a dirigido a TGFBR3. c La expresión de miR-18a fue elevada y la expresión de TGFBR3 disminuyó en macrófagos M2 transfectados con míR-18a. d La expresión de miR-18a se redujo y la expresión de TGFBR3 se elevó en macrófagos M2 transfectados con inhibidores de miR-18a. e La expresión de miR-18a se elevó y la expresión de TGFBR3 disminuyó en las líneas celulares NPC frente a las células NP96. f La expresión de miR-18a fue elevada y la expresión de TGFBR3 disminuyó en células SUNE-1 transfectadas con míR-18a. g La expresión de miR-18a se redujo y la expresión de TGFBR3 se elevó en células CNE2 transfectadas con inhibidores de miR-18a. h La expresión de miR-18a se elevó y la expresión de TGFBR3 se redujo en células SUNE-1 cocultivadas con macrófagos M2 transfectados con míR-18a. yo la expresión de miR-18a se redujo y la expresión de TGFBR3 se elevó en células CNE2 cocultivadas con macrófagos M2 transfectados con inhibidores de miR-18a; * P <0,05; ** P <0,01. Los datos de medición se expresaron como media ± desviación estándar, N =3. Las comparaciones entre dos grupos fueron analizadas por t prueba. Las comparaciones entre varios grupos se analizaron mediante ANOVA de una vía, seguido de la prueba post hoc de Tukey
La eficacia de transfección de imitadores de miR-18a o inhibidores de miR-18a en macrófagos M2 se manifestó mediante la determinación de la expresión de miR-18a y TGFBR3 en macrófagos M2 mediante RT-qPCR y ensayo de transferencia Western. Era obvio que (Fig. 2c, d) la sobreexpresión de miR-18a elevó la expresión de miR-18a y redujo la expresión de TGFBR3 en macrófagos M2. Por el contrario, la inhibición de miR-18a redujo la expresión de miR-18a y aumentó la expresión de TGFBR en macrófagos M2. No se observó ninguna diferencia en la expresión de miR-18a, mientras que la expresión de TGFBR3 disminuyó cuando los macrófagos M2 se transfectaron con si-TGFBR3. No se observaron diferencias en la expresión de miR-18a, mientras que la expresión de TGFBR3 creció tras la transfección de OE-TGFBR3 a macrófagos M2.
Se ensayó la expresión de miR-18a y TGFBR3 en líneas celulares CNE2, TW03, C666-1, SUNE-1 y NP96 mediante RT-qPCR y ensayo de transferencia Western. En las células NPC, en comparación con las células NP96, la expresión de miR-18a aumentó y la expresión de TGFBR3 disminuyó (Fig. 2e). Debido a que las células CNE2 y las células SUNE-1 se manifestaron con las diferencias más grandes y más pequeñas en la expresión de miR-18a de las células NP96, se seleccionaron para los ensayos de regulación negativa y aumento de miARN en curso, respectivamente.
Para identificar los efectos de miR-18a y TGFBR3 en células NPC, se transfectaron células SUNE-1 con imitadores de miR-18a o si-TGFBR3, mientras que células CNE2 con inhibidores de miR-18a o OE-TGFBR3. El ensayo RT-qPCR y Western blot demostraron que miR-18a imita la expresión elevada de miR-18a y la expresión reducida de TGFBR3 en células SUNE-1. La transfección de si-TGFBR3 no tuvo efecto sobre la expresión de miR-18a mientras que redujo la expresión de TGFBR3 en las células SUNE-1. Los inhibidores de miR-18a redujeron la expresión de miR-18a y elevaron la expresión de TGFBR3 en las células CNE2. La transfección de OE-TGFBR3 en células CNE2 no afectó la expresión de miR-18a, pero elevó la expresión de TGFBR3 (Fig. 2f, g).
Para explorar los efectos de miR-18a de macrófagos M2 en células NPC, miR-18a imita o macrófagos M2 transfectados con si-TGFBR3 o inhibidores de miR-18a o macrófagos M2 transfectados con OE-TGFBR3 se cocultivaron con SUNE-1 o células CNE2 en la cámara Transwell, respectivamente. El ensayo RT-qPCR y Western blot evaluó la expresión de miR-18a y TGFBR3 en células SUNE-1 o CNE2. Células SUNE-1 cocultivadas con macrófagos M2 transfectados con imitación de miR-18a o no transfectados demostrados con expresión de miR-18a elevada y expresión de TGFBR3 degradada. No se notó diferencia en la expresión de miR-18a y la expresión de TGFBR3 reducida en células SUNE-1 que se habían cocultivado con macrófagos M2 transfectados con si-TGFBR3 (Fig. 2h). Después de cocultivo con macrófagos M2 no transfectados, las células CNE2 se caracterizaron por un aumento de la expresión de miR-18a y una disminución de la expresión de TGFBR3. Sin embargo, la expresión reducida de miR-18a y la expresión elevada de TGFBR3 se presentaron en células CNE2 que se cocultivaron de antemano con macrófagos M2 transfectados con inhibidores de miR-18a. No se reconoció ninguna diferencia en la expresión de miR-18a, y la expresión de TGFBR3 aumentó en las células CNE2 cocultivadas con macrófagos M2 transfectados con OE-TGFBR3 (Fig. 2i).
miR-18a de M2 Macrophages promueve la viabilidad de células NPC y la capacidad de formación de colonias
Se aplicaron el ensayo MTT y el ensayo de formación de colonias para identificar los impactos de miR-18a y TGFBR3 sobre la viabilidad y la capacidad de formación de colonias de las células SUNE-1 y las células CNE2. Se transfectaron imitadores de miR-18a, inhibidores de miR-18a, si-TGFBR3 o OE-TGFBR3 en células SUNE-1 o células CNE2. Se indicó que en las células SUNE-1, la regulación positiva de miR-18a o la regulación negativa de TGFBR3 mejoraron la viabilidad celular y el número de colonias elevado (Fig. 3a, e). En las células CNE2, la viabilidad celular se vio afectada y el número de colonias se redujo por la inhibición de miR-18a o la sobreexpresión de TGFBR3 (Fig. 3b, f).
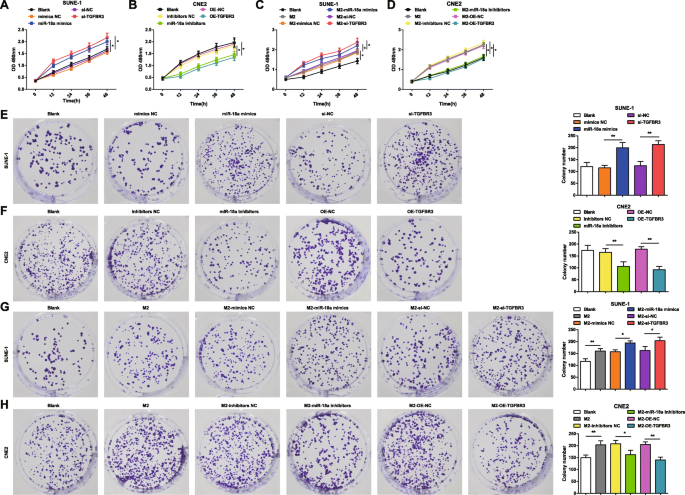
miR-18a de macrófagos M2 induce la viabilidad de las células NPC y la capacidad de formación de colonias. un miR-18a imita o si-TGFBR3 aumenta la viabilidad de las células SUNE-1. b Los inhibidores de miR-18a o OE-TGFBR3 disminuyeron la viabilidad de las células CNE2. c El co-cultivo con imitadores de miR-18a o macrófagos M2 transfectados con si-TGFBR3 aumentó la viabilidad de las células SUNE-1. d El cocultivo con inhibidores de miR-18a o macrófagos M2 transfectados con OE-TGFBR3 disminuyó la viabilidad de las células CNE2. e miR-18a imita o si-TGFBR3 aumenta el número de colonias de células SUNE-1. f Los inhibidores de miR-18a o OE-TGFBR3 disminuyeron el número de colonias de células CNE2. g El cocultivo con imitadores de miR-18a o macrófagos M2 transfectados con si-TGFBR3 aumentó el número de colonias de células SUNE-1. h El cocultivo con inhibidores de miR-18a o macrófagos M2 transfectados con OE-TGFBR3 disminuyó el número de colonias de células CNE2; * P <0,05; ** P <0,01. Los datos de medición se expresaron como media ± desviación estándar, N =3. Las comparaciones entre varios grupos se analizaron mediante ANOVA de una vía, seguido de la prueba post hoc de Tukey
Para examinar los efectos de miR-18a del macrófago M2 sobre la viabilidad y la capacidad de formación de colonias de las células NPC, se cocultivaron macrófagos M2 transfectados con imitadores de miR-18a, inhibidores de miR-18a, si-TGFBR3 o OE-TGFBR3 con células SUNE-1 o células CNE2 en la cámara Transwell. El ensayo de formación de colonias y MTT mostró que las células SUNE-1 cocultivadas con macrófagos M2 no transfectados o transfectados con imitadores de miR-18a o transfectados con si-TGFBR3 presentaban viabilidad celular reforzada y un mayor número de colonias (Fig.3c, g) .
Las células CNE2 cocultivadas con macrófagos M2 no transfectados se destacaron con una viabilidad celular reforzada y un aumento de colonias. Sin embargo, las células CNE2 cocultivadas con macrófagos M2 transfectados con inhibidores de miR-18a o transfectados con OE-TGFBR mostraron una viabilidad celular disminuida y colonias disminuidas (Fig. 3d, h).
miR-18a de M2 Macrophages promueve la invasión de células NPC y las habilidades de migración
Para una mejor comprensión de cómo miR-18a y TGFBR3 afectaron la migración y la invasión de células NPC, se implementaron la prueba de raspado y el ensayo Transwell. Los resultados revelaron que las células SUNE-1 transfectadas con imitadores de miR-18a o si-TGFBR3 se caracterizaron por una mayor distancia de migración celular y células de invasión (Figuras 4a y 5a).
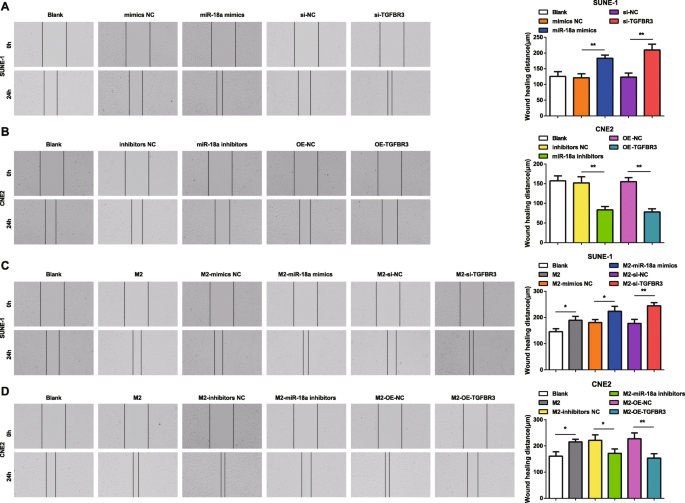
El miR-18a de los macrófagos M2 promueve la capacidad de migración de células NPC. un miR-18a imita o si-TGFBR3 aumenta la migración de las células SUNE-1. b Los inhibidores de miR-18a o OE-TGFBR3 disminuyeron la migración de las células CNE2. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages increased migration of SUNE-1 cells. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages decreased migration of CNE2 cells; * P <0.05; ** P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
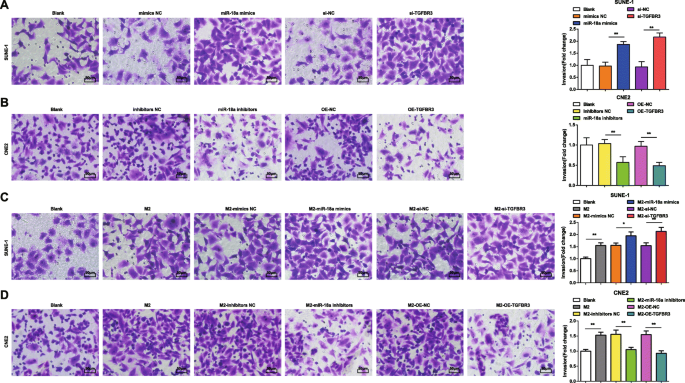
miR-18a from M2 macrophages promotes NPC cell invasion ability. un miR-18a mimics or si-TGFBR3 increased invasion of SUNE-1 cells. b miR-18a inhibitors or OE-TGFBR3 decreased invasion of CNE2 cells. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages increased invasion of SUNE-1 cells. b Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages decreased invasion of CNE2 cells; * P <0.05; ** P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
In CNE2 cells transfected with miR-18a inhibitors or OE-TGFBR3, the reductions appeared in cell migration distance and invasion cells (Figs. 4b and 5b).
How miR-18a from M2 macrophages influenced invasion and migration abilities of NPC cells were deciphered by miR-18a mimics-, miR-18a inhibitors-, si-TGFBR3-, or OE-TGFBR3-transfected M2 macrophages co-culturing with SUNE-1 or CNE2 cells. The results highlighted that SUNE-1 cells co-cultured with untransfected, or miR-18a mimics-transfected or si-TGFBR3-transfected M2 macrophages were manifested with increased cell migration distance and invasion cells (Figs. 4c and 5c).
Both cell migration distance and invasion cells increased in CNE2 cells co-cultured with untransfected M2 macrophages. Upon co-culture with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages, CNE2 cells were showed with reduced migration distance and invasion cells (Figs. 4d and 5d).
miR-18a from M2 Macrophages Arrests Fewer NPC Cell at G0/G1 Phase and Suppresses Apoptosis
Cell cycle distribution and apoptosis were tested by flow cytometry to stratify the effects of miR-18a and TGFBR3 on NPC cells. It was indicated that transfection of miR-18a mimics or si-TGFBR3 reduced SUNE-1 cells arrested in the G0/G1 phase, increased cells in the S and G2/M phases, and reduced cell apoptosis rate (Figs. 6a and 7a).
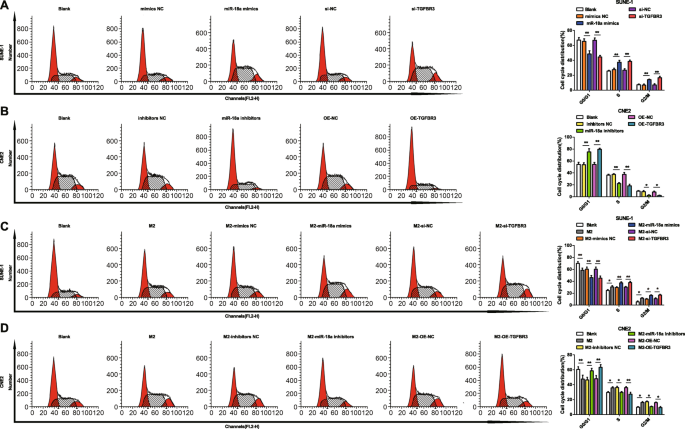
miR-18a from M2 macrophages arrests fewer NPC cell at G0/G1 phase. un miR-18a mimics or si-TGFBR3 decreased SUNE-1 cells in the G0/G1 phase, and increased SUNE-1 cells in the S and G2/M phases. b miR-18a inhibitors or OE-TGFBR3 increased CNE2 cells in the G0/G1 phase, and decreased CNE2 cells in the S and G2/M phases. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages decreased SUNE-1 cells in the G0/G1 phase, and increased SUNE-1 cells in the S and G2/M phases. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages increased CNE2 cells in the G0/G1 phase, and decreased CNE2 cells in the S and G2/M phases; * P <0.05; ** P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
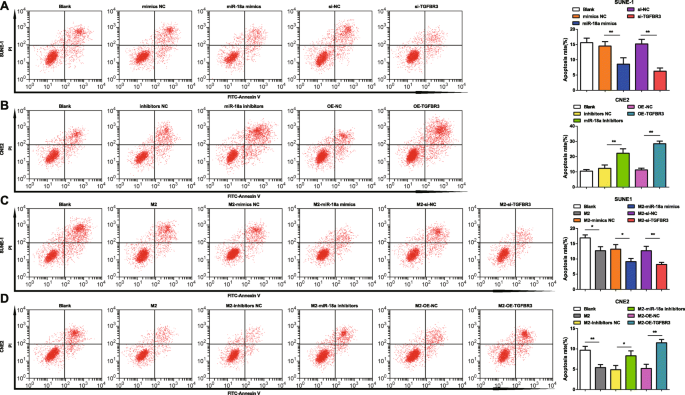
miR-18a from M2 macrophages inhibits NPC cell apoptosis. un miR-18a mimics or si-TGFBR3 decreased apoptosis of SUNE-1 cells. b miR-18a inhibitors or OE-TGFBR3 increased apoptosis of CNE2 cells. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages decreased apoptosis of SUNE-1 cells. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages increased apoptosis of CNE2 cells; * P <0.05; ** P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
Upon transfection with miR-18a inhibitors or OE-TGFBR3, CNE2 cells in the G0/G1 phase trended toward an elevation while those in the S and G2/M phases toward a reduction, and cell apoptosis rate raised (Figs. 6b and 7b).
With the purpose to decode the mechanism of miR-18a from M2 macrophages in NPC cell cycle distribution and apoptosis, M2 macrophages transfected with miR-18a mimics, miR-18a inhibitors, si-TGFBR3, or OE-TGFBR3 were co-cultured with SUNE-1 cells or CNE2 cells in the Transwell chamber. Co-cultured with untransfected, or miR-18a mimics-transfected or si-TGFBR3-transfected M2 macrophages, reduced SUNE-1 cells were displayed in the G0/G1 phase and increased cells in the S and G2/M phases, and SUNE-1 cell apoptosis rate decreased (Figs. 6c and 7c).
Co-cultured with untransfected M2 macrophages, CNE2 cells in the G0/G1 phase reduced, cells in the S and G2/M phases increased, and apoptosis rate declined. On the contrary, co-cultured with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages, CNE2 cells in the G0/G1 phase elevated while those in the S and G2/M phases decreased, and apoptosis rate elevated (Figs. 6d and 7d).
miR-18a from M2 Macrophages Reduces p-Smad1/t-Smad1 and Elevates p-Smad3/t-Smad3 in NPC Cells
Western blot assay detected TGF signaling pathway-related proteins in NPC cells to further explain the effects of miR-18a and TGFBR3 on TGF signaling pathway.
It was explained that transfection of miR-18a mimics or si-TGFBR3 reduced p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3 in SUNE-1 cells (Fig. 8a).
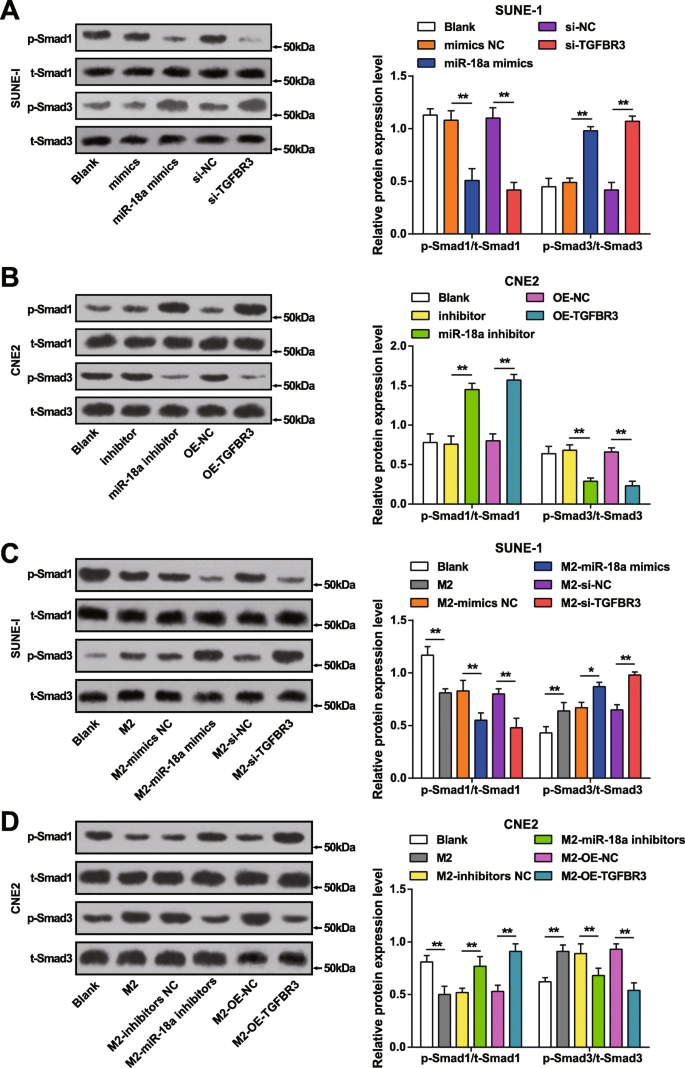
miR-18a from M2 macrophages decreases p-Smad1/t-Smad1 and increases p-Smad3/t-Smad3 in NPC cells. un miR-18a mimics or si-TGFBR3 in SUNE-1 cells decreased p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3. b miR-18a inhibitors or OE-TGFBR3 in CNE2 cells increased p-Smad1/t-Smad1 and decreased p-Smad3/t-Smad3. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages decreased p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages increased p-Smad1/t-Smad1 and decreased p-Smad3/t-Smad3; * P <0.05; ** P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
Transfection with miR-18a inhibitors or OE-TGFBR3 led to declined p-Smad3/t-Smad3 and increased p-Smad1/t-Smad1 in CNE2 cells (Fig. 8b).
miR-18a from M2 macrophages influencing TGF signaling pathway in NPC cells was determined by Western blot assay through testing TGF signaling pathway-related proteins in SUNE-1 cells and CNE2 cells which had co-cultured with miR-18a mimics-, miR-18a inhibitors-, si-TGFBR3-, or OE-TGFBR3-transfected M2 macrophages in the Transwell chamber.
SUNE-1 cells co-cultured with untransfected, miR-18a mimics-transfected, or si-TGFBR3-transfected M2 macrophages were manifested with reduced p-Smad1/t-Smad1 and incremental p-Smad3/t-Smad3 (Fig. 8c).
Co-cultured with untransfected M2 macrophages, CNE2 cells trended toward declined p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3. In an opposite way, CNE2 cells were demonstrated with increased p-Smad1/t-Smad1 and decreased p-Smad3/t-Smad3 when co-cultured with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages (Fig. 8d).
miR-18a from M2 Macrophages Induces Tumor Growth in Nude Mice with NPC
Tumor xenografts were conducted on nude mice to further elucidate the impacts of miR-18a and TGFBR3 on tumor growth of NPC.
It was indicated that injected with miR-18a mimics-transfected or si-TGFBR3-transfected SUNE-1 cells, mice were manifested with enlarged tumor volume and heavier tumor weight (Fig. 9a).
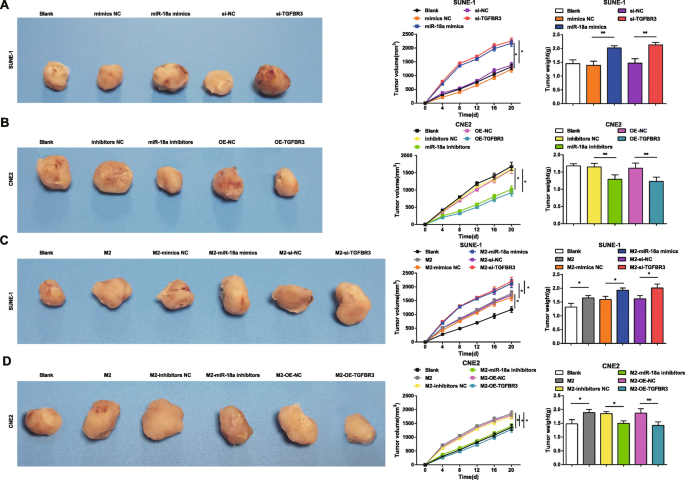
miR-18a from M2 macrophages promotes tumor growth in nude mice with NPC. un miR-18a mimics or si-TGFBR3 in SUNE-1 cells increased tumor volume and weight. b miR-18a inhibitors or OE-TGFBR3 in CNE2 cells decreased tumor volume and weight. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages increased tumor volume and weight. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages decreased tumor volume and weight; * P <0.05; ** P <0.01. Measurement data were expressed as mean ± standard deviation, three nude mice in each group. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
Reduced tumor volume and weight were presented in mice with injection of miR-18a inhibitors-transfected or OE-TGFBR3-transfected CNE2 cells (Fig. 9b).
Tumor growth was observed in mice which had injected with miR-18a mimics-, miR-18a inhibitors-, si-TGFBR3-, or OE-TGFBR3-transfected M2 macrophages to illustrate the mechanism of miR-18a from M2 macrophages in NPC.
After co-culture with untransfected, miR-18a mimics-transfected or si-TGFBR3-transfected M2 macrophages, SUNE-1 cells were injected into mice and mice were observed with larger tumor volume and heavier tumor weight (Fig. 9c).
CNE2 cells were co-cultured with M2 macrophages and injected into mice with the results suggesting growing tumor volume and weight. Both tumor volume and weight were inclined to reduce when mice were injected with CNE2 cells which had co-cultured with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages (Fig. 9d).
Discussion
NPC refers to a polygenic disease threatened by a wide range of factors [21]. MiRNAs are previously implied to participate in the pathogenesis of NPC via regulation of their target genes which are indicators of cellular processes and pathways [22]. Concretely, miR-18a advances NPC progression by miRNA biogenesis impairing [6]. Given that this study goes forward to decipher the combined interactions of miR-18a from M2 macrophages and TGFBR3 in NPC with the conclusion elucidating that miR-18a from M2 macrophages stimulates NPC progression via TGFBR3 inhibition (Fig. 10).
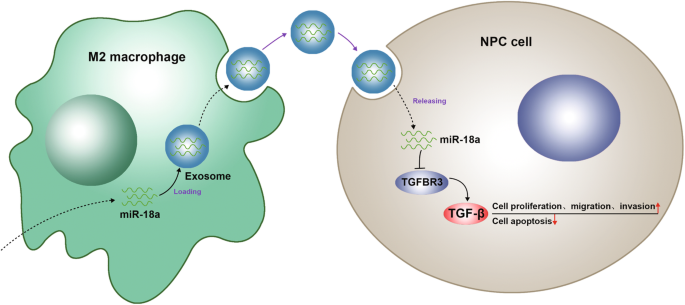
Schematic representation of macrophage-derived exosomal miR-18a in NPC and the involvement of TGFBR3-mediated TGF-β signaling pathway
At the start of this study, macrophages are stimulated by IL-4 to differentiate to M2 macrophages which are found to enrich the expression of miR-18a. As we know, during macrophage polarization, miRNA’s expression was altered [23]. In addition, M2 polarization enriches genes which are involved in the cell cycle and metabolic processes, and the M2 phenotype is conducive to tumor growth and angiogenesis in neoplastic tissues [24]. Based on the M2 macrophages-enriched miR-18a, a series of experiments were successfully conducted.
Initially, our study has uncovered that miR-18a is highly expressed while TGFBR3 is poorly expressed in NPC cells. Drawn from a previous study, it is concluded that miR-18a is overexpressed in NPC tissues with its association with lymph node metastasis and clinical stage [5]. Besides that, miR-17-92 cluster members including miR-18a are documented to be overexpressed in NPC tissues [25]. Furthermore, upregulated miR-18a is reported to demonstrate in NPC tissues which is connected with tumor node metastasis stage and tumor size [4]. Experimentally, except for the downregulated TGFBR3 in tongue squamous cell carcinoma [26], there has been another study depicting reduced TGFBR3 in clear-cell renal cell carcinomas accompanied by unwanted prognosis [27]. Anyhow, the results in this study are consistent with these study findings to some extent.
In order to explore the roles of miR-18a and TGFBR3 in NPC cell progression, we have conducted a series of assays with the results indicating that upregulated miR-18a or downregulated TGFBR3 triggers NPC cell progression while miR-18a repression or TGFBR3 elevation has the opposite effects on NPC cells. Widely, suppression of miR-18a is evidenced to hamper cell progression in malignancies including ovarian cancer, colitis-associated colorectal cancer, and hepatocellular carcinoma [28,29,30]. Narrowly, an existed study has pronounced that upregulated miR-18a promotes NPC cell progression via mediation of DICER1 [6]. In addition, it is noticed that overexpressed miR-18a in NPC is believed to connect with NPC metastasis and repressed miR-18a partially contributes to better prognosis of NPC patients [31]. Lately, it is surveyed that downregulation of miR-18a is capable of discouraging NPC proliferation, invasion, and migration [4]. Additionally, a decrease in TGFBR3 expression is regarded to link with laryngeal squamous cell carcinoma (LSCC) invasion and miR-223/TGFBR3 axis regulation takes part in LSCC progression inhibition [32]. TGFBR3 elevation is documented to restrict NPC cell viability, induce apoptosis, and activate pro-apoptosis signaling pathways [14]. Previously, a study has indicated that upregulation of TGFBR3 promotes apoptosis and cells arrested in the G2/M phase, resulting in impaired cell viability and migration in salivary gland adenoid cystic carcinoma [33]. Intriguingly, it is formerly described that induction of TGFBR3 contributes to disrupt intrahepatic cholangiocarcinoma progression [34].
Despite the protective role of lowly expressed miR-18a and overexpressed TGFBR3 in NPC cell in vitro, we have performed tumor xenografts in nude mince in vivo for further verification with the results explaining that miR-18a knockdown or TGFBR3 elevation restrains tumor growth in nude mice. As demonstrated in a prior study, miR-18a-injected nude mice show with enhanced tumor growth [5] and conversely, the miR-18a antagomir-injected nude mice are displayed with suppressed tumor growth in NPC [4]. In the light of the TGFBR3 reduction in tumor growth, it is suggested that poorly expressed TGFBR3 provokes tumor formation in clear-cell renal cell carcinoma [27]. In the opposite way, an increase in TGFBR3 is recognized to hinder tumor growth in lung cancer with the presence of long non-coding RNA ADAM metallopeptidase with thrombospondin type 1 motif, 9 antisense RNA 2 elevation, and miR-223-3p suppression [35]. This study has also predicted and verified that TGFBR3 is a target gene of miR-18a. But, more studies still need to be conducted for further verification.
Conclusion
Generally speaking, this study elaborates the concrete mechanisms that miR-18a from M2 macrophages inhibits TGFBR3 expression to exacerbate the progression of NPC via TGF-β signaling pathway, the results of which is abrogated by miR-18a knockdown or TGFBR3 elevation. This study updates the therapeutic target for NPC. However, a large cohort researches are still in need for in-depth explorations.
Disponibilidad de datos y materiales
No aplica.
Nanomateriales
- Compuesto híbrido Nanoestructurado de sílice / oro-celulosa-Amino-POSS mediante el proceso Sol-Gel y sus propiedades
- Eficacia antitumoral y farmacocinética mejoradas de la bufalina mediante liposomas pegilados
- Preparación de nanoesferas poliméricas impresas con iones de paladio (II) y su eliminación del paladio (II) de la solución acuosa
- Nanoconjuntos de ácido 5-aminolevulínico-escualeno para fotodetección y terapia de tumores:estudios in vitro
- Promoción del crecimiento celular SH-SY5Y mediante nanopartículas de oro modificadas con 6-mercaptopurina y un péptido penetrante de neuronas
- Las nanopartículas de maghemita actúan como nanozimas, mejorando el crecimiento y la tolerancia al estrés abiótico en Brassica napus
- Síntesis y caracterización de BiOCl modificado y su aplicación en la adsorción de tintes de baja concentración a partir de una solución acuosa
- Material y propiedades ópticas de los puntos cuánticos de carbono fluorescente fabricados a partir de jugo de limón mediante una reacción hidrotermal
- Crecimiento propio de MOCVD y fotoluminiscencia mejorada espectacularmente de nanocables de núcleo-carcasa de InGaAs / InP
- La liberación de CpG con nanopartículas magnéticas de Fe3O4 modificadas inhibe el crecimiento tumoral y las metástasis pulmonares espontáneas para mejorar la inmunoterapia
- Nanoalginatos mediante síntesis de micelas inversas:encapsulación de doxorrubicina y citotoxicidad del cáncer de mama



