SOX2 regula el eje lncRNA CCAT1 / MicroRNA-185-3p / FOXP3 para afectar la proliferación y autorrenovación de las células madre del cáncer de cuello uterino
Resumen
Se ha presentado el papel de los ARN no codificantes largos (lncRNA) en el cáncer de cuello uterino (CC). Nuestro objetivo es discutir el efecto de la región determinante del sexo Y-box 2 (SOX2) / lncRNA colon-related transcript-1 (CCAT1) / microRNA-185-3p (miR-185-3p) / forkhead box protein 3 (FOXP3) ) sobre la capacidad de proliferación y autorrenovación de las células madre CC. Se probaron las expresiones de MiR-185-3p, SOX2, CCAT1 y FOXP3 en tejidos y células CC. Se verificó la relación entre la expresión de SOX2 / CCAT1 y las características clínico-patológicas en pacientes con CC. Se realizaron investigaciones de pérdida y ganancia de función en CD44 + Células HeLa para discutir las funciones biológicas y la capacidad de autorrenovación. Finalmente, se verificaron las relaciones entre SOX2, CCAT1, FOXP3 y miR-185-3p. La expresión de miR-185-3p disminuyó, mientras que las expresiones de SOX2, CCAT1 y FOXP3 aumentaron en los tejidos y células CC. Las expresiones de SOX2 y CCAT1 se relacionaron con el tamaño del tumor, la metástasis en los ganglios linfáticos y la federación internacional de ginecología y obstetricia estadio de CC. La regulación a la baja de SOX2 o CCAT1 y la regulación al alza de miR-185-3p dieron como resultado la inhibición de la proliferación, la invasión, la migración y el número de esferas celulares, así como la aceleración de la apoptosis de CD44 + Células HeLa. SOX2 podría unirse a CCAT1, lo que afectó la expresión de miR-185-3p, y FOXP3 fue el objetivo de miR-185-3p.
Introducción
El cáncer de cuello uterino (CC) es la cuarta causa principal de mortalidad en mujeres con un estimado de 570.000 casos y 311.000 muertes en todo el mundo en 2018 [1]. Esta compleja enfermedad participa en una variedad de factores, incluidos los efectos genéticos y la infección viral [2]. Con el desarrollo de la prueba conjunta del virus del papiloma humano y la vacunación contra el virus del papiloma humano, los procedimientos de diagnóstico temprano de la displasia cervical y el cáncer dan como resultado una reducción de la incidencia, morbilidad y mortalidad de CC [3]. Para los pacientes con CC temprana, se recomienda la cirugía, como la cirugía para preservar la fertilidad, la biopsia de cono, la traquelectomía radical, la disección de los ganglios linfáticos pélvicos, la radioterapia pélvica y la braquiterapia [4]. Debido a metástasis o recurrencia en pacientes con CC avanzada, el pronóstico sigue siendo malo [5]. Por tanto, sigue siendo urgente identificar marcadores pronósticos y estrategias terapéuticas nuevos y eficaces para mejorar el tratamiento del CC.
La región determinante del sexo Y-box 2 (SOX2) es un miembro crítico de la familia de factores de transcripción SOX y se manifiesta principalmente en células madre embrionarias y adultas y también se expresa en células madre tumorales [6]. Ha revelado que SOX2 modula la radiorresistencia en CC mediante la vía de señalización hedgehog [7]. Otro estudio ha demostrado que SOX2 es crucial para mantener la subpoblación de células madre cancerosas en líneas celulares CC [8]. Los ARN largos no codificantes (lncRNA) son una clase de moléculas de ARN con 200 nucleótidos de longitud [9]. La transcripción 1 asociada al cáncer de colon de LncRNA (CCAT1) se encuentra en el cromosoma humano 8q24.21 y se considera un "punto caliente" que da lugar a mutaciones genéticas en el cáncer de colon [10]. Un estudio ha informado de que CCAT1 acelera la proliferación celular y la invasión de CC [11]. Según Jia et al., CCAT1 mejora drásticamente la proliferación, migración e invasión de células CC [12]. Además, otro estudio ha revelado que CCAT1 mejora el grado maligno de la enfermedad inflamatoria intestinal a través de la destrucción de la barrera intestinal al reducir el microARN-185-3p (miR-185-3p) [13]. MiRNA puede controlar de forma inversa la expresión de genes mediante la reducción del mRNA y la supresión de la traducción [14]. Los experimentos in vitro en un estudio anterior han revelado que miR-185-3p modula la radiorresistencia del carcinoma nasofaríngeo [15]. Otro estudio ha implicado que miR-185 participa en la resistencia al cisplatino del cáncer de ovario in vivo e in vitro [16]. La proteína forkhead box 3 (FOXP3) es un factor de transcripción perteneciente a la familia de proteínas FOX, que se encuentra por primera vez en las células reguladoras T (Treg) y desempeña un papel vital en el mantenimiento y proceso de las células Treg [17]. Un estudio informa que FOXP3 está conectado a la linfangiogénesis de CC [18]. Otro estudio revela que el nivel de FOXP3 está estrechamente relacionado con el estadio de la federación internacional de ginecología y obstetricia (estadio FIGO) y el tamaño del tumor de CC [19]. En este estudio, por lo tanto, examinamos los efectos del eje SOX2 / CCAT1 / miR-185-3p / FOXP3 sobre la capacidad de proliferación y autorrenovación de las células madre CC.
Materiales y métodos
Aprobación de ética y consentimiento para participar
Los experimentos en los que participaron seres humanos se llevaron a cabo de conformidad con los principios expresados en la Declaración de Helsinki. El estudio fue aprobado por la Junta de Revisión Institucional del Primer Hospital de la Universidad de Jilin. Todos los participantes firmaron un documento de consentimiento informado.
Temas de estudio
Desde diciembre de 2016 a diciembre de 2018, se recolectaron 39 casos de tejidos CC y los tejidos normales adyacentes correspondientes de pacientes con CC y se conservaron en nitrógeno líquido. Los criterios de inclusión fueron los siguientes:(1) Los pacientes fueron confirmados como CC mediante patología de biopsias en canales transcervical y cervical, citología de frotis cervical, prueba de yodo cervical, espéculo vaginal y resección de vértebras cervicales. (2) Los pacientes no recibieron radioterapia ni quimioterapia 2 semanas antes de la operación. Los criterios de exclusión fueron los siguientes:(1) pacientes sometidos a radioterapia o quimioterapia, (2) pacientes en desacuerdo con la recolección de la muestra y (3) pacientes con trastornos del sistema inmunológico.
Selección y cultivo de células
Las líneas celulares CC (SiHa, HeLa, CaSki, HCC94 y C33A) y la línea celular H8 inmortalizada del epitelio cervical humano se compraron de Shanghai Bioleaf Biotech Co., Ltd. (Shanghai, China). Las células CC SiHa, HeLa y HCC94 se cultivaron en medio de Eagle modificado de Dulbecco (DMEM) con alto contenido de glucosa que contenía suero bovino fetal (FBS) al 10%, mientras que las células CaSki, C33A y H8 en el medio Roswell Park Memorial Institute (RPMI) -1640 contenía 10 % FBS (37 ℃ y 5% CO 2 ). Las células se separaron y se subcultivaron cada 2 días.
Clasificación de células madre CC (CD44 + Células HeLa)
Las células madre CC se separaron de la línea celular CC HeLa mediante cultivo en suspensión celular. Las células CC HeLa se cultivaron con medio de Dulbecco modificado de Iscove sin suero (IMDM) en una placa de Petri de adhesión ultrabaja durante 21 días con el medio intercambiado semicuantitativamente cada 3-5 días. Algunas células se suspendieron en esferas y se obtuvieron células formadoras de esferas HeLa (SFC). Se identificó y analizó la propiedad de las células esféricas. Las células se separaron con tripsina y se ajustaron a 1 × 10 6 células / mL. Las células se agregaron con anticuerpo CD44 y se clasificaron mediante citometría de flujo. Las células HeLa con CD44 positivo eran células madre tumorales HeLa, mientras que con CD44 negativo eran células no madre HeLa. Se cultivaron células madre CC en DMEM / F12 y se añadieron 20 ng / ml de factor de crecimiento de fibroblastos básico (bFGF), 20 ng / ml de factor de crecimiento epidérmico (EGF) y B27. El medio contenía penicilina y estreptomicina al 1% [20].
Tratamiento celular
CD44 + Las células HeLa se transfectaron con sh-SOX2, control negativo (NC) sh-SOX2, sh-CCAT1, sh-CCAT1 NC, miR-185-3p imita, imita NC, sh-CCAT1 y miR-185-3p inhibidor así como sh-CCAT1 e inhibidor NC. Todas las secuencias de oligonucleótidos fueron suministradas por GenePharma (Shanghai, China). Separadas por tripsina, las células se sembraron en una placa de 6 pocillos con 3 × 10 6 células / pocillo. Cuando se alcanzó una confluencia del 60%, las células se cambiaron a medio sin suero y se incubaron durante 1 h. La transfección fue facilitada por el reactivo de transfección Lipofectamine 2000 (Invitrogen, Carlsbad, California, EE. UU.).
Reacción en cadena de la polimerasa cuantitativa con transcripción inversa (RT-qPCR)
El ARN en tejidos y células fue extraído por Trizol (Invitrogen). El ARN (1 µg) se invirtió en ADNc mediante el kit RTasa del virus de la leucemia murina de Moloney (Invitrogen). Se añadió ADNc al sistema de PCR en tiempo real. Los cebadores fueron ideados por Shanghai Sangon Biotechnology Co., Ltd. (Shanghai, China) (Tabla 1). U6 fue el control de carga de miR-185-3p, mientras que la gliceraldehído fosfato deshidrogenasa (GAPDH) de SOX2, CCAT1 y FOXP3. Los resultados fueron analizados por 2 −ΔΔCt método.
Ensayo de transferencia Western
Se extrajo la proteína total en células y tejidos. La concentración de proteína se determinó mediante kits de ácido bicinconínico (AmyJet Scientific, Wuhan, Hubei, China). La proteína se mezcló con tampón de carga y se hirvió durante 5 min, seguido de un baño de hielo y centrifugación. La proteína se trató con electroforesis en gel de poliacrilamida-dodecilsulfato de sodio al 10% y se transfirió a una membrana. La membrana se bloqueó con leche desnatada al 5% durante 1 h, se probó con anticuerpos primarios SOX2 (1:1000, Jiangsu Rui sitan Co., Ltd., Jiangsu, China), FOXP3 (1:1000, Abcam Inc., Cambridge, MA , EE. UU.), GAPDH (1:1000, Cell Signaling Technology, Beverly, MA, EE. UU.) Y se volvieron a sondar con un anticuerpo secundario marcado con peroxidasa de rábano picante, cubierto por una película conservante y expuesto. Se adoptó para el desarrollo el generador de imágenes Bio-rad Gel Doc EZ (Bio-rad, California, EE. UU.). Las imágenes de proteínas se analizaron con el software ImageJ2x.
Ensayo del kit de recuento celular (CCK) -8
El ensayo CCK-8 se implementó con los kits (Beyotime, Shanghai, China). Celdas (1 × 10 4 ) se sembraron en una placa de 96 pocillos y se incubaron. Cultivadas durante 0, 24, 48 y 72 h, las células se agregaron con 10 μL / pocillo de solución de CCK-8 y se incubaron durante 1 h. El valor de densidad óptica se determinó con un lector de microplacas de longitud de onda completa Multiskan Spectrum a 450 nm. Se tomaron seis pozos para contar el valor medio. La curva de crecimiento celular se representó con el tiempo como ordenada y la viabilidad celular relativa como ordenada. El valor de densidad óptica representó la proliferación celular.
Citometría de flujo
Celdas (1 × 10 6 ) se centrifugaron a 1500 r / min, se suspendieron con 200 μL de tampón de unión, se incubaron con 5 μL de yoduro de propidio (PI) y 5 μL de isotiocianato de anexina V-fluoresceína (FITC) y se añadieron a su vez 400 μL de tampón. La tasa de apoptosis celular se verificó mediante un citómetro de flujo (BD Biosciences, NJ, EE. UU.).
Prueba de Scratch
Las células transfectadas se separaron y se prepararon en suspensión celular. Suspensiones de células (1 × 10 6 células / ml) se sembraron en una placa de 6 pocillos y se cultivaron hasta una confluencia del 80-90%. Los rasguños transversales y longitudinales en la placa de 6 pocillos se hicieron uniformemente con una punta de pipeta aséptica de 10 μl a lo largo de la regla. El medio original se reemplazó por medio completo y las células se cultivaron continuamente. La migración de las células se observó bajo un microscopio a las 0 hy 72 hy se fotografió en el mismo sitio.
Ensayo Transwell
Las células transfectadas se tripsinizaron, se centrifugaron a 1000 gy se aclararon con DMEM. Se colocó una cámara Transwell completamente hidratada en una placa de 24 pocillos que contenía FBS-DMEM al 10% (600 ml / pocillo). Suspensión celular (5 × 10 5 células / mL, 200 μL) en la cámara superior Transwell (recubierta con Matrigel 1:8 80 μL), mientras que 500 μL 20% FBS-DMEM en la cámara inferior. Las células se cultivaron continuamente durante 24 h, se fijaron con 500 μL de paraformaldehído (PFA) al 4% y se tiñeron con una solución de tinción de violeta cristal al 0,1%. Luego, las células en la superficie y el borde de la cámara superior se limpiaron con un hisopo de algodón. Se seleccionaron cinco campos al azar y las células se contaron con un microscopio Nikon Eclipse TE2000-S (Nikon, Japón).
Ensayo de formación de esferas
Las células se sembraron en una placa de 6 pocillos de baja adsorción que contenía medio de suspensión sin suero con 200 células / pocillo. Después de 2 w, se observó la tasa de formación de esferas de células con un microscopio Nikon Eclipse TE2000-S (Nikon), y la tasa de formación de esferas se calculó como el número promedio de esferas / número de células sembradas × 100%.
Ensayo de inmunoprecipitación de cromatina (ChIP)
El ensayo de ChIP se operó con el kit ChIP (Upstate, NY, EE. UU.). Se utilizaron anticuerpos IgG de conejo normal (12–370, Millipore, EE. UU.) SOX2 (1:1000, Re-stem Biotech, Jiangsu, China) para inmunoprecipitar el complejo de ADN-proteína reticulado. CD44 + Las células HeLa se fijaron con PFA al 1% y se incubaron para producir entrecruzamiento de ADN-proteína. Luego, el ADN se cortó en un fragmento de cromatina de 200 a 300 pb por ultrasonidos. El ADN de cromatina precipitado se recuperó y se analizó mediante RT-qPCR.
Ensayo de extracción de ARN
Los plásmidos de tipo salvaje (WT) y de tipo mutante (MUT) de miR-185-3p marcados con biotina (50 nM) se transfectaron en CD44 + Células HeLa, respectivamente. Las células se incubaron con lisado celular específico (Ambion, Austin, Texas, EE. UU.) Después de 48 h de transfección. Se subenvasó el lisado celular (50 ml). El lisado residual se incubó con perlas de estreptavidina M-280 (Sigma, St. Louis, MO, EE.UU.) prerrevestidas con ARNt libre de ARNasa y de levadura (Sigma). Seguido de eso, las células se limpiaron dos veces con lisado frío, tres veces con tampón bajo en sal y una vez con tampón alto en sal. Se estableció una sonda de miR-185-3p antagonista como NC. Trizol extrajo el ARN total y RT-qPCR evaluó el nivel de CCAT1.
Ensayo génico indicador de luciferasa dual
Los sitios de unión potenciales E1 y E2 de SOX2 en la región promotora CCAT1 fueron predichos por https://jaspar.genereg.net/. Se sintetizó la secuencia promotora CCAT1 que contenía SOX2 y el sitio de unión E1 de CCAT1, y se formaron los vectores CCAT1 3'UTR WT (E1-WT) y CCAT1 3'UTR MUT (E1-MUT). Los vectores se clonaron en pmirGLO (Beyotime). Después de eso, CCAT1-WT / pmirGLO o CCAT1-MUT / pmirGLO se cotransfectaron con sh-SOX2 o sh-SOX2 NC a CD44 + Células HeLa, respectivamente, durante 2 dy luego se lisaron. La actividad de la luciferasa se evaluó mediante el sistema de detección de luciferasa (Takara, Dalian, China).
El sitio web de bioinformática se utilizó para predecir y analizar los sitios de unión de CCAT1 y miR-185-3p. Los sitios de unión de CCAT1 y miR-185-3p se verificaron mediante un ensayo del gen indicador de luciferasa dual. Se compuso CCAT1 3'UTR que contiene el sitio de unión de miR-185-3p. CCAT1 3′UTR WT y CCAT1 3′UTR MUT se construyeron y cotransfectaron con imitaciones NC y miR-185-3p a CD44 + Células HeLa durante 2 d. Luego, las células se lisaron y se probó la actividad luciferasa mediante el sistema de detección de luciferasa (Takara). Se aplicó el mismo método para verificar la relación de focalización entre miR-185-3p y FOXP3.
Análisis estadístico
Todos los datos fueron evaluados por el software SPSS 21.0 (IBM Corp. Armonk, NY, EE. UU.). Los datos de medición se indicaron como media ± desviación estándar. Se aplicó la prueba t para determinar la discrepancia entre dos grupos y el análisis de varianza unidireccional (ANOVA) seguido de la prueba de comparaciones múltiples de Tukey para determinar la discrepancia entre los grupos. La variable de clasificación se evaluó mediante la prueba exacta de Fisher. A p un valor inferior a 0,05 se consideró significativo.
Resultados
Disminuye la expresión de miR-185-3p, mientras que las expresiones SOX2, CCAT1 y FOXP3 aumentan en los tejidos CC, y las expresiones SOX2 y CCAT1 están vinculadas al tamaño del tumor, al ganglio linfático Metástasis (LNM) y estadio FIGO
Al detectar el papel del eje SOX2 / CCAT1 / miR-185-3p / FOXP3 en la capacidad de proliferación y autorrenovación de las células madre CC, se detectaron expresiones de miR-185-3p, SOX2, CCAT1 y FOXP3 en tejidos CC y tejidos normales adyacentes. probado por RT-qPCR y ensayo de transferencia de Western. Se manifestó que (Fig. 1a-c) la expresión de miR-185-3p se redujo, mientras que las expresiones de SOX2, CCAT1 y FOXP3 aumentaron en los tejidos CC.
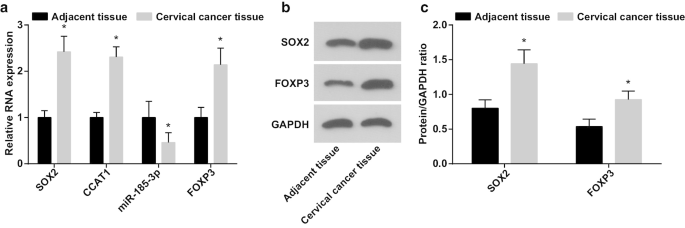
La expresión de miR-185-3p disminuye, mientras que las expresiones de SOX2, CCAT1 y FOXP3 aumentan en los tejidos CC. un Comparación de la expresión de ARNm de SOX2, CCAT1, miR-185-3p y ARNm de FOXP3 en CC y tejidos normales adyacentes. b Bandas proteicas de expresión proteica SOX2 y FOXP3 en CC y tejidos normales adyacentes. c Comparación de la expresión de la proteína SOX2 y FOXP3 en CC y tejidos normales adyacentes. * p <0,05 frente a tejidos normales adyacentes. Los datos de medición se representaron como media ± desviación estándar y las comparaciones entre dos grupos se evaluaron mediante la prueba t
Se analizó la relación entre la expresión de SOX2 / CCAT1 y las características clínico-patológicas de CC (Tabla 2). SOX2 y CCAT1 sobreexpresados en CC se relacionaron con el tamaño del tumor, el estadio LNM y FIGO, lo que indica que las expresiones de SOX2 y CCAT1 fueron más altas en pacientes con tumores de mayor tamaño, LNM y estadio FIGO avanzado de los pacientes con CC.
La expresión de miR-185-3p se reduce, mientras que las expresiones SOX2, CCAT1 y FOXP3 aumentan en CD44 + Células HeLa
A continuación, se probaron las expresiones de miR-185-3p, SOX2, CCAT1 y FOXP3 en las líneas celulares H8 y CC de células inmortalizadas en epitelio cervical humano SiHa, CasKi, HeLa, HCC94 y C33A. Se sugirió que (Fig. 2a-c) la expresión de miR-185-3p estaba degradada, mientras que SOX2, CCAT1 y FOXP3 estaban elevadas en las líneas celulares CC. Entre ellos, SOX2 / CCAT1 / miR-185-3p / FOXP3 en células HeLa tuvieron la mayor diferencia de expresión de las células H8; por tanto, las células HeLa se seleccionaron y clasificaron; Los HeLa-SFC se obtuvieron mediante cultivos en suspensión celular y la expresión de CD44 en la superficie celular antes y después de la clasificación se identificó mediante citometría de flujo. Los resultados destacaron que la tasa positiva de CD44 en HeLa-SFC fue marcadamente más alta que antes de la clasificación, lo que sugiere una clasificación exitosa de las células madre CC (Fig. 2d). Las células madre clasificadas se denominaron CD44 + Células HeLa. Luego, se probaron las expresiones de miR-185-3p, SOX2, CCAT1 y FOXP3 en HeLa y CD44 + Células HeLa (fig. 2e-g). Se manifestó que las expresiones SOX2, CCAT1 y FOXP3 estaban reguladas al alza y que miR-185-3p estaba regulado a la baja en CD44 + Células HeLa.
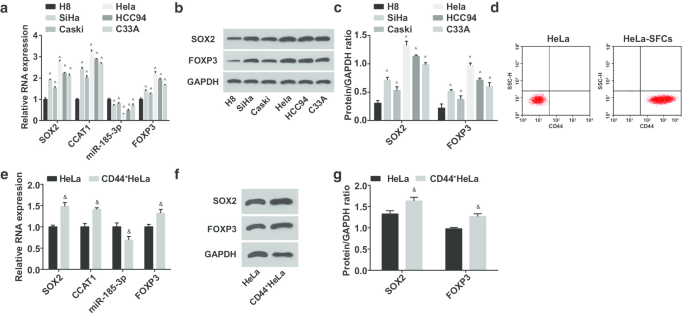
La expresión de miR-185-3p se reduce, mientras que las expresiones SOX2, CCAT1 y FOXP3 se elevan en CD44 + Células HeLa. un Comparación de la expresión de ARNm de SOX2, CCAT1, miR-185-3p y ARNm de FOXP3 en líneas celulares H8 y CC. b Bandas proteicas de expresión proteica SOX2 y FOXP3 en líneas celulares H8 y CC. c Comparación de la expresión de proteínas SOX2 y FOXP3 en líneas celulares H8 y CC. d Detección de la tasa de expresión de CD44 en células HeLa antes y después de la clasificación por citometría de flujo. e Comparación de la expresión de ARNm de SOX2, CCAT1 y miR-185-3p entre células HeLa y CD44 + Células HeLa. f Banda de proteína de expresión de la proteína SOX2 en células HeLa y CD44 + Células HeLa. g Comparación de la expresión de la proteína SOX2 entre células HeLa y CD44 + Células HeLa. ^ p <0,05 frente a células H8. y p <0,05 frente a células HeLa. Los datos de medición se representaron como media ± desviación estándar, las comparaciones entre dos grupos se evaluaron mediante la prueba t, y las comparaciones entre múltiples grupos se evaluaron mediante ANOVA de una vía seguida de la prueba post hoc de Tukey
La regulación a la baja de SOX2 y la regulación a la baja de CCAT1 disminuyen la tasa de proliferación, migración, invasión y formación de esferas y aumentan la apoptosis de CD44 + Células HeLa
Posteriormente, SOX2 y CCAT1 fueron silenciados en CD44 + Células HeLa para explorar sus efectos sobre la proliferación y autorrenovación de las células madre CC. Detectada por el ensayo CCK-8, citometría de flujo, prueba de raspado, ensayo Transwell y experimento de formación de esferas, se suprimieron la tasa de proliferación, migración, invasión y formación de esferas y la apoptosis de CD44 + Las células HeLa fueron promovidas por la inhibición de SOX2 y CCAT1 (Fig. 3a-i). Fue indicativo de que silenciar SOX2 o CCAT1 inhibía la proliferación y la autorrenovación de las células madre CC.
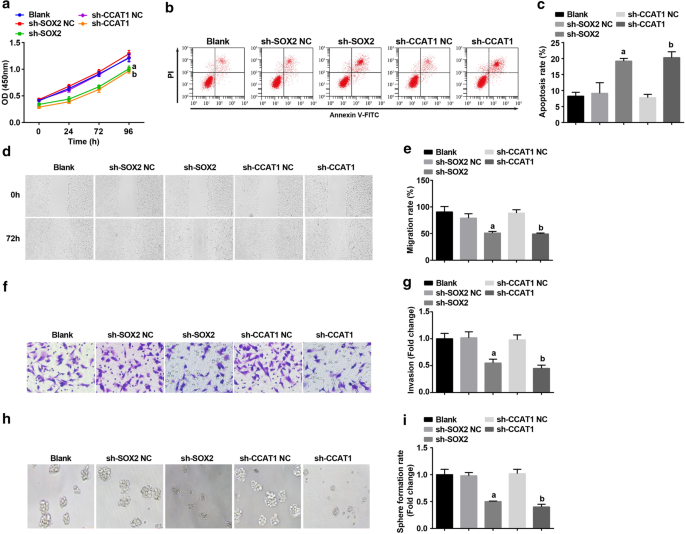
La regulación a la baja de SOX2 y la regulación a la baja de CCAT1 disminuyen la tasa de proliferación, migración, invasión y formación de esferas y aumentan la apoptosis de CD44 + Células HeLa. un El ensayo CCK-8 probó la curva de crecimiento celular en células tratadas con sh-CCAT1 o sh-SOX2. b La citometría de flujo detectó apoptosis celular en células tratadas con sh-CCAT1 o sh-SOX2. c Comparación de la tasa de apoptosis celular en células tratadas con sh-CCAT1 o sh-SOX2. d Migración celular en células tratadas con sh-CCAT1 o sh-SOX2 probadas mediante prueba de rayado. e Comparación de la migración celular en células tratadas con sh-CCAT1 o sh-SOX2. f Detección de la capacidad de invasión de células en células tratadas con sh-CCAT1 o sh-SOX2 mediante ensayo Transwell. g Comparación de la capacidad de invasión de células tratadas con sh-CCAT1 o sh-SOX2. h El experimento de formación de esferas probó la capacidad de autorrenovación en células tratadas con sh-CCAT1 o sh-SOX2. yo Comparación de la tasa de formación de esferas en células tratadas con sh-CCAT1 o sh-SOX2. a p <0,05 frente al grupo sh-SOX2 NC. b p <0,05 frente al grupo sh-CCAT1 NC. Los datos de medición se representaron como media ± desviación estándar, y las comparaciones entre múltiples grupos se evaluaron mediante ANOVA de una vía seguida de la prueba post hoc de Tukey
El miR-185-3p agotado invierte el papel de la regulación descendente de CCAT1 en CD44 + Células HeLa
Luego, examinamos si miR-185-3p estaba involucrado en el proceso de CCAT1 que regula la proliferación y autorrenovación de las células madre CC. CD44 + Se transfectó HeLa con el imitador de miR-185-3p o se cotransfectó con sh-CCAT1 y el inhibidor de miR-185-3p. Los resultados mostraron que la regulación positiva de miR-185-3p redujo notablemente la tasa de proliferación, migración, invasión y formación de esferas, mientras que aumentó la tasa de apoptosis de CD44 + Células HeLa. Las células tratadas con el inhibidor de miR-185-3p podrían revertir el papel de CCAT1 regulado negativamente en la proliferación, migración, invasión, apoptosis y formación de esferas celulares de CD44 + Células HeLa (Fig. 4a-i).
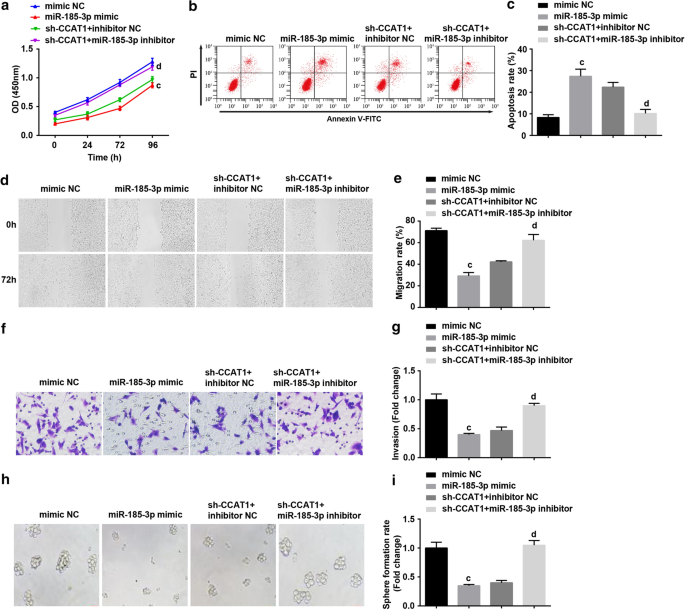
El miR-185-3p sobreexpresado suprime la tasa de proliferación, migración, invasión y formación de esferas y aumenta la apoptosis de CD44 + Células HeLa. un El ensayo CCK-8 probó la curva de crecimiento celular en células tratadas con el mímico de miR-185-3p. b La citometría de flujo detectó la apoptosis celular en células tratadas con míR-185-3p. c Comparación de la tasa de apoptosis celular en células tratadas con míR-185-3p. d Migración celular en células tratadas con el mímico de miR-185-3p probado mediante prueba de raspado. e Comparación de la migración celular en células tratadas con míR-185-3p mimic. f Detección de la capacidad de invasión de células tratadas con míR-185-3p por ensayo de Transwell. g Comparación de la capacidad de invasión de células tratadas con mimético de miR-185-3p. h El experimento de formación de esferas probó la capacidad de autorrenovación en células tratadas con el mímico de miR-185-3p. yo Comparación de la tasa de formación de esferas en células tratadas con míR-185-3p mimic. a p <0,05 frente al grupo sh-SOX2 NC. b p <0,05 frente al grupo sh-CCAT1 NC. c p <0,05 frente al grupo NC mímico. d p <0,05 frente al grupo NC inhibidor de sh-CCAT1 +. Los datos de medición se representaron como media ± desviación estándar, y las comparaciones entre múltiples grupos se evaluaron mediante ANOVA de una vía seguida de la prueba post hoc de Tukey
El SOX2 de baja expresión y CCAT1 de baja expresión disminuyen la expresión de FOXP3 y aumentan la expresión de miR-185-3p en CD44 + Células HeLa
Posteriormente, examinamos la expresión de SOX2 / CCAT1 / miR-185-3p / FOXP3 en CD44 + Las células HeLa después de la transfección de sh-SOX2, sh-CCAT1, miR-185-3p imitan y cotransfección de sh-CCAT1 y el inhibidor de miR-185-3p. Las expresiones de SOX2, CCAT1 y FOXP3 se redujeron, mientras que la expresión de miR-185-3p se elevó en las células tratadas con sh-SOX2. Las expresiones de CCAT1 y FOXP3 se redujeron y la expresión de miR-185-3p mejoró en las células tratadas con sh-CCAT1. La expresión de miR-185-3p se elevó y la expresión de FOXP3 disminuyó en las células introducidas con el mímico de miR-185-3p. La expresión de FOXP3 se elevó y la expresión de miR-185-3p se redujo en las células transfectadas sucesivamente con el inhibidor de sh-CCAT1 y miR-185-3p (Fig. 5a-d).
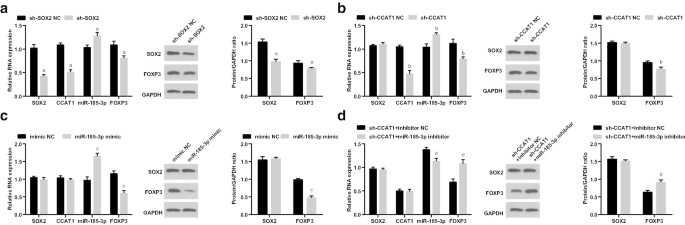
SOX2 de baja expresión y CCAT1 de baja expresión disminuyen la expresión de FOXP3 y aumentan la expresión de miR-185-3p en CD44 + Células HeLa. un Expresión de SOX2 / CCAT1 / miR-185-3p / FOXP3 en los grupos sh-SOX2 NC y sh-SOX2. b Expresión de SOX2 / CCAT1 / miR-185-3p / FOXP3 en los grupos sh-CCAT1 NC y sh-CCAT1. c Expresión de SOX2 / CCAT1 / miR-185-3p / FOXP3 en los grupos mímicos NC y miR-185-3p. d Expresión de SOX2 / CCAT1 / miR-185-3p / FOXP3 en los grupos inhibidor de sh-CCAT1 + NC y sh-CCAT1 + miR-185-3p. a p <0,05 frente al grupo sh-SOX2 NC. b p <0,05 frente al grupo sh-CCAT1 NC. c p <0,05 frente al grupo NC mímico. d p <0,05 frente al grupo NC inhibidor de sh-CCAT1 +. Los datos de medición se representaron como media ± desviación estándar, y las comparaciones entre múltiples grupos se evaluaron mediante ANOVA de una vía seguida de la prueba post hoc de Tukey
SOX2 se une a CCAT1, lo que afecta la expresión de miR-185-3p, y FOXP3 es un gen objetivo de miR-185-3p
Los sitios de unión potenciales del factor de transcripción de la región promotora CCAT1 fueron predicados y analizados por el sitio web https://jaspar.genereg.net/, y mostró que SOX2 y CCAT1 tenían sitios de unión potenciales en la región promotora CCAT1 (Fig. 6a). ChIP-qPCR informó que (Fig.6b):En contraste con el grupo IgG, más fragmentos del promotor CCAT1 se enriquecieron en el grupo SOX2 en el sitio de unión E1, lo que demostró que SOX2 se unió al promotor CCAT1 en el sitio E1 y SOX2 fue involucrados en la regulación de CCAT1. El ensayo del gen indicador de luciferasa dual mostró que (Fig. 6c):La actividad de luciferasa se suprimió en las células cotransfectadas con sh-SOX2 y E1-WT, lo que indica que SOX2 podría unirse a CCAT1.
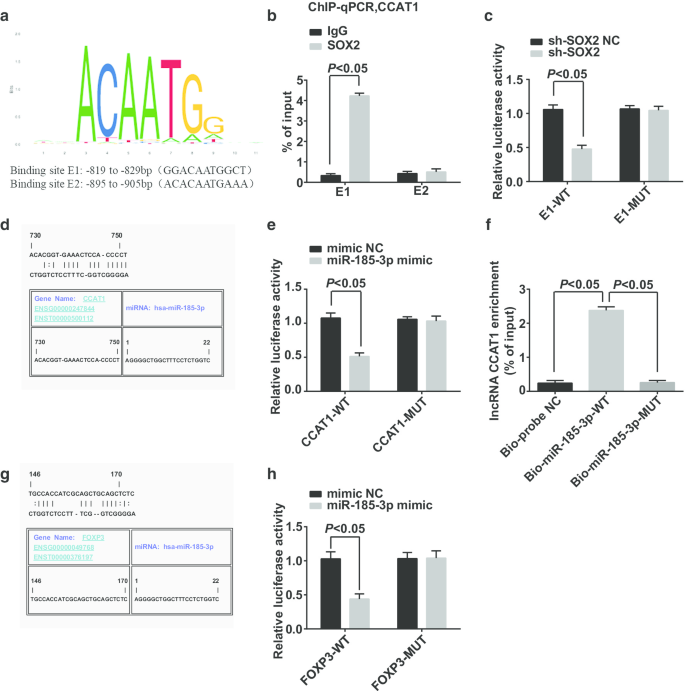
SOX2 puede unirse a CCAT1, lo que afecta la expresión de miR-185-3p, y FOXP3 es un gen diana de miR-185-3p. un Predicción de sitios de unión en regiones promotoras SOX2 y CCAT1 por sitios bioinformáticos. b El experimento ChIP-qPCR verificó la relación de unión entre SOX2 y CCAT1. c El sitio de unión de SOX2 y CCAT1 verificado por ensayo de gen indicador de luciferasa dual. d Predicción de sitios de unión en CCAT1 y miR-185-3p por sitios bioinformáticos. e Verificación de la unión de CCAT1 y miR-185-3p mediante ensayo de gen indicador de luciferasa dual. f La relación de unión entre CCAT1 y miR-185-3p en células verificada por ensayo de reducción de ARN. g Predicción de la relación de focalización entre miR-185-3p y FOXP3 por el sitio web de bioinformática. h Identificación de la relación de dirección entre miR-185-3p y FOXP3 mediante el ensayo del gen indicador de luciferasa dual. Los datos de medición se representaron como media ± desviación estándar y las comparaciones entre dos grupos se evaluaron mediante la prueba t
El sitio web de Jefferson predijo que CCAT1 podría unirse a miR-185-3p (Fig. 6d). El ensayo del gen indicador de luciferasa dual informó que (Fig. 6e) la actividad de luciferasa disminuyó en las células introducidas con el mímico de miR-185-3p y CCAT1-WT, lo que sugiere que miR-185-3p podría unirse a CCAT1. Se utilizó un ensayo de reducción de ARN para verificar si CCAT1 podía unirse con miR-185-3p. Los resultados revelaron que (Fig.6f) el nivel de enriquecimiento de CCAT1 en células tratadas con Bio-miR-185-3p-WT aumentó notablemente, mientras que el nivel de enriquecimiento de CCAT1 en células tratadas con Bio-miR-185-3p-MUT mostró ninguna diferencia significativa. Este resultado demostró que CCAT1 podría adsorber miR-185-3p, afectando así la expresión de miR-185-3p.
La relación objetivo entre miR-185-3p y FOXP3 fue predicha por el sitio web de Jefferson (Fig. 6g). El ensayo del gen indicador de luciferasa dual verificó que (Fig. 6h) la actividad relativa de luciferasa de las células se redujo drásticamente después de que el imitador FOXP3-WT y miR-185-3p cotransfectaran a CD44 + Las células HeLa, mientras que FOXP3-MUT cotransfectadas con el imitador de miR-185-3p no afectó la actividad luciferasa relativa de las células, lo que sugiere que miR-185-3p se dirigió a FOXP3.
Discusión
La CC es la cuarta neoplasia maligna frecuente en mujeres en el mundo, seguida de los cánceres de mama, colon y pulmón [3]. Se ha informado de que las células CC con expresión SOX2 positiva muestran las características de las células madre cancerosas [21]. Un estudio ha informado que CCAT1 es un lncRNA oncogénico esencial relacionado con CC y ejerce un papel facilitador en el crecimiento e invasión de células CC [11]. Otro estudio revela que miR-185-3p podría predecir la radiosensibilidad del carcinoma nasofaríngeo y regular el crecimiento y la apoptosis de las células cancerosas [22]. Se ha informado de que el autoanticuerpo circulante contra FOXP3 refleja el progreso continuo de la lesión cervical y puede ser un biomarcador potencial para el pronóstico temprano de CC [23]. El estudio actual fue diseñado para explorar cómo el eje SOX2 / CCAT1 / miR-185-3p / FOXP3 influyó en la capacidad de proliferación y autorrenovación de las células madre CC.
Según nuestros hallazgos, las expresiones de SOX2 y CCAT1 aumentaron en los tejidos y células CC que estaban vinculados al tamaño del tumor, LNM y FIGO avanzado. La regulación a la baja de SOX2 y CCAT1 funcionalmente probada disminuyó la proliferación, migración, invasión y número de células esféricas y aumentó la apoptosis de las células madre CC. Al igual que en nuestro estudio, las tendencias de expresión de SOX2 aumentan en CC [24, 25]. Moreover, SOX2 expression is also up-regulated in CC cells derived from cancer stem cells [26]. Overexpressed SOX2 was suggested to link with clinicopathological characteristics of patients with several types of cancer, not limited to CC. For example, it was suggested that up-regulated SOX2 shows in cervical squamous cell carcinoma patients staged in FIGO I-II [27]. Moreover, SOX2 expression is linked to LNM in oral squamous cell carcinoma [28]. When it comes to the molecular function of SOX2 for cancer progression, there is an observational work presenting that down-regulated SOX-2 suppresses cell migration and invasion of cervical squamous cell carcinoma [29]. Meanwhile, another research has offered a proof that up-regulated SOX2 enhances CC cell clonogenicity, proliferation and tumorigenicity in vitro and in vivo than control cells [30].
Concerning to the regulatory relation between SOX2 and CCAT1, an existed study has presented that silencing SOX2 markedly reduces CCAT1 mRNA level [31]. As to the role of CCAT1 in cancers, a study has showed that CCAT1 expression is markedly elevated in CC tissues versus in the adjacent normal tissues [11, 12]. Of note, CCAT1 overexpression in CC is positively related to the tumor size [12]. In terms of the role of CCAT1 in cancer cell activity, there is a research highlighting that overexpressed CCAT1 accelerates CC cell proliferation, colony formation and invasion [11]. Interestingly, a previous research has demonstrated that the cell viability, invasive and migratory abilities are declined via knocking down CCAT1 [12]. Anyway, the functional effect of SOX2 and CCAT1 in other cancers was similar to that in CC.
Afterward, our research revealed that CCAT1 could bind to miR-185-3p, the down-regulated CCAT1 in CC and overexpressing miR-185-3p suppressed the proliferation and self-renewal abilities of CC stem cells. It is reported that CCAT1 and miR-185-3p are negatively correlated [13]. Furthermore, a result reported that a reduction is seen in miR-185-3p expression in radioresistant nasopharyngeal carcinoma cases [22]. Regarding to the suppressive function of miR-185-3p in cancer cell aggressiveness, a study has revealed that up-regulation of miR-185-3p suppresses the invasive and metastatic properties of nasopharyngeal carcinoma cells [32]. Furthermore, Zou et al. have suggested that restored miR-185 represses breast cancer cell growth and invasion [33]. There is a article finding that up-regulation of miR-185 declines the proliferation, invasion and colony formation capacities of non-small cell lung cancer cells in vitro [34]. It is presented that in vitro cell proliferation, invasion and migration as well as in vivo tumor growth are suppressed via miR-185-overexpressing in non-small cell lung cancer cells [35]. From those studies, the anti-tumor role of miR-185-3p in the present study was consistent with previous researches.
To proceed, we unveiled that miR-185-3p targeted FOXP3, the overexpressed gene in CC to regulate CC stem cell activities. In fact, FOXP3, the regulator of SOX2 cancer stem-like cell marker in colon cancer [36], has been investigated in CC, showing an up-regulation in CC cells [19] [37]. It was evidenced that elevating FOXP3 promotes the formation of tumor spheres and stimulates the stemness of non-small cell lung cancer cells [38].
Conclusion
Collectively, we explored for the first time that SOX2 transcription could activate CCAT1, thereby inhibiting miR-185-3p and regulating FOXP3 to promote the proliferation and self-renewal of CC stem cells, which is a potential avenue to treat CC. Additionally, however, limitations in this present study still exist in the relatively small trial size in the designed experiment. Thus, clinical researches might be further carried out to detect the efficacy for the treatment of CC.
Disponibilidad de datos y materiales
No aplica.
Nanomateriales
- Demostración de un biosensor flexible basado en grafeno para la detección rápida y sensible de células de cáncer de ovario
- Nanopartículas para la terapia del cáncer:avances y desafíos actuales
- Administración de medicamentos basados en células para aplicaciones de cáncer
- Las nanotecnologías desactivan las células madre cancerosas
- Efecto de la distribución de nanopartículas de oro en TiO2 sobre las características ópticas y eléctricas de las células solares sensibilizadas por colorante
- Los andamios nHAC / PLGA híbridos con óxido de grafeno facilitan la proliferación de células MC3T3-E1
- Estudios TEM y STEM sobre las morfologías transversales de películas antirreflectantes de SiO2 de banda ancha de doble / triple capa
- Fabricación y caracterización de nanoclips de ZnO mediante el proceso mediado por poliol
- Dielectroforesis de ondas de frecuencia modulada de vesículas y células:cambios de sentido periódicos en la frecuencia de cruce
- Influencia de nanopartículas de agricultura con diferentes tamaños y concentraciones incrustadas en una capa compacta de TiO2 en la eficiencia de conversión de las células solares de perovskita
- Factores que afectan el éxito y la seguridad del proceso de punzonado



