Biosensor ultrasensible para la detección de ADN de Vibrio cholerae con nanoesferas compuestas de poliestireno-co-ácido acrílico
Resumen
Un biosensor electroquímico ultrasensible para la determinación de Vibrio cholerae patógenos ( V . cholerae ) El ADN se desarrolló a partir de una matriz portadora de ADN compuesta de nanoesferas de látex de poliestireno-co-ácido acrílico (PSA) y nanopartículas de oro (PSA-AuNP). Se usó voltamperometría diferencial de pulsos (DPV) usando un marcador de oligonucleótidos de antraquninona electroactivo para medir la respuesta del biosensor. La carga de nanopartículas de oro (AuNP) en el electrodo de partículas de látex de ADN ha amplificado significativamente la corriente faradaica de hibridación del ADN. Junto con el uso de una sonda informada, el biosensor ha demostrado una alta sensibilidad. El biosensor de ADN produjo un rango de respuesta lineal amplio y reproducible para el ADN objetivo de 1.0 × 10 −21 a 1.0 × 10 −8 M (desviación estándar relativa, RSD =4.5%, n =5) con un límite de detección (LOD) de 1.0 × 10 −21 M ( R 2 =0,99). El biosensor obtuvo valores de recuperación satisfactorios entre 91 y 109% ( n =3) para la detección de V . cholerae ADN en muestras enriquecidas y podría reutilizarse para seis ensayos de ADN consecutivos con un valor de RSD de repetibilidad del 5% ( n =5). La respuesta del biosensor electroquímico fue estable y se mantuvo al 95% de su respuesta original hasta 58 días de período de almacenamiento.
Antecedentes
Vibrio cholerae , un patógeno transmitido por los alimentos, puede causar epidemias de cólera en humanos a través de la condición de diarrea acuosa aguda. El brote de cólera sigue siendo un problema grave en algunas partes del mundo, p. Ej. Asia y África, y conduce a un nivel socioeconómico bajo [1, 2, 3, 4]. Este patógeno entérico es una de las principales causas de morbilidad y mortalidad, especialmente en los países en desarrollo [5]. La epidemia y la pandemia de cólera en varias regiones son causadas principalmente por V. cholerae serogrupos O1 y O139 [1, 2, 6]. V. cholerae El serogrupo O1 tiene dos serotipos principales, a saber, Inaba y Ogawa, que pueden alternar entre las epidemias de cólera. El tercer serotipo, Hikojima, también existe, pero es raro e inestable. Los genes responsables de la biosíntesis del antígeno O1 se han denominado rfb. La mutación que define los serotipos Inaba y Ogawa es una única mutación por deleción en el gen rfbT [7]. Sin embargo, también se ha informado que los brotes ocasionales transmitidos por alimentos en seres humanos con diarrea grave son causados por V no O1 / no O139 V. cholerae a través de la ingestión de mariscos poco cocidos [8] o la exposición a un medio acuático contaminado [9]. La primera epidemia de V. cholerae O139 ocurrió en 1992 en Bangladesh e India y luego se extendió rápidamente a otros países del sudeste asiático [10]. En todo el mundo, en 2005, se notificaron a la Organización Mundial de la Salud (OMS) un total de 131.943 casos de cólera y 2272 muertes [11].
La búsqueda de un método eficaz de seguimiento o diagnóstico de V. cholerae bacterias es imprescindible para controlar la epidemia de cólera. Identificación tradicional de V. cholerae a menudo se logra mediante el aislamiento y la selección de las bacterias, donde implica un enriquecimiento previo en agua de peptona alcalina (APW) seguido del aislamiento de V. cholerae en el medio de cultivo tiosulfato, citrato, sal biliar, sacarosa, agar (TCBS) e identificación mediante prueba de aglutinación en portaobjetos con antisueros específicos [12]. Sin embargo, esta técnica requiere mucho tiempo y trabajo, y el resultado obtenido varios días después habría significado un retraso en el diagnóstico clínico y el tratamiento del paciente. Método molecular que implica amplificación por PCR para la detección de V. cholerae [13] ha reducido el tiempo de diagnóstico [14], pero el método de PCR requiere una infraestructura costosa y profesional capacitada que es difícil de realizar en países con pocos recursos. Se han informado pruebas de diagnóstico rápido basadas en el principio de inmunocromatografía para la detección discreta o simultánea de V. cholerae serogrupos O1 y O139. Algunas otras técnicas basadas en inmunoensayos utilizadas para la detección de V. cholerae son tales como ensayo inmunoabsorbente ligado a enzimas (ELISA), coaglutinación, inmunofluorescencia y microbalanza de cristal de cuarzo (QCM). Sin embargo, la mayoría de estas técnicas requieren instrumentación sofisticada, un tiempo de ensayo prolongado y personal altamente calificado con conocimientos técnicos detallados [15,16,17,18,19,20].
Los métodos electroquímicos han atraído una atención considerable en la detección de ácidos nucleicos debido a su alta sensibilidad, especificidad, simplicidad y protocolo económico, así como detección rápida y compatible con la tecnología de microfabricación [21, 22]. Además, el método electroquímico que se combina con tecnologías de miniaturización se puede utilizar para el análisis descentralizado in situ, por ejemplo, el dispositivo biosensor de ADN basado en chips de microfluidos, que es muy útil para entornos prácticos [23]. Existe una amplia gama de electrodos utilizados en las mediciones electroquímicas, como el electrodo de carbono vítreo (GCE), el electrodo de pasta de carbono (CPE), el electrodo de oro y el electrodo de platino. Recientemente, los estudios se han concentrado en el uso de electrodos serigrafiados (SPE) debido a algunas de sus propiedades únicas, como proporcionar una baja corriente de fondo y una amplia ventana de potencial, rentable ya que la tinta de carbón es económica y puede producirse en masa. .
Hay algunos métodos electroquímicos reportados para la detección de V. cholerae consistía en una serie de pasos complejos. Un electroquímico V. cholerae genosensor informado por Liew et al. [24] utilizó un método de adsorción electroquímica para inmovilizar la sonda de ADN en el SPE de carbono. Las partículas de PSA multicapa modificadas con AuNPs liofilizadas con polielectrolitos formaron un bioconjugado con avidina para funcionar como marcador informador en el ensayo de hibridación de ADN en sándwich. Sin embargo, fue necesaria la adición de estabilizador de sorbitol para preservar los bioconjugados de PSA-AuNPs-avidina para alargar el período operativo del biosensor de ADN hasta 30 días. Electroquímico enzimático V. cholerae El biosensor de ADN ha sido diseñado recientemente por Yu et al. [25], mediante el cual la sonda de ADN marcada con fosfatasa alcalina conjugada con anti-fluoresceína tiolada (anti-FCAP) se unió a la SPE de oro a través de la química del oro-tiol. El ADN diana se marcó con una fluoresceína universal para permitir el reconocimiento de la hibridación del ADN logrado mediante la conversión enzimática de α-naftilfosfato en α-naftol electroactivo. No obstante, este esquema de detección requirió un largo tiempo de ensayo de aproximadamente 95 min para la hibridación del ADN, marcando los híbridos de ADN con enzima funcional seguido de incubación del electrodo en sustrato de α-naftilfosfato electroinactivo antes de poder realizar una medición amperométrica. Low y los miembros del equipo [26] desarrollaron otro biosensor de ADN electroquímico enzimático basado en SPE de carbono recubierto de avidina conjugado con una sonda de ADN biotinilado [26]. También se usó una sonda indicadora marcada con digoxigenina (DIG) en esta estrategia de doble hibridación que flanqueaba la secuencia de ADNc. Se empleó anti-DIG (anti-DIG-HRP) ligado a peroxidasa de rábano picante como marca electroquímica, que podría catalizar simultáneamente la oxidación de 3,3 ', 5,5'-tetrametilbencidina (TMB) con la reducción de H 2 O 2 para producir una transferencia de electrones a la superficie del electrodo para la transducción electroquímica del evento de hibridación del ADN. Patel et al. [22] para la detección rápida de V. cholerae y se utilizó azul de metileno como indicador de hibridación de ADN. Sin embargo, el rango de detección lineal del sistema se limita a niveles de μM, lo que limita su aplicación en muestras clínicas.
La nanopartícula de látex y oro se ha empleado previamente como marca de hibridación de ADN mediante la unión de avidina / biotina a la sonda de ADN en la detección de V. cholerae [24], patógeno de peces Aphanomyces invadans [27], E. coli [28], e hibridación inespecífica de ADN [29], mediante la cual las esferas de látex se recubrieron con una capa múltiple de polielectrolito antes de que los coloides cargados negativamente de nanopartículas de oro se unieran electrostáticamente. Kawde y Wang [29] unieron las partículas de látex de PSA a una sonda indicadora de ADN que se utilizaría como marca de hibridación de ADN mediante la carga de partículas de látex recubiertas de estreptavidina con AuNP recubiertas de biotina. Kuan y col. [24, 27] y Liew et al. [24, 27] también informó del mismo método de unión avidina-biotina utilizando conjugados de sonda de oro-PSA-ADN. Pinijsuwan y col. [28] informaron sobre el uso de un método electrostático para la carga de partículas de PSA unidas a la sonda indicadora de ADN y las partículas de PSA recubiertas de oro de polielectrolito se utilizaron como marca para la hibridación que amplificó la respuesta de la corriente DPV.
En este estudio, informamos sobre un enfoque diferente de inmovilización de ADN que utiliza nanopartículas de látex y oro como sustrato de inmovilización de la sonda de ADN para desarrollar un sistema de detección altamente sensible para V. cholerae ADN. La inmovilización del ADN se realizó con un procedimiento muy simple y rápido utilizando la química de clorhidrato de 1-etil-3- (3-dimetilaminopropil) carbodiimida / N-hidroxisuccinimida (EDC / NHS) como reactivo de acoplamiento para mejorar la eficiencia de inmovilización [30] en el látex carboxilado superficie de la partícula. La detección de hibridación de ADN se basó en el ensayo de tipo sándwich, que implicó una reacción de hibridación entre la sonda de ADN inmovilizada y la secuencia diana seguida de una sonda de señal / informadora. Se empleó sal sódica monohidratada del ácido antraquinona-2-sulfónico (AQMS) como marca electroquímica para controlar el evento de hibridación. Las partículas de látex de tamaño submicrónico propuestas mejoraron la capacidad de unión de la sonda de ADN y la sensibilidad del biosensor de ADN se mejoró con la incorporación de nanopartículas de oro altamente conductoras (AuNP). El biosensor de ADN demostró una sensibilidad excepcional para la detección de V. cholerae ADNc y límite de detección extremadamente bajo a niveles zeptomolares en comparación con la tecnología avidina-biotina informada hasta ahora [24, 26].
Métodos
Productos químicos y reactivos
El estireno (St) y el ácido acrílico (AA) se adquirieron de Fluka. HAuCl 4 · 3H 2 O, citrato trisódico deshidratado, dodecilsulfato de sodio (SDS), clorhidrato de 1-etil-3- (3-dimetilaminopropil) carbodiimida (EDC) y N-hidroxisuccinimida (NHS) se obtuvieron de Sigma-Aldrich. El persulfato de amonio (APS), el ácido bromhídrico y el bromo fueron suministrados por Riedel-De Haën, Ajax Finechem y Panreac, respectivamente. Todas las soluciones químicas se prepararon con agua desionizada. Tanto los oligonucleótidos sintéticos diana de 30 bases como los que no coinciden se obtuvieron de Bio Service Unit (NSTDA). El ADN no complementario (ncDNA) y la sonda de señal eran de Sigma y la sonda de captura modificada con 5'-amino era de Bioneer. La sonda de captura se preparó en 0,05 M de tampón de fosfato de potasio (pH 7), mientras que las soluciones de ADN diana, diana no coincidente, sonda indicadora y ADN no complementario se prepararon en tampón de fosfato de sodio (0,05 M, pH 7). Las secuencias de oligonucleótidos utilizadas en el presente estudio se muestran en la Tabla 1.
Aparato
La medición electroanalítica se realizó con un potenciostato / galvanostato (Autolab PGSTAT12, Metrohm) equipado con el software GPES (4.0.007). Los experimentos voltamétricos se realizaron con un sistema convencional de tres electrodos que consta de un electrodo de trabajo serigrafiado (SPE) con pasta de carbón (Quasense, Bangkok, Tailandia), un electrodo de referencia Ag / AgCl (3 M de KCl) y una varilla de platino ( 2 mm de diámetro) contraelectrodo. La técnica de voltamperometría diferencial de pulso (DPV) se utilizó para todas las investigaciones electroquímicas a un potencial de paso de 0,02 V y una velocidad de exploración de 0,5 V / s de 0,0 a +1,0 V en 4,5 ml de tampón de medición (0,05 M de tampón de fosfato de potasio) a pH 7 y temperatura ambiente. Todos los potenciales medidos en este estudio fueron referidos a electrodo Ag / AgCl, y se prepararon soluciones homogéneas utilizando un baño sonicador (Elma S30H). Se utilizó un microscopio electrónico de barrido (SEM, LEO 1450VP) para determinar el tamaño y la distribución de las esferas de látex.
Métodos
Síntesis de nanopartículas de oro coloidal
Las AuNP coloidales se prepararon mediante reducción con citrato de sodio siguiendo el método de Turkevich [31]. Brevemente, aproximadamente 10 ml de 5 mM de HAuCl 4 · 3H 2 Se disolvió O en 180 ml de agua desionizada y se hirvió en condiciones de agitación continua en un dispositivo agitador magnético combinado de placa caliente. Diez mililitros de 0,5% ( w / v ) de citrato trisódico se añadió a la solución hirviendo y se observó que el color de la solución cambiaba gradualmente de rojo pálido a rojo rubí.
Preparación de partículas de látex
Las partículas de látex se prepararon mediante una reacción de copolimerización en emulsión sin jabón como describen Polpanich et al. [23] con algunas modificaciones. En resumen, se purgaron aproximadamente 190 g de agua desionizada con nitrógeno gaseoso en un matraz de tres bocas sumergido en un baño de agua durante aproximadamente 1 h con agitación a 350 rpm. Luego se añadieron veinte gramos de St y 0,5 g de AA y la temperatura se mantuvo a 70ºC. Después de eso, se añadieron aproximadamente 0,2 g de APS en 10 ml de agua desionizada seguido de vertido en la formulación en el matraz de tres bocas para iniciar la reacción de polimerización por radicales, y el proceso de polimerización se prosiguió durante 8 h. Las esferas de látex de carboxilo sintetizadas se recolectaron por centrifugación con agua desionizada dos veces a 13.000 rpm durante 20 min [23, 27, 28] y se resuspendieron en agua desionizada a temperatura ambiente (25 ° C) hasta su uso. La morfología y el tamaño medio de las partículas de látex de PSA se determinaron mediante microscopía electrónica de barrido (SEM).
Modificación de la superficie SPE y la inmovilización de la sonda de ADN
Antes de la modificación de la superficie, el SPE de carbono se enjuagó a fondo con agua desionizada y luego se recubrió con la suspensión de PSA a 3 mg / ml y se dejó secar al aire en condiciones ambientales, seguido de un vertido con 5 mg / ml de AuNP coloidales. . Las características electroquímicas del carbono SPE antes y después de la modificación con partículas de látex y AuNP se examinaron con el método CV. El SPE de carbono modificado con látex-AuNPs (PSA-AuNPs-SPE) se enjuagó luego con agua desionizada y se sumergió en 0,1 M de tampón de fosfato de potasio (pH 5) que contenía reactivos de reticulación de carbodiimida, es decir, 0,002 M de EDC y 0,005 M de NHS durante 2 h [32] antes de remojar durante la noche en 0,05 M de tampón de fosfato de potasio (pH 7) que contiene 5 µM de sonda de captura. Después de eso, el PSA-AuNPs-SPE (ADN-PSA-AuNPs-SPE) modificado con ADN se lavó a fondo con tampón de fosfato de potasio (0,05 M, pH 7) para eliminar la sonda adsorbida físicamente. El electrodo de ADN se sumergió en 0,05 M de tampón de fosfato de sodio a pH 7 que contenía ADN diana lineal (1 μM) y etiqueta de hibridación de ADN AQMS (5 mM) para hibridación parcial durante 1 hy luego se sumergió en 0,05 M de tampón de fosfato de sodio (pH 7) acondicionada con 1 µM de sonda indicadora y 5 mM de AQMS durante otra 1 h para el proceso de hibridación completo del ADN. Finalmente, el electrodo de ADN se enjuagó con tampón de fosfato de potasio (0,05 M, pH 7) para la eliminación de secuencias de oligonucleótidos no hibridadas. La respuesta electroquímica de cada sustancia extendida adherida al SPE se investigó con el método DPV. La Figura 1 representa el procedimiento paso a paso para el desarrollo de V. cholerae Biosensor de ADN basado en soporte sólido coloidal de PSA-AuNP.
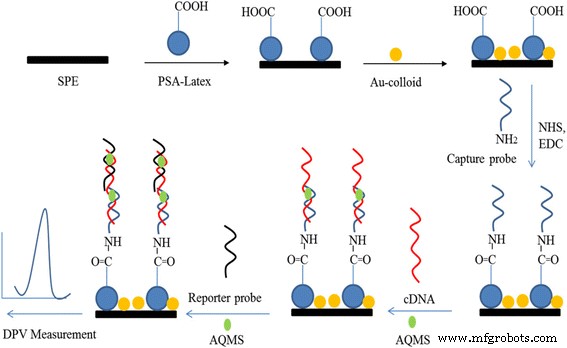
Representación esquemática del procedimiento de fabricación paso a paso del biosensor de ADN
Optimización de la hibridación de oligonucleótidos sintéticos
El efecto de varios parámetros como la sonda de ADN y las concentraciones de AQMS, el pH, la fuerza iónica y la capacidad tampón sobre la respuesta de hibridación de la sonda de ADN inmovilizada con la sonda de señal y el ADN diana se ha examinado antes de la determinación del rango lineal dinámico del ADN. biosensor. Además, la inmovilización de la sonda de captura y la duración de la hibridación del ADN, así como la vida útil y la regeneración del biosensor, también se evaluaron antes de la V desarrollada. cholerae El biosensor de ADN estaba listo para la aplicación en el experimento de recuperación y aumento. Las cargas de la sonda de captura y AQMS se optimizaron variando sus concentraciones de 1 a 6 μM y de 0,1 a 5 mM, respectivamente, mientras que la concentración de ADN diana y la sonda indicadora se mantuvo a 5 μM en 0,05 M de tampón de fosfato de sodio (pH 7,0). Los estudios del efecto del pH y de la concentración del tampón se realizaron cambiando el pH y la concentración del tampón de fosfato de sodio de un pH de 5,5 a 8,0 y de 0,001 a 1,000 M, respectivamente. La presencia de diferentes cationes en la respuesta de hibridación del ADN del biosensor electroquímico de ADN se realizó agregando Na + , K + , Ca 2+ y Fe 3+ iones a 1,0 M en el tampón de hibridación de ADN que contiene 1 mM de AQMS y 5 μM de ADNc y sonda de detección de pH 7,0. El efecto de la fuerza iónica se examinó variando la concentración de NaCl en el rango de 0,1 a 3,0 M a un pH de 7,0. Luego se determinó el rango dinámico del biosensor de ADN en 1.0 × 10 −21 a 1.0 × 10 −8 M V. cholerae ADNc utilizando una concentración de sonda de señal constante a 5 μM y pH 7,0. La duración de la inmovilización de la sonda de ADN se determinó sumergiendo el electrodo de ADN en 5 μM de solución de sonda de captura (pH 7,0) entre 1 y 13 h, y la respuesta de DPV se tomó cada 1-2 h. Mientras tanto, el tiempo de hibridación del ADN se determinó permitiendo que la reacción de hibridación del ADN ocurriera entre 15 y 180 minutos, y la respuesta del biosensor de ADN se registró cada 15-30 minutos. La vida útil del biosensor de ADN se determinó midiendo periódicamente la respuesta del biosensor de ADN hacia la detección de 5 μM de V. cholerae ADNc durante 120 días. El análisis se realizó en cinco repeticiones utilizando un nuevo electrodo de ADN para cada ensayo de hibridación en sándwich. La regeneración del electrodo de ADN se realizó usando una solución 0.1 M de NaOH durante 4 min, y la rehibridación del biosensor de ADN (60 min) se logró usando una solución de rehibridación que contenía 5 μM de ADNc y sonda de detección y 1 mM de AQMS a una fuerza iónica de 2.0 M en 0,05 M de tampón de fosfato de potasio (pH 7,0). El experimento de regeneración se realizó en seis repeticiones.
V. cholerae Cuantificación utilizando un biosensor electroquímico de ADN basado en PSA-AuNP
Varios V. cholerae cepas bacterianas, a saber, J2126-I, J2126-II, J3324-I, J3324-II, J3330-I, J3330-II, CDHI 5294-II y UVC1324, incluidas Citrobacter freundii (CF-I) y Citrobacter freundii (CF-II) se obtuvieron del Laboratorio de Microbiología, Facultad de Ciencias Aplicadas, Universidad AIMST, Kedah. A continuación, se realizó la extracción de ADN genómico sobre estas bacterias utilizando QIAGEN Genomic-tip 500 / G de acuerdo con el protocolo del fabricante. A continuación, el ADN extraído se diluyó 100 veces usando tampón de fosfato de sodio (0,05 M, pH 7,0). Aproximadamente 300 ml del ADN extraído que contenía 2,0 M de NaCl y 1 mM de AQMS se sometieron a ultrasonidos durante 15 min para liberar las roturas del ADN. Luego, la sonda de ADN inmovilizada se remojó durante 1 h para permitir que tuviera lugar el proceso de hibridación del ADN y se lavó cuidadosamente con tampón de fosfato de potasio 0,05 M (pH 7,0) para eliminar el ADN no unido. Se midió la evaluación de la respuesta del biosensor de ADN basada en la corriente máxima de DPV y se comparó con la respuesta de corriente generada por el electrodo de ADN sin reacción con el ADNc como señal de control. Cada experimento se llevó a cabo por triplicado en las mismas condiciones experimentales. t común La prueba se utilizó para determinar una diferencia significativa entre la respuesta del biosensor de ADN y la respuesta del control. La recuperación de V. cholerae J3324 y V. cholerae ADN de UVC1324 a 1.0 × 10 −4 μg μL −1 , 1.0 × 10 −5 μg μL −1 y 1.0 × 10 −6 μg μL −1 añadido al tampón de hibridación se llevó a cabo utilizando el biosensor electroquímico de ADN propuesto basado en PSA-AuNP.
Resultados y discusión
Morfología de las partículas de látex y comportamiento electroquímico de la SPE modificada con nanopartículas de látex y oro.
La Figura 2 muestra la micrografía SEM de las esferas de látex carboxiladas con un tamaño medio de partícula de 186,1 ± 4,6 nm. La distribución uniforme del tamaño de las esferas de PSA permitió la inmovilización homogénea de las moléculas de ADN en la superficie del látex para mejorar la respuesta de reproducibilidad del biosensor de ADN. Se utilizó un microscopio electrónico de barrido (SEM, LEO 1450VP) para determinar el tamaño y la distribución de las esferas de látex.

Micrografía SEM de las esferas de látex de PSA sintetizadas a 10.000 aumentos
Los resultados electrodinámicos del SPE modificado se tabulan en la Tabla 2. La separación de potencial pico (ΔEp) indica la cinética de transferencia de electrones del sistema. El SPE modificado con PSA (PSA-SPE) mostró el valor ΔEp más alto debido al lento proceso de transferencia de carga en la capa de partículas de copolímero coloidal, lo que hizo que el sistema se moviera hacia un estado cuasi-reversible. Sin embargo, cuando los AuNP se cargaron en el PSA-SPE, la disminución de ΔEp implica una mejora en la tasa de transferencia de electrones en la superficie del electrodo.
La Figura 3 muestra los voltamogramas de pulso diferencial de la respuesta de oxidación de AQMS en el SPE modificado con látex y la respuesta de hibridación secuencial de V. cholerae Biosensor de ADN. Se observó una diferencia significativa de corriente máxima de DPV entre electrodos que contenían solo microesferas modificadas con látex y sin ADN de captura inmovilizado (experimentos (a) y (b)) y modificados con sondas de ADN de captura inmovilizadas en presencia de ADNc y sonda informada (experimento (g )). Esto indica que las sondas de captura de ADN aminado se inmovilizaron con éxito sobre las esferas de látex de PSA carboxilado recubiertas mediante el protocolo de acoplamiento EDC / NHS [33]. El experimento (g) también muestra una respuesta de DPV mucho más alta en comparación con los electrodos de ADN en presencia de ncDNA y sonda informadora informada (experimento (d)), en ADN no coincidente y sonda informadora informada (experimento (e)), y ADNc diana con ninguna sonda informada (experimento (f)). Esto se debe a la hibridación completa del ADN diana con las sondas de captura y reportero a través de la reacción de hibridación en sándwich en la superficie del biosensor de ADN como se demuestra en el experimento (g). Esto también muestra que el uso de la sonda informada podría mejorar la señal de la hibridación del ADN. Sin embargo, la corriente de DPV resultante de la hibridación observada en presencia de ADN diana sin la incorporación de una sonda informada (experimento (f)) es aún más alta que las señales de corriente de DPV observadas para ADN no hibridado (experimentos (c), (d) y (e)).
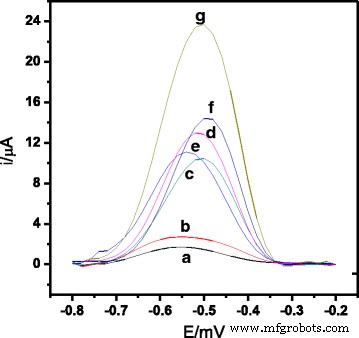
Señal de voltamogramas de pulso diferencial de AQMS de los electrodos ( a ) PSA-SPE, ( b ) PSA-AuNPs-SPE, ( c ) capturan probe-PSA-AuNPs-SPE y cuando están en presencia de ( d ) ncDNA y sonda informadora, ( e ) ADN discordante y sonda informadora, ( f ) ADNc solo y ( g ) ADNc y sonda informadora
Efecto de la carga de la sonda de ADN y la concentración de AQMS
El efecto de la concentración de la sonda de ADN sobre la respuesta de hibridación del ADN se observó a través de la respuesta de oxidación electroquímica de AQMS. La Figura 4a muestra que la respuesta del biosensor de ADN aumentó gradualmente con el aumento de la cantidad de sonda de ADN inmovilizada en el PSA-AuNPs-SPE de 1 a 4 µM. Esto se atribuyó a la creciente cantidad de AQMS electroactivo intercalado en el ADN bicatenario (dsDNA) para producir una transferencia de electrones a través de la hélice de ADN inmovilizada. Se observó que la respuesta DPV del biosensor de ADN se estabilizaba entre 4 y 6 μM de sonda de ADN, lo que indica que se logró una carga de sonda de ADN óptima en la superficie del electrodo [34]. Por lo tanto, se seleccionó una sonda de captura de 4 µM como carga de sonda de ADN óptima en los experimentos posteriores. La concentración de la etiqueta AQMS también se ha optimizado en el electrolito de medición entre 0,1 y 5,0 mM, y se encontró que la concentración de AQMS a 1 mM era suficiente para una reacción de intercalación de ADN óptima (Fig. 4b).
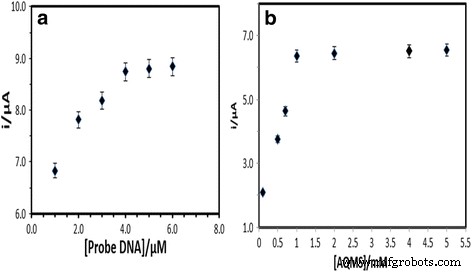
El efecto de la sonda de captura ( a ) y concentraciones de AQMS ( b ) sobre la respuesta del biosensor de ADN realizada con ADNc 5 μM y sonda de señal en tampón de fosfato de sodio 0,05 M (pH 7,0)
Efecto del pH, la fuerza iónica y la capacidad tampón
La velocidad de la reacción de hibridación del ADN depende en gran medida del pH de la solución. Como puede verse en la Fig. 5a, en un entorno más ácido, la protonación de la cadena principal de fosfodieter del ADN redujo la solubilidad de la molécula de ADN, lo que finalmente disminuyó la velocidad de la reacción de hibridación del ADN. Mientras que en medio básico, rompió los enlaces de hidrógeno débiles que mantienen unidos los pares de bases de ADN. La reacción de hibridación de ADN óptima fue más favorable en condiciones neutrales, por lo que promovió que más sondas de captura se hibridaran con el ADN diana y posteriormente permitió la intercalación de las sondas redox AQMS para hacer que el reconocimiento de la hibridación del ADN se hiciera realidad. Por tanto, se utilizó 0,05 M de tampón de fosfato de sodio a pH 7,0 como medio de hibridación de ADN para estudios posteriores de biosensores de ADN. Iones cargados positivamente como Ca 2+ , Na + , K + y Fe 3+ Los iones pueden interactuar con la cadena fosfodiéster cargada negativamente del ADN. Esta reacción iónica neutralizará la carga de la molécula de ADN, disminuyendo así las repulsiones estéricas entre las moléculas de ADN para facilitar la reacción de hibridación del ADN. La Figura 5b representa el efecto de algunos cationes sobre la respuesta de hibridación del ADN. Se observó que la respuesta de hibridación del ADN aumentaba en presencia de iones cargados positivamente en el orden de Na + > K + > Fe 3+ > Ca 2+ . Ambos Ca 2+ y Fe 3+ Se encontró que los iones reducen considerablemente la respuesta de hibridación del ADN debido a las interacciones iónicas de Ca 2+ y Fe 3+ iones con iones de fosfato de la solución tampón, lo que condujo a la formación de compuestos de fosfato insolubles. Esto ha reducido el contenido iónico del medio, aumentando así la repulsión electrostática entre las moléculas de ADN. La corriente de hibridación de ADN más alta se obtuvo en presencia de Na + ion debido a su tamaño más pequeño y mayor afinidad hacia la cadena principal de ADN azúcar-fosfato en comparación con K + ion para superar el impedimento estérico y la repulsión electrostática entre los grupos fosfato cargados negativamente de los ADN.
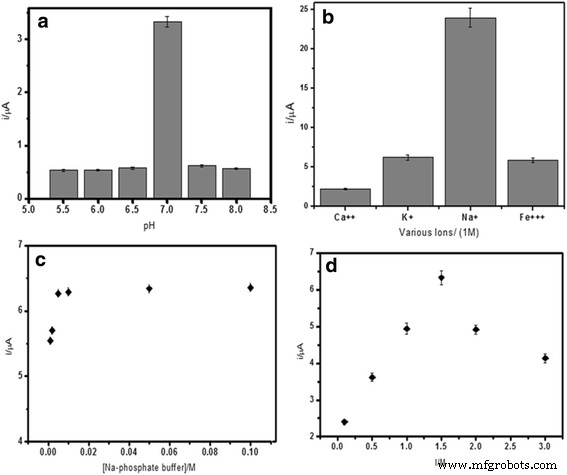
El efecto del pH ( a ) varios cationes ( b ), concentración de tampón ( c ) y fuerza iónica ( d ) sobre la respuesta de hibridación del ADN de la V. cholerae Biosensor de ADN. La hibridación se realizó con ADNc 5 μM y sonda indicadora seguida de intercalación con AQMS 1 mM
Además, la fuerza iónica de la solución también afectaría la respuesta del biosensor de ADN. La Figura 5c, d muestra la capacidad de tampón óptima, y la fuerza iónica se logró utilizando 0,05 M de tampón de fosfato de sodio con el pH fijado a pH 7,0 y 2,0 M de NaCl, respectivamente. En esta condición, favoreció al máximo la reacción de hibridación del ADN; por tanto, se obtuvo una alta respuesta de DPV. Con una capacidad tampón y una fuerza iónica óptimas de la solución, la repulsión electrostática entre las moléculas de ADN disminuyó y, por lo tanto, se mejoró la reacción de hibridación del ADN. Por el contrario, cuando se utilizó un contenido iónico demasiado bajo o demasiado alto, el impedimento estérico y la repulsión electrostática se volvieron dominantes y restringieron la hibridación de las moléculas de ADN.
Establecimiento de V. cholerae Curva de calibración del biosensor de ADN
A partir del resultado que se muestra en la Fig. 6a, la respuesta del biosensor de ADN aumentó proporcionalmente con el aumento de la concentración de ADNc de 1.0 × 10 -21 a 1.0 × 10 −8 M ( R 2 =0,99) con un límite de detección de 1,0 × 10 −21 M. El límite de detección se calculó sobre la base de tres veces la desviación estándar de la respuesta del biosensor en la curva de respuesta que se aproxima al límite de detección dividido por la pendiente de calibración lineal. El amplio rango de detección lineal del biosensor de ADN se debió a las partículas de látex de PSA esféricas y altamente monodispersas en un rango de tamaño submicrónico utilizadas como matriz portadora para la inmovilización del ADN. La capa rica en ácido acrílico en la superficie de las partículas de látex ofreció un gran sitio de unión para la unión de las sondas de captura de ADN para crear una superficie cubierta máxima por la capa receptora de ADN. Además, la incorporación de AuNP en el SPE modificado con PSA amplificó aún más la señal analítica de la respuesta de hibridación del ADN, y esto hizo que el biosensor de ADN tuviera una alta sensibilidad (Fig. 6b).
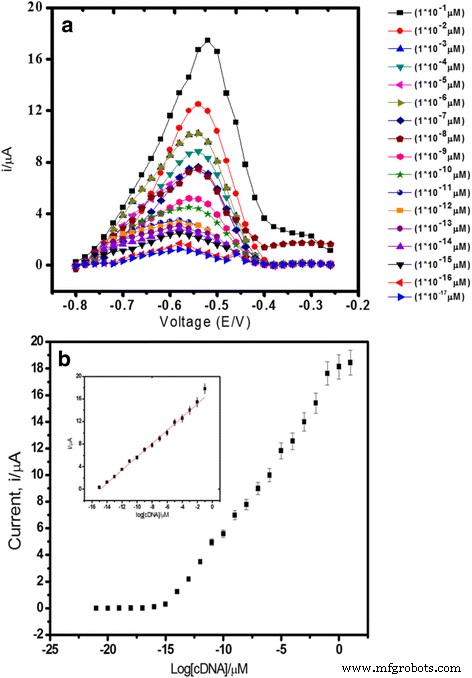
Differential pulse voltammograms (a ) and DNA biosensor linear range (b ) obtained using various cDNA concentrations from 1.0 × 10 −15 to 1.0 × 10 −1 μM V. cholerae target DNA and 5 μM signal probe
DNA Probe Immobilisation and Hybridization Times
It took about 8 h for the capture probe to be immobilised on the PSA copolymer particles surface, as illustrated by the DNA biosensor response in Fig. 7a, which showed a current increment from 1.0 to 8.0 h of capture probe immobilisation time, after which no obvious change in the DPV current was observed. Longer immobilisation time resulted in a higher amount of DNA probes immobilised onto the latex. After 8.0 h of exposure to the DNA probes, the hydrophilic functional latex with reactive carboxyl groups at the surface was presumably fully attached with the DNA probes. DNA hybridisation time, on the other hand, is the rate limiting step, which determines the response time of the DNA biosensor. Based on the DNA biosensor response trend in Fig. 7b, the response time of the V. cholerae DNA biosensor developed in this study was estimated to be about 60 min for the dual hybridisation processes to complete.
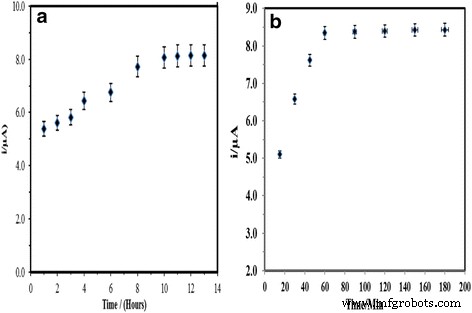
DNA probe immobilisation duration on the immobilised PSA latex colloidal particles (a ) and DNA hybridization duration of the DNA biosensor (b ) in 0.05 M potassium phosphate buffer at pH 7.0 containing 5 μM target DNA and reporter probe and 1 mM AQMS at 2.0 M ionic strength
Long-Term Stability and Regeneration of V.cholerae DNA Biosensor
Figure 8 shows the shelf life of the V. cholerae DNA biosensor. The DNA biosensor showed the highest response to the detection of 5 μM of V. cholerae cDNA for the first month of the experimental period. The electrochemical DNA biosensor was able to retain 95% of its initial DPV current after 58 days of storage period. The DNA hybridisation response was then gradually decreased to about 75% of its original response on the 75th day and exhibited 40% of its initial performance on the 100th operational day. The bioactivity of the immobilised capture probe was finally declined to 30% after 3 months of storage period. The reproducibility of each calibration point, which was repeated on five replicate DNA electrodes, gave satisfactory relative standard deviation (RSD) between 2.4 and 4.5% (n =5).
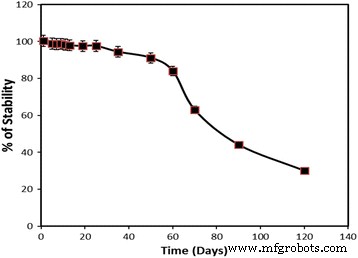
The life span profile of the fabricated V. cholerae DNA sensing electrode. The electrode was stored in 0.05 M potassium phosphate buffer (pH 7.0) at 4 °C after every DPV measurement was taken
Regeneration of biosensor indicates whether the biosensor is reusable for a series of consecutive analyses. The regeneration method used in this study was conducted based on previously reported protocol in other studies [11, 21] with slight modifications. In this study, 0.1 M of NaOH solution was used as the regeneration solution to break the hydrogen bonds between base pairs of hybridised dsDNA. With the result from Fig. 9, it is notable that the DNA biosensor response declined significantly after incubation in 0.1 M of NaOH and the percentage of the DNA biosensor response reduced from 35.1 to 5.2% relative to the DNA biosensor initial response after incubation in 0.1 M of NaOH solution from 30 to 240 s. The DNA biosensor response decreased with the increasing incubation time signifies the hydrogen bonds between hybridised dsDNA were broken up by the alkaline regeneration solution. However, rehybridisation of the DNA biosensor was able to attain almost 100% of its initial response for a consecutive six DNA analyses with a reversibility RSD of 5%.
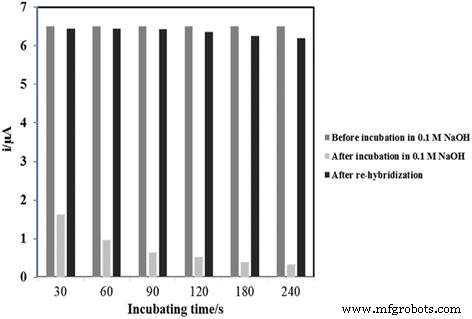
Repeatability of V. cholerae DNA biosensor using 0.1 M NaOH regeneration solution and rehybridization solution containing 5 μM cDNA and detection probe and 1 mM AQMS at 2.0 M ionic strength in 0.05 M potassium phosphate buffer (pH 7.0)
Determination of V. cholerae Bacteria with the Developed DNA Biosensor
The optimised DNA biosensor has been applied to quantify the V. cholerae DNA extracted from various V. cholerae bacterial strains. Table 3 presents the results acquired from DNA tests carried out with the hybridisation medium spiked with different strains of V. cholerae DNAs and other bacterial species at a concentration within the calibration range of the DNA biosensor. The DNA biosensor showed superior selectivity towards V. cholerae J3324–I, V. cholerae J3324–II, and V. cholerae UVC1324 with high DPV current response and low current signals were obtained for the evaluation of both Citrobacter freundii (CF-I) and Citrobacter freundii (CF-II).
Recovery of V. cholerae J3324 and V. cholerae UVC1324 DNAs at three different concentrations spiked into the hybridisation buffer demonstrated 91.4 ± 2.2% to 108.9 ± 4.8% (n =3) of recoveries percentage (Table 4). This result suggests that the proposed PSA-AuNPs-based electrochemical DNA biosensor could be adopted for highly reliable and accurate detection of V. cholerae DNA in environmental and clinical samples.
Performance Comparison with Other Reported V. cholerae DNA Biosensors
Based on the data summarised in Table 5, the proposed electrochemical DNA biosensor based on PSA-AuNPs immobilisation material shows an exceptional broad linear quantification range compared to other planar two-dimensional electrodes as the DNA supporters. This clearly demonstrates the advantage of the micro-sized latex particles where the polymeric PSA is capable to intensify the probe binding capacity with a simple loading method via the classical EDC/NHS coupling compared to avidin-biotin technology [24, 26] and ultra-low detection limit in zeptomolar range with reasonable assay time.
Conclusions
This study reports the development of an electrochemical DNA biosensor for the detection of one of the most devastating high-risk V. cholerae pathogens. The PSA-AuNPs-modified DNA biosensor can be used for direct detection of DNA of interest from the extracted DNA without the need of amplification reaction via conventional PCR method, which is commonly used in those previously reported V. cholerae DNA biosensors. In addition, no further dilution of the extracted DNA is needed as the high-capacity AuNPs-doped latex microspheres-based DNA biosensor is highly sensitive for the quantitation of DNA at extremely low level in sub zeptomolar range. Therefore, the electrochemical DNA biosensor is greatly suitable as a surveillance and diagnostic tool to control the epidemic of the fatal intestinal infection.
Nanomateriales
- El potencial para integrar datos visuales con IoT
- Demostración de un biosensor flexible basado en grafeno para la detección rápida y sensible de células de cáncer de ovario
- La detección fotoelectroquímica mejorada de ácido úrico en electrodo de carbono vítreo modificado con nanopartículas de Au
- Un biosensor de ADN electroquímico altamente sensible de nanocompuesto acrílico-oro para la determinación del género del pez Arowana
- Nanoesferas de carbono monodispersas con estructura porosa jerárquica como material de electrodo para supercondensador
- Estabilidad mejorada de nanopartículas magnéticas de oro con poli (ácido 4-estirenosulfónico-ácido co-maleico):propiedades ópticas personalizadas para la detección de proteínas
- Síntesis controlada de BaYF5:Er3 +, Yb3 + con morfología diferente para la mejora de la luminiscencia de conversión ascendente
- Aptasensor fluorescente a base de óxido de grafeno para la detección de encendido de CCRF-CEM
- Compuesto de nano fibras de TPU conductor de electricidad con alta capacidad de estiramiento para sensor de deformación flexible
- La fabricación de nanomambas de grafeno uniformes de gran superficie para la detección directa de terahercios a temperatura ambiente y alta velocidad
- Impossible Objects se asocia con BASF para la impresión 3D compuesta



