Detección de microARN-335-5p en una superficie de electrodo interdigitado para determinar la gravedad de los aneurismas de la aorta abdominal
Resumen
El aneurisma de la aorta abdominal (AAA) se refiere al agrandamiento de la arteria inferior de la aorta abdominal, y se necesita con urgencia la identificación de una herramienta de detección temprana para el diagnóstico. En el estudio actual, se utilizó una superficie de detección de electrodo interdigitado (IDE) para identificar el miARN-335-5p, que refleja la formación de AAA. La uniformidad del material de sílice se observó mediante perfilometría 3D, y la superficie altamente conductora modificada químicamente mejoró la detección a través del modo I-V. El miARN-335-5p dirigido se detectó de una manera dependiente de la dosis y, basándose en regresión lineal y análisis 3σ, se determinó que la sensibilidad era 1 fM con una sonda biotinilada. La alta especificidad se demostró mediante la discriminación de la secuencia diana de las secuencias no complementarias y con desajustes simples y triples. Estos resultados demostraron la detección de alto rendimiento de miARN-335-5p con buena reproducibilidad para determinar la gravedad de AAA.
Introducción
Los aneurismas de la aorta abdominal (AAA) son enfermedades potencialmente mortales que se definen como una aorta abdominal con un diámetro> 3 cm o más grande de lo normal [1, 2]. Esta condición ocurre con aterosclerosis o acumulación de placa, que debilita las paredes de la aorta abdominal y da como resultado una protuberancia hacia afuera, similar a un globo. Con el tiempo, la pared de la arteria se ensancha y esta situación es comparable al envejecimiento de las mangueras de jardín. La presión del bombeo de sangre a través de la aorta hace que esta área debilitada se abulte hacia afuera, lo que se denomina aneurisma. El AAA se forma cuando la porción debilitada de la aorta da lugar a complicaciones [3, 4, 5, 6]. El AAA puede provocar la muerte por rotura de pequeños aneurismas. En la actualidad, para diagnosticar esta afección se utilizan exploraciones físicas, angiografías por tomografía axial computarizada, resonancia magnética y ecografía [7,8,9,10]. Sin embargo, no existen métodos de detección de AAA, que se identifican comúnmente al analizar otros problemas de salud. Esta situación da como resultado un retraso en la identificación de AAA, lo que en última instancia provoca problemas de salud innecesarios. Para superar este problema, los investigadores deben desarrollar métodos de detección temprana y una estrategia potencial es el desarrollo de un sistema de detección.
La detección temprana, rápida y sensible de enfermedades de manera cuantitativa es un objetivo vital para los diagnósticos clínicos. Las actuales plataformas de biosensores han cumplido varias demandas y requieren un entorno de laboratorio y una formación adecuados. Por lo tanto, la mayoría de los métodos no son portátiles, lo que es necesario para la detección ideal en el punto de atención [11, 12]. Además, para ayudar a los médicos a tomar decisiones de manera precisa y rápida, es muy deseable un análisis de los cambios en los niveles de biomarcadores. Los biomarcadores circulantes que se expresan en áreas específicas deben investigarse más a fondo para diagnosticar AAA y seguir el progreso del tratamiento. La identificación de estos tipos de biomarcadores circulantes ayudará a diagnosticar la enfermedad y realizar un seguimiento del paciente después del tratamiento. Para cubrir estas necesidades, este estudio propone generar sensores de biomarcadores apropiados para AAA. El sensor (electrodo interdigitado) propuesto en este estudio tiene potencial para análisis de alto rendimiento con una amplia gama de biomarcadores. Es un sistema de dielectrodos con espacios y dedos alternos que operan bajo medidas dieléctricas [13,14,15].
Los biomarcadores pueden ser cualquier biomolécula, que incluye ADN, ARN, proteínas, carbohidratos, lípidos y sus formas modificadas [16, 17]. Además, los investigadores han propuesto que diferentes biomarcadores, como los ARN no codificantes, se expresan en el sistema celular, pero no se traducirán en proteínas [18]. Los ARN no codificantes generalmente no se traducen en proteínas y generalmente tienen secuencias cortas [18,19,20,21]. Se han informado diferentes clases de ARN no codificantes, como microARN (miARN), ARN ribosómico, ARN de transferencia, ARN nucleolar pequeño, ARN nuclear pequeño, ARN de telomerasa, ARNsn, ARN Xist, ARN de bóveda y ARN 7SL. Los miARN funcionan principalmente en la regulación transcripcional y postranscripcional de la expresión génica y, a menudo, dan lugar al silenciamiento génico [22]. Recientemente, los investigadores describieron la importancia de los miARN para la predicción de AAA y notificaron una reducción en la expresión de miARN-335-5p en pacientes con AAA [23]. Se ha demostrado que la combinación de factores clínicos y la expresión de microARN mejoraron drásticamente la predicción de enfermedades y mostraron una mayor precisión [24]. Los investigadores se han centrado específicamente en miRNA-335-5p, que mostró un rango significativamente mínimo en individuos con AAA de rápido crecimiento [2, 23]. Además, una disminución en los niveles de miARN-335-5p mejoró la confianza en la detección de AAA en crecimiento. En otras palabras, la salida negativa (niveles más altos) de miR-335-5p indica la gravedad del AAA y minimiza la laboriosa detección. Este hallazgo fue demostrado por Wanhainen et al. [23] y reveló que los miARN son biomarcadores útiles para detectar AAA y eliminar el riesgo de AAA de rápido crecimiento. El estudio actual demuestra la aplicación de la detección de miARN-335-5p mediante un sensor de electrodo interdigitado (IDE) para determinar la gravedad del AAA en las personas afectadas.
Materiales y métodos
Reactivos y biomoléculas
La estreptavidina, el 1,1'-carbonildiimidazol (CDI) y la solución tampón de fosfato (PBS) se adquirieron en Sigma-Aldrich, EE. UU. La etanolamina se adquirió en Fisher Scientific, Reino Unido. Todos los oligos se sintetizaron comercialmente de Apical Scientific Sdn. Bhd., Malasia.
Diseño de patrones en una máscara cromada
Inicialmente, el patrón del sensor dieléctrico se diseñó utilizando el software AutoCAD. Las dimensiones deseadas eran una longitud de 7500 μm, un ancho de 4100 μm, 20 pares de huecos de dedos, un tamaño de hueco de 85 μm, un tamaño de electrodo de 100 μm, un espesor de electrodo de 40 nm y una longitud de dedo de 4000 μm. El patrón se imprimió en una fotomáscara en blanco y se pegó en la superficie del vidrio cromado. Este vidrio cromado se fijó bajo un sistema de exposición UV para el procedimiento de transferencia del patrón. Se colocó un sustrato de dióxido de silicio depositado con una película delgada de aluminio en la dirección opuesta contra el vidrio cromado y se expuso a la luz ultravioleta durante 10 s. El patrón fue transferido seguido por el proceso de revelado usando el revelador de resistencia.
Fabricación del electrodo interdigitado
El electrodo de superficie se produjo mediante un proceso de fabricación microelectrónica convencional para fabricar la superficie de la siguiente manera:(i) Se preparó una oblea de 4 pulgadas con óxido nativo existente. (ii) La oblea se limpió usando grabado de óxido tamponado (BOE) y una solución de piraña para eliminar el óxido nativo y los contaminantes inorgánicos. (iii) Se hizo crecer una capa de óxido de 2800 Å de espesor mediante una hora de oxidación térmica en húmedo a una temperatura de 1000 ° C. (iv) La oblea con la capa de óxido se dividió en cuatro para un mejor manejo. (v) La capa de metal se depositó con l como capa de adhesión para la capa conductora. La capa de aluminio (40 nm) se produjo a 3 cm de largo con un diámetro de 0,5 mm mediante un método de deposición física basado en evaporador térmico, (vi) Una capa de fotorresistente positivo fue una capa de rotación sobre el sustrato, (vii) La oblea fue horneado suave a 90 ° C durante 60 s. (viii) La oblea se alineó con una máscara de cromo y se expuso a luz ultravioleta durante 10 s. (ix) La oblea se enjuagó en solución reveladora RD6 en 15 s para eliminar el área no expuesta, lo que resultó en el patrón de electrodo interdigitado (IDE) con óxido. (x) La oblea se horneó a 90 ° C durante 120 s. (xi) La oblea se grabó con agua regia para eliminar el área expuesta de ambas capas. (xii) Se produjo el patrón de la superficie, y el fotorresistente restante se lavó usando acetona. Luego, se llevó a cabo un análisis de nanoprofilometría 3D (perfilometría 3D de Hawk, EE. UU.) Para observar la imagen nítida de los espacios entre los electrodos. Las mediciones de corriente (A) se realizaron utilizando Keithley 6487 con un barrido lineal de 0 a 2 V en un paso de 0,1 V (a 20 s).
Funcionalización química de superficie:estrategia de estreptavidina-biotina tetravalente
Para la funcionalización química de la superficie, la superficie de sílice se lavó inicialmente a fondo con hidróxido de potasio 1 M (pH 9,0) para activar la superficie. Esta superficie se modificó adicionalmente por CDI (0,5 M) con un período de incubación de 1 h para reaccionar con estreptavidina 100 nM (durante 1 h), diluida del stock original en solución salina tamponada con fosfato 10 mM (PBS; pH 7,4). Después de este paso, los espacios sin reaccionar se bloquearon usando etanolamina 1 M (durante 1 h). Luego, la estreptavidina tetravalente se hizo reaccionar con 1 µM de miARN-335-5p de sonda biotinilada (durante 10 min) a temperatura ambiente. Una vez completada cada modificación, la superficie se lavó minuciosamente con PBS a menos que se indique lo contrario.
Diseño de la sonda y las secuencias objetivo a partir de miRNA-335-5p
El miARN-335-5p de longitud completa (5′-UGUUUUGAGCGGGGGUCAAGAGCAAUAACGAAAAAUGUUUGUCAUAAACCGUUUUUCAUUAUUGCUCCUGACCUCCUCUCAUUUGCUAUAUUCA-3 ′) con el código de acceso MIMAT0000765, se ha recopilado del banco de datos. La región deseada para este estudio como objetivo es 5′-UCAAGAGCAAUAACGAAAAAUGU-3 ′. Para capturar la región de miARN-335-5p objetivo, la sonda se diseñó como 5'-ACAUUUUUCGUUAUUGCUCUUG-3 '. En el extremo 3 ', se marcó la biotina para interactuar con la estreptavidina inmovilizada en la superficie de detección. La estructura secundaria del miARN-335-5p de longitud completa fue predicha por el software mfold en línea (http://unafold.rna.albany.edu/?q=mfold).
Interacciones sonda-objetivo:secuencias de miRNA-335-5p
Tras la unión de la sonda biotinilada a la superficie de estreptavidina como se detalla anteriormente, la secuencia de miARN-335-5p diana interactuó (durante 10 min) a temperatura ambiente desde las concentraciones de femtomolar bajo a nanomolar bajo. Estas concentraciones variaron de 1 fM a 1 nM con diluciones seriadas de 10 veces en PBS. Los aditamentos se probaron independientemente de concentraciones más bajas a más altas, y tras la formación del dúplex, la superficie se lavó con volúmenes de 5 veces de PBS y se tomaron las medidas.
Mediciones I-V
Las caracterizaciones eléctricas se realizaron mediante mediciones I-V utilizando un Picoamperímetro Keithley 6487 con suministro de voltaje / corriente. Las medidas se registraron a los voltios antes mencionados en la superficie desnuda, seguidos de cada modificación química de la superficie. De manera similar, para el análisis interactivo entre la sonda y el miARN-335-5p diana en cada concentración, la medición se realizó después del paso de lavado con 10 volúmenes de reacción. Todas las modificaciones, interacciones y mediciones se mantuvieron en condiciones húmedas a menos que se indique lo contrario.
Resultados y discusión
Fabricación del sensor IDE y análisis de superficie
El estudio actual utilizó miRNA-335-5p como herramienta para dilucidar la gravedad de AAA mediante análisis interactivo en un sensor de electrodo interdigitado (IDE) (Fig. 1). Para el análisis interactivo con la sonda y el objetivo diseñado a partir de la longitud completa de miARN-335-5p, se diseñó y fabricó una superficie IDE utilizando técnicas fotolitográficas convencionales. Las modificaciones de la superficie paso a paso se llevaron a cabo y se midieron con un amperímetro (Fig. 2b, c). La alineación de la máscara diseñada para la fabricación de IDE y la uniformidad se confirmaron mediante análisis de nanoprofilometría 3D de alta resolución. Esta observación confirmó que la fabricación y la distancia entre cada dedo estaban bien alineadas (Fig. 2d). Los IDE tienen un tamaño de electrodo de ~ 100 μm con un espaciado de ~ 85 μm, y se forma una gran red intercalada con millones de biomarcadores en serie / paralelo. Para asegurar el éxito de los IDE fabricados, utilizamos diferentes dispositivos desnudos para determinar la reproducibilidad bajo el sistema dieléctrico. Este sistema mide las vibraciones moleculares entre los dielectrodos causadas por el momento dipolar (Fig. 2c). Este IDE dieléctrico es un sistema bien aceptado que se ha utilizado ampliamente para diferentes análisis biomoleculares y químicos [15, 25, 26, 27, 28]. En este estudio, todas las modificaciones químicas de la superficie y los análisis interactivos se midieron mediante el modo de corriente I-V.
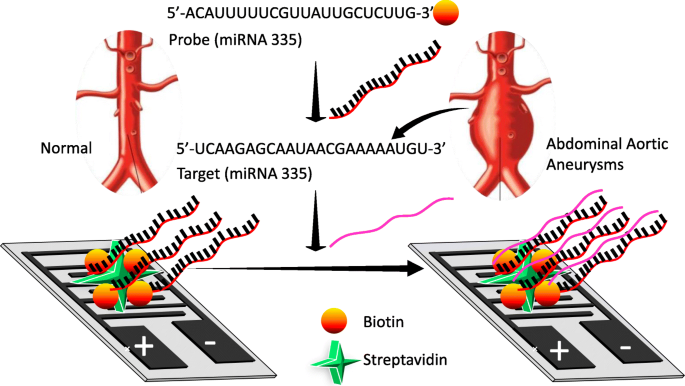
Representación de la detección de miRNA-335-5p en un IDE. La sonda y el objetivo diseñados se muestran junto con la formación de dúplex en la superficie IDE
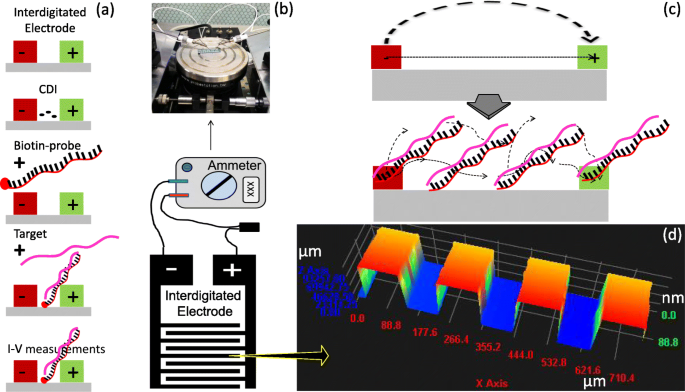
Medidas de superficie en un IDE. un Pasos de medición. b Modo de medición. Se muestran el patrón IDE y el sistema de conexión. También se muestra una medición dieléctrica. c Mecanismo de superficie durante la medición. Se muestra un momento dipolar. d El perfil de perfilometría 3D del IDE fabricado
Ensamblaje molecular de superficie de alta densidad:estrategia de estreptavidina-biotina
Se ha demostrado que las uniones moleculares de alta densidad en la superficie de detección o los biochips son cruciales y dependen en gran medida de la funcionalización de la superficie [29, 30]. Este estudio tuvo como objetivo determinar la superficie adecuada necesaria para un gran número de adaptaciones moleculares, que es obligatorio para la expresión, el descubrimiento y el análisis molecular de genes. Para estas posibles aplicaciones, la generación de superficies de sistemas analíticos requiere el acoplamiento adecuado de moléculas para inmovilizarlas en formatos adecuados. Estas superficies de material se estudiaron para investigar el mecanismo de interacción de la superficie, ya que son baratas y proporcionan diversos grados de densidades de empaquetamiento, morfología y espesor. Este hallazgo facilitará la correcta unión de moléculas y evitará la pérdida de actividad biomolecular. Además, la orientación correcta de la inmovilización biomolecular se conserva con la confirmación estructural secundaria adecuada.
Para lograr moléculas de alta densidad en la superficie, inicialmente lavamos a fondo la superficie de sílice con una solución alcalina y la modificamos con CDI. Para capturar altos niveles de sondas de biotinilación en la superficie de CDI, inmovilizamos la sonda con estreptavidina. La estreptavidina es una proteína tetravalente con cuatro sitios para unirse a la biotina y se considera una molécula de alta afinidad en las ciencias biológicas [31, 32]. La superficie de IDE tenía un nivel de corriente de 2.08E− 10 A, y después de la fijación de CDI, se incrementó a 9.2E− 06 A. Este hallazgo indica claramente que la superficie de IDE modificada por CDI es apropiada para nuestros análisis. Cuando se añadió estreptavidina, el nivel actual se incrementó aún más a 2,36E - 05 A. Con esta estrategia, inmovilizamos un mayor número de sondas biotiniladas diseñadas a partir de miRNA-335-5p de longitud completa en la superficie IDE (Fig. 3a). Las interacciones de biotina se llevaron a cabo después de la etapa de bloqueo en la superficie IDE para enmascarar las áreas que no reaccionaron. Con cada modificación de la superficie, se determinaron los cambios en la corriente (Fig. 3b). Como se muestra en la Fig.3c, después de la adición de etanolamina, los cambios de corriente fueron 2.53E - 05 A, y cuando se agregó 1 nM de secuencia de captura biotinilada, el nivel de la corriente se redujo a 1.29E - 08 A. Este cambio en la corriente confirmó la inmovilización de la sonda de captura en la superficie de detección del IDE. Generalmente, un oligonucleótido monocatenario lleva una carga más negativa con la cadena principal de fosfato 5-carbonatado expuesta (sonda), mientras que la carga neta de etanolamina es neutra, como lo indica el proveedor. Esta gran diferencia de carga resultó en una variación aparente en la corriente medida, como se muestra en la Fig. 3c. Además, este parámetro se revirtió de nuevo tras la formación de dúplex con la hebra diana. Este hallazgo se debe a la reducción en la exposición de la cadena principal de fosfato, y las cargas positivas adicionales se originan en las nucleobases en la cadena de miARN (Fig. 4a).
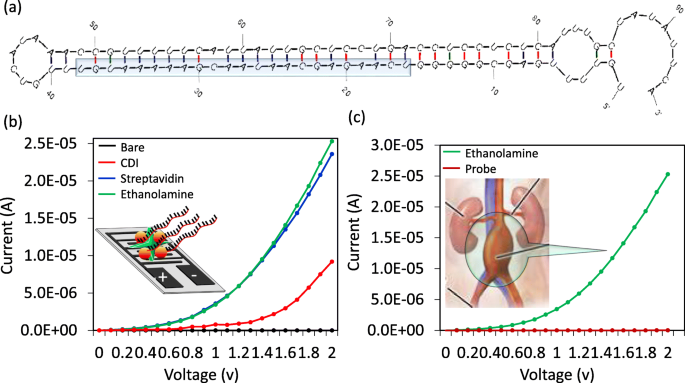
un Estructura secundaria predicha del miRNA-335-5p. El miARN-335-5p de longitud completa se muestra con las regiones de tallo y bucle. Se indica la región objetivo seleccionada para el análisis interactivo. b Mediciones I-V en el IDE para las modificaciones químicas y biológicas de la superficie (círculo negro, desnudo; círculo rojo, CDI; círculo azul oscuro, estreptavidina; círculo verde, etanolamina). La figura insertada se muestra esquemáticamente. c Mediciones de I-V en el IDE para el accesorio de la sonda. (círculo verde, etanolamina; círculo rojo oscuro, sonda). La imagen de la complicación AAA se muestra como el recuadro de la figura
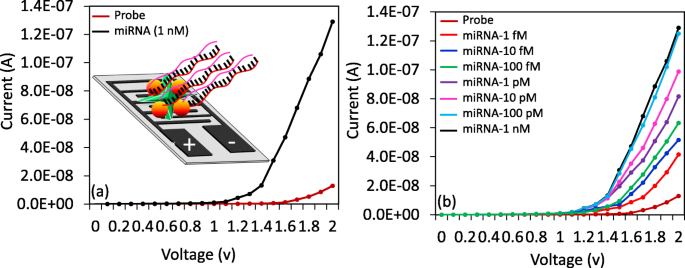
Análisis interactivo de la sonda y el objetivo. un Interacción de dosis más alta de la sonda y miARN-335-5p (diana). (círculo rojo oscuro, sonda; círculo negro, objetivo). b Análisis dependiente de la dosis de miARN-335-5p. Pruebas desde el femtomolar inferior al rango nanomolar inferior. (círculo rojo oscuro, sonda; círculo naranja, 1 fM; círculo azul, 10 fM; círculo verde, 100 fM; círculo violeta, 1 pM; círculo rosa, 10 pM; círculo azul claro, 100 pM; círculo negro, 1 nM)
Análisis interactivo dependiente de la dosis en el IDE
Tras la unión de la sonda biotinilada en la superficie de detección, se realizó el análisis interactivo del objetivo con diferentes dosis. Esta validación se realizó desde el rango femtomolar bajo hasta el rango nanomolar bajo. Para asegurar este rango, realizamos el análisis preliminar con 1 nM de secuencias diana de miARN-335-5p. Después de la adición de 1 nM de la secuencia de miARN-335-5p diana en la superficie modificada con la sonda de captura, el nivel de corriente se cambió de 1,29 E-08 A a 1,29E-07 A (Fig. 4a); este resultado indicó que se produjo la hibridación entre la sonda de captura y la secuencia diana. Después de esta confirmación, para determinar el límite de detección, titulamos la secuencia objetivo de 1 fM a 1 nM, que cayó de forma independiente en las superficies modificadas con la sonda de captura. Los cambios actuales se detectaron antes y después de la inmovilización. Las diferencias en la corriente para las hibridaciones de 1 fM, 10 fM, 100 fM, 1 pM, 10 pM, 100 pM y 1 nM fueron 2,87, 3,87, 5,04, 6,88, 8,59, 11,21 y 11,61E - 08 A, respectivamente. Según los resultados obtenidos, se detectaron cambios de corriente claros dependientes de la dosis en todas las concentraciones probadas de la secuencia objetivo (Fig. 4b).
Alto rendimiento analítico:sensibilidad, especificidad y reproducibilidad
Sobre la base de los resultados anteriores con incrementos dependientes de la concentración, se realizó un análisis de regresión lineal. La sensibilidad de la detección se estimó mediante el cálculo de 3σ, y cayó a 1 fM (Fig. 5a, b). La concentración fue menor de 1 fM, mostrando la señal de fondo y valores más bajos. Además, se realizó un análisis de especificidad para discriminar las secuencias diana de las secuencias no complementarias y con desajustes simples y triples. Era evidente que la sonda diseñada mostró una reacción más fuerte con las secuencias complementarias de miARN-335-5p perfectas que las otras secuencias (Fig. 6a). Una secuencia con un solo emparejamiento incorrecto dio como resultado una respuesta de corriente más alta que las secuencias no complementarias y con un triple desajuste debido a la formación de dúplex parcial por la secuencia de un solo emparejamiento incorrecto en la secuencia de la sonda. Se llevaron a cabo análisis de reproducibilidad con todas las modificaciones químicas y biológicas en la superficie IDE por triplicado, y se promediaron los datos. Los datos promediados indican que no hubo cambios significativos cuando se realizaron los diferentes experimentos (Fig. 6b). Además, para respaldar el rendimiento del IDE, se compararon las características de los métodos actualmente disponibles (Tabla 1).
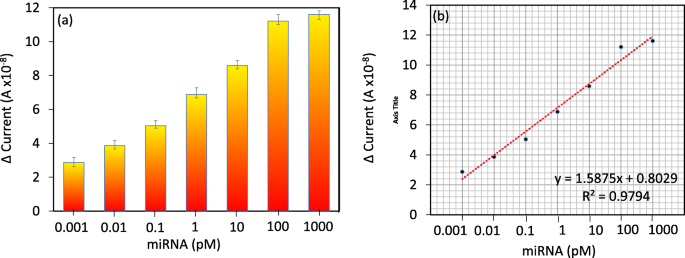
Dependencia de la dosis. un Interacción dependiente de la dosis de miARN-335-5p con la sonda. b Análisis de regresión lineal con diferentes concentraciones de miARN-335-5p. LOD se consideró la concentración más baja de un analito frente a la señal de fondo (S / N =3:1)
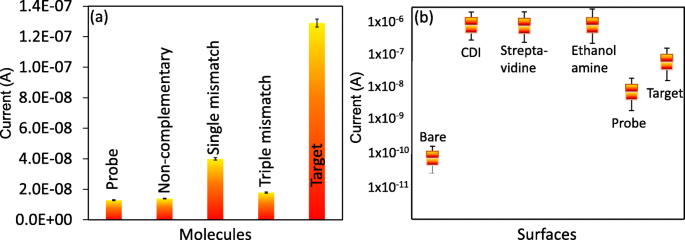
Análisis de alto rendimiento. un Análisis de especificidad. Las secuencias diana se discriminaron de las secuencias no complementarias y las secuencias con desajuste simple y triple. b Prueba de reproducibilidad. Los valores promediados se muestran con experimentos por triplicado
Conclusiones
El desarrollo de un método analítico que utiliza biomarcadores biológicos ayuda a diagnosticar el AAA durante los exámenes físicos. En la investigación actual, se desarrolló la detección mediada por miRNA-335-5p para predecir la gravedad de AAA, ya que se descubrió que miRNA-335-5p se expresaba con AAA. La salida de la detección de IDE generada se mostró de menor a mayor nivel de detección para facilitar la predicción de la gravedad de AAA. El nivel de detección más bajo fue 1 fM con alta especificidad y reproducibilidad para el análisis de alto rendimiento. El método de estreptavidina-biotina tetravalente utilizado en esta investigación produjo un buen resultado y se puede utilizar para otros análisis de biomarcadores.
Disponibilidad de datos y materiales
Todos los datos están completamente disponibles sin restricciones.
Abreviaturas
- AAA:
-
Aneurisma de aorta abdominal
- CDI :
-
1,1′-carbonildiimidazol
- PBS:
-
Solución tampón de fosfato
- IDE:
-
Electrodo interdigitado
- UV:
-
Ultravioleta
Nanomateriales
- N, N-dimetilformamida reguladora de la fluorescencia de los puntos cuánticos de MXene para la determinación sensible de Fe3 +
- El diseño de la capa de emisión para multiplicadores de electrones
- La detección fotoelectroquímica mejorada de ácido úrico en electrodo de carbono vítreo modificado con nanopartículas de Au
- Un biosensor de ADN electroquímico altamente sensible de nanocompuesto acrílico-oro para la determinación del género del pez Arowana
- Biosensor ultrasensible para la detección de ADN de Vibrio cholerae con nanoesferas compuestas de poliestireno-co-ácido acrílico
- Defectos en la superficie del nanofósforo de MgAl2O4 dopado con Ti
- La preparación de la nanoestructura yema-cáscara de Au @ TiO2 y sus aplicaciones para la degradación y detección del azul de metileno
- Aptasensor fluorescente a base de óxido de grafeno para la detección de encendido de CCRF-CEM
- ¿Cuál es el mejor acabado superficial SPI para su pieza moldeada por inyección?
- El material de electrodo adecuado para su aplicación de soldadura por resistencia
- La cura para algunos problemas comunes de unión de electrodos



