Los estudios de establecimiento y aplicación de un indicador de pH de lisosoma preciso basado en nanopartículas autodescomponibles
Resumen
El pH ácido de los lisosomas está estrechamente relacionado con la autofagia; así, bien conocido de los lisosomas precisos, los cambios de pH darán más información sobre el proceso y el estado de la autofagia. Sin embargo, hasta ahora, solo podían indicarse cambios de pH en un rango relativamente amplio, nunca llegó la detección exacta del pH de los lisosomas. En nuestro estudio, establecimos un indicador de pH de endo / lisosoma basado en el SiO 2 autodescomponible Sistema de nanopartículas con parámetros de síntesis específicos. El azul de metileno (MB) concentrado central en las nanopartículas estructurales huecas centrales presentó una liberación sensible en función de los valores de pH de 4,0 a 4,8, que es exactamente el rango de pH de los lisosomas. Se ha construido la correlación lineal de los valores de densidad óptica (DO) y los valores de pH, que se ha utilizado para la detección del pH de los lisosomas en 6 líneas celulares diferentes. Además, mediante este sistema, logramos detectar con precisión los cambios promedio de pH de los lisosomas antes y después de la endocitosis de NP de silicio mesoporoso negro (BPSi), aclarando el mecanismo de terminación de la autofagia después de la endocitosis de BPSi. Por lo tanto, el indicador de pH luminal basado en nanopartículas autodescomponibles puede proporcionar una nueva metodología y estrategia para conocer mejor el pH del lisosoma y luego indicar más detalles sobre el proceso de autofagia u otra señalización importante sobre el metabolismo.
Introducción
Los lisosomas sirven como destino final para las macromoléculas, donde estas macromoléculas son degradadas por enzimas hidrolíticas activadas por un pH bajo [1]. El pH ácido de los lisosomas mantenido por la H + -ATPasa de tipo vacuolar (v-ATPasa) [2] que bombea protones desde el citoplasma hacia el lumen lisosomal fue para mantener la actividad de ~ 60 tipos de enzimas hidrolíticas [3]. Además, los informes de la literatura reciente revelaron que el pH ácido de los lisosomas está estrechamente relacionado con la autofagia [4], por lo que los cambios de pH de los lisosomas bien conocidos darán más información sobre el proceso y el estado de la autofagia. Según nuestros estudios y la revisión de la literatura, la endocitosis de nanopartículas con carga positiva de amina probablemente aumentará el cambio de pH en los endo / lisosomas, como las nanopartículas decoradas con PEG de amina primaria y secundaria o alguna decoración hidrófila en la superficie de la partícula [5, 6].
El aumento del pH inducido por la endocitosis de las nanopartículas de amina aumentará drásticamente la localización nuclear del factor de transcripción EB (TFEB) [7], lo que da como resultado no solo una regulación positiva de la transcripción de la vía, sino que también causa una disfunción lisosomal, lo que finalmente da como resultado el bloqueo del flujo autofágico [ 7,8,9]. Como TFEB regula la autofagia y, en consecuencia, su sobreexpresión conduce a un aumento significativo en la producción de autofagosomas en células cultivadas.
Por lo tanto, para predecir el proceso de autofagia y los detalles de la autofagia, el pH preciso del lisosoma y la medición de su cambio son muy cruciales. Hasta ahora, a partir del valor de pH del endo / lisosoma que indica las revisiones de la literatura [10] y los productos comerciales para los valores de pH del endo / lisosoma detectados, solo podían indicarse cambios de pH en un rango relativamente amplio, y la detección exacta del pH del lisosoma nunca ha llegado . Por lo tanto, para conocer los detalles de la autofagia, el establecimiento de un método preciso de detección de cambios de pH luminal es un enfoque importante.
Basado en nuestras experiencias previas sobre SiO 2 autodescomponible nanopartículas, en este estudio, establecimos un indicador de pH preciso que podría realizar la detección de cambios de pH luminal. SiO 2 Las nanopartículas tienen buenas ventajas en cuanto a tamaño ajustable y biocompatibilidad [11]. Al establecer parámetros de síntesis específicos, el SiO 2 autodescomponible establecido El indicador de pH podría liberar sensiblemente la carga útil de azul de metileno (MB) en un pH de 4,0 a 4,8, que es exactamente el rango de pH de los lisosomas. Además, la liberación de MB presentó una correlación lineal con los cambios en el valor de pH (esquema 1). Luego, probamos la viabilidad del indicador de pH en los niveles celulares mediante la introducción de 6 líneas celulares diferentes, logramos determinar los cambios promedio de pH de los lisosomas antes y después de la endocitosis de NP de silicio mesoporoso negro (BPSi), aclarando el mecanismo de terminación de la autofagia después de BPSi. endocitosis. Por lo tanto, el indicador de pH luminal basado en nanopartículas autodescomponibles puede proporcionar una nueva metodología y estrategia para conocer mejor el pH del lisosoma y luego indicar más detalles sobre el proceso de autofagia u otras señales importantes sobre el metabolismo.
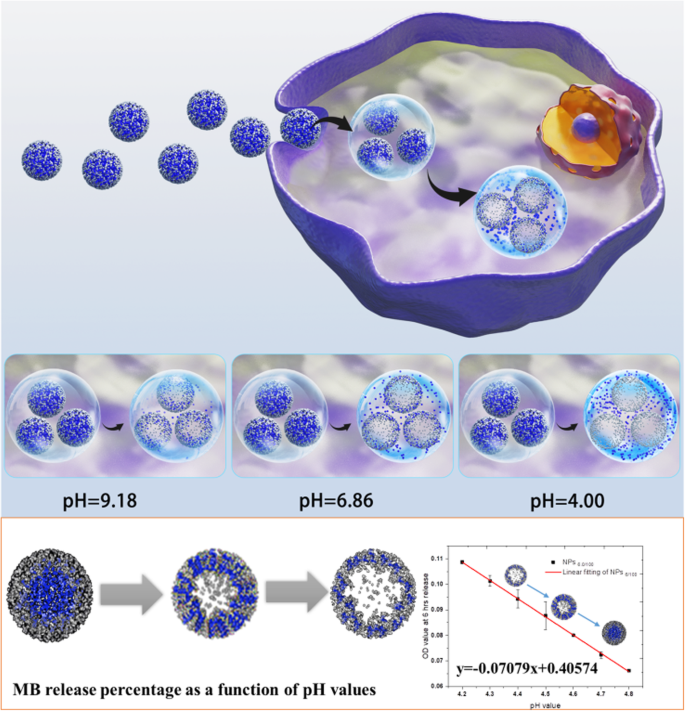
Ilustración esquemática de MB @ SiO 2 medición del pH del lisosoma en células vivas
Materiales y métodos
Sección de materiales
El siliciuro de sodio (NaSi) y las obleas de Si (diámetro 20 cm, p + (100), 0.01–0.02 Ω cm) fueron provistas por SiGNa Chemistry Inc. y Ocmetic Inc., respectivamente. Bromuro de amonio (NH 4 Br, 99%), bromuro de sodio (NaBr, 99%), tolueno (anhidro, 99,8%), ácido clorhídrico (HCl, 37%), MB y ortosilicato de tetraetilo (TEOS) se compraron a Sigma-Aldrich. Se compraron metoxi-PEG-silano de 0,5 kDa y metoxi-PEG-silano de 2 kDa de Fluorochem Ltd. y Laysan Bio Inc. por separado. El medio RPMI 1640 fue proporcionado por Life Technologies. El suero fetal bovino (FBS) se compró de la tecnología biológica TianHang. Bicarbonato de sodio, sulfato de estreptomicina, penicilina G, HEPES, solución de lisozima, CellLight Early-endosomes-GFP, LysoTracker ™ Red DND-99, Pierce® BCA Protein Assay Kit, quimioluminiscencia mejorada, pHrodo ™ rojo conjugado de transferrina, solución de imágenes de células vivas y El reactivo Trizol se compró a Thermo Fisher Scientific. Sinopharm proporcionó etanol y amoníaco-agua. El kit BioRT Master HiSensi cDNA First-Strand Synthesis se compró en Hangzhou Bioer Technology Co., Ltd. El lisado RIPA se compró en Heart Biological Technology Co., Ltd. P62, TFEB y el anticuerpo β-actina se compraron en Proteintech Group , Inc. El anticuerpo LC 3B fue proporcionado por Abcam. El 2- (4-piridil) -5 - ((4- (2-dimetilaminoetilaminocarbamoil) metoxi) fenil) oxazol (PDMPO) fue proporcionado por Yeasen Biotech Co., Ltd.
Objetivo, diseño y entorno del estudio
El objetivo de este estudio es (1) explorar la influencia de las nanopartículas de BPSi en la autofagia en las células HepG2, (2) descubrir el mecanismo subyacente de los cambios en el valor del pH del lisosoma que afectan a la autofagia, (3) establecer un indicador preciso del pH del lisosoma que pueda medir el pH de los lisosomas con precisión y, finalmente, (4) indican la influencia de la fluctuación del pH en la autofagia. Para realizar el propósito del estudio anterior, empleamos el experimento de secuenciación del transcriptoma para explorar los cambios del gen del transcriptoma en las células HepG2 después de alimentar nanopartículas de BPSi y lo verificamos mediante RT-qPCR y experimentos Western. Se utilizaron tintes fluorescentes como PDMPO para medir el cambio de pH lisosómico en células HepG2 después de la alimentación con BPSi. Para medir el pH de los lisosomas con precisión, desarrollamos MB @ SiO 2 nanopartículas con 10 parámetros y probaron las caracterizaciones de estos 10 tipos de nanopartículas a través de experimentos como DLS y HR-TEM. El estudio de la eficiencia de carga de MB y la cinética de liberación de los sistemas de nanopartículas autodescomponibles de la serie 10 se probaron en diferentes soluciones de pH y células HepG2. Para verificar la ubicación intracelular de las nanopartículas después de ingresar a la célula, hicimos un experimento de TEM celular y microscopía confocal de células vivas. Finalmente, medimos los cambios de pH lisosómico en 6 tipos de células después de alimentar BPSi para verificar la universalidad de MB @ SiO 2 nanopartículas para medir los cambios de pH lisosómico.
Síntesis de nanopartículas BPSi
Las nanopartículas de BPSi se prepararon mediante nuestro método anterior [12] y fueron suministradas por nuestro colaborador (Wujun Xu, Departamento de Física Aplicada, Universidad de Finlandia Oriental). La preparación de BPSi, NaSi, sal de amonio y NaBr (NaSi:NH 4 Br:NaBr de 1:4:4, p / p / p) se molió en una caja de guantes con una atmósfera de Ar. Se les permitió reaccionar en un horno de tubo bajo un N 2 atmósfera a 240 ° C durante 5 h (ecuación 1). Después de enfriarse a temperatura ambiente, las micropartículas obtenidas se purificaron enjuagando con soluciones de HCl 0,5 M y HF 1,0 M por separado. Las micropartículas se molieron en bolas en etanol a 1000 rpm durante 15 min, y las nanopartículas de BPSi con el diámetro deseado se recolectaron ajustando la velocidad de centrifugación.
$$ \ mathrm {NaSi} + {\ mathrm {NH}} _ 4 \ mathrm {Br} \ to \ mathrm {NaBr} + {\ mathrm {NH}} _ 3+ \ mathrm {Si} / \ mathrm {H} + { \ mathrm {H}} _ 2 $$ (1)Mediante el experimento Dynamic Light Scattering, se estudiaron la distribución del diámetro y la carga superficial de la nanopartícula. Todos los NP se dispersaron en el medio después de la esterilización mediante ultrasonidos leves (5 s para que se dispersen uniformemente en la solución, limpiador ultrasónico SB-5200DT, Ningbo Scientz Biotechnology Co., Ltd.) justo antes de su introducción en las células.
Establecimiento del sistema de nanopartículas autodescomponibles de la serie 10
Las nanopartículas autodescomponibles de la serie 10 se sintetizaron mediante las metodologías que informamos antes [13,14,15,16], con parámetros modificados. En un procedimiento típico, primero se agrega una cierta cantidad de MB a una mezcla de etanol (75 mL) con una solución de amoníaco-agua (25%, 3.4 mL), luego se agrega cierta cantidad de TEOS. La serie MB @ SiO 2 autodescomponible Los NP se obtuvieron después de agitar durante 24 h y se lavaron 3 veces antes de secarlos. Las cantidades de MB y TEOS agregadas en los protocolos se describen en la Tabla 1. El significado de 1.0 / 100 en los NP 1.0 / 100 representó el inventario de MB y TEOS cuando sintetizamos las nanopartículas, con 1.0 mg de MB y 100 μL de TEOS. Y los significados de 1.5 / 100 en los NP 1.5 / 100 y otros son consistentes con el de 1.0 / 100.
Cultivo celular
Para probar la eficiencia y universalidad del indicador de pH basado en nanopartículas autodescomponibles, intentamos probarlo en líneas celulares de cáncer específicas derivadas de tumores. Por lo tanto, seleccionamos líneas celulares de cáncer de hígado, cáncer de pulmón, cáncer de colon y melanocitoma como objetos de investigación. Las líneas celulares de células de cáncer de colon humano HCT116, HCT8 y HCT15; células de cáncer de hígado humano HepG-2; células de cáncer de pulmón humano A549; y las células de melanoma de ratón B16 se mantuvieron en medio RPMI 1640 (Life Technologies) suplementado con FBS inactivado por calor al 10%, bicarbonato de sodio 2,0 g / L, sulfato de estreptomicina 0,1 g / L, penicilina G 0,06 g / L y 5,958 g / L HEPES. Las células se mantuvieron en una incubadora de cultivo celular estándar a 37 ° C en una atmósfera humidificada con 5% de CO 2 .
Caracterizaciones de los sistemas de nanopartículas autodescomponibles de la serie 10
La morfología de todas las nanopartículas de la serie se caracterizó por HR-TEM con modo STEM, y el mapeo de Si se estudió mediante mapeo de elementos EDS. El análisis de distribución del tamaño de las nanopartículas se realizó mediante el software ImageJ calculando los diámetros de las nanopartículas en imágenes STEM seleccionadas al azar. El potencial zeta de las nanopartículas y el índice de polidispersidad (PDI) se ha medido mediante el estudio de dispersión dinámica de luz (DLS), en tampones en serie con valores de pH específicos. Los datos se analizaron con SPSS15.0 y los resultados estadísticos se presentaron como media ± SD
Estudio de cinética de liberación y eficiencia de carga de MB de los sistemas de nanopartículas autodescomponibles de la serie 10
Para estudiar la eficiencia de carga de MB y la cinética de liberación, primero se estableció la curva estándar de MB en concentraciones en serie. La absorción de MB se realizó mediante espectro UV-Vis con la absorbancia a 660 nm, que es la λ max del monómero MB. La eficiencia de carga de MB se calculó mediante la siguiente ecuación, eficiencia de carga de MB (%) =la cantidad de MB encapsulados / (cantidad total de MB de entrada).
La liberación de MB de las nanopartículas de la serie 10 se estudió en agua pura y tampones de pH con diferentes valores de pH (pH 4,0, pH 6,86 y pH 9,18) y solución de lisozima (Thermo Scientific ™ # 90082). Además, también se investigó la cinética de liberación de MB después de una duración específica en diferentes tampones de pH. Luego se estudiaron los valores de DO a 660 nm y los porcentajes de liberación de MB en función del tiempo.
Más detalladamente, los estudios de liberación de MB se realizaron mediante los protocolos siguientes; nanopartículas de la serie 10 disueltas en 15 ml de tampón estándar de pH 4,0, 6,86 y 9,18 con solución de lisosoma, respectivamente; y realizó la liberación de MB en un mezclador Hula a 37 ° C. Durante los siguientes 15 días, se recogió 1 mL de cada muestra, luego se centrifugó a 12000 rpm durante 10 min. Se midieron el sobrenadante y el precipitado en cuanto a sus espectros de absorción a 200-800 nm.
Además, la liberación de MB en tampones de pH precisos con solución de lisozima en el rango de pH de 4,1 a 5,5 también se investigó con los mismos protocolos anteriores. Las duraciones de tiempo específicas (6 h, 12 h y 24 h) se colocaron como puntos de tiempo de observación. Las absorciones a 660 nm se registraron en cada muestra. La relación lineal de absorción de cada solución de pH y la suma de los cuadrados de los residuos se contaron en cada punto de tiempo, respectivamente.
Para detectar los perfiles de liberación de MB en las células, se cultivaron células HepG-2 en un 2 de 75 cm matraz de cultivo y se alimentó con NP (300 μg / mL) cuando las células proliferaron al 70% del matraz de cultivo. Cada 30 minutos más tarde, se recolectaron las células. Las células se congelaron y descongelaron repetidamente para liberar completamente el MB en las células. Los lisados celulares se centrifugaron a 12000 r / min durante 10 min. Se obtuvo el sobrenadante y se midió su absorbancia a 660 nm para calcular la cantidad total de MB liberado. En este estudio, las células HepG2 fueron elegidas como objetos de investigación, debido a su rápida proliferación celular, lo que podría minimizar la variación entre los 10 grupos evaluados.
Colocalización celular de las nanopartículas de la serie 10 y rendimiento de liberación en 6 líneas celulares diferentes
Se empleó el TEM celular para estudiar la colocalización de la nanopartícula en los endo / lisosomas siguiendo los protocolos estándar de TEM celular. Las células se sembraron con una intensidad de 1 × 10 6 células / matraz y se incubaron durante 24 h, permitiendo la fijación celular. Se incubaron diez series de nanopartículas en medio con la misma concentración (100 μg / mL) con las células durante otras 12 y 24 h, respectivamente. A continuación, las células se lavaron con PBS 3 veces para eliminar el exceso de nanopartículas, luego se fijaron en una solución de glutaraldehído al 2,5% durante más de 1 día. A continuación, las células fijadas se lavaron y se tiñeron con tetróxido de osmio, 1% en agua desionizada durante 1 h, seguido de lavado con PBS 3 veces y agua desionizada 2 veces. A continuación, se llevó a cabo el protocolo clásico de TEM celular [17, 18], y se recogieron secciones con un grosor de 90 nm para la observación de TEM. Se estudió la liberación de MB en función de los valores de pH en 6 líneas celulares con ambos NP 6/100 y NP 7.5 / 80 . Además, los valores de DO de la liberación de MB y los porcentajes de liberación de MB se registraron para el análisis de datos.
Investigación de la absorción intracelular de MB @ SiO 2 Nanopartículas
Se utilizó microscopía confocal de células vivas para evaluar la captación celular y el destino intracelular de MB @ SiO 2 nanopartículas. Los endosomas tempranos de células HepG-2 se tiñeron (CellLight Early-endosomes-GFP, BacMam 2.0 ThermoFisher Scientific C10586, con excitación / emisión ~ 488/510 nm) durante 16 h. Y luego las células se incubaron con NP 6/100 (Excitación / emisión de MB:640 / 650-700 nm) a la concentración de nanopartículas de 100 μg / mL en intervalos de tiempo específicos (2 h, 2,5 h, 3 h, 5 hy 6 h). Antes de tomar las imágenes, el lysotracker se tiñó con LysoTracker ™ Red DND-99 (Thermo Fisher Scientific L7528, excitación / emisión:577/590 nm) durante 40 min. Después de eso, retire la solución de tinción y lave las células 2-3 veces en PBS. Las imágenes se tomaron con un microscopio confocal Nikon A1R.
Secuenciación de transcriptomas para evaluar el cambio en la expresión genética después de la alimentación con BPSi
La extracción de ARN total del grupo de control y del grupo tratado con BPSi se realizó utilizando el reactivo Trizol siguiendo los procedimientos operativos estándar. La calidad de la muestra de ARN total inicial para el experimento de secuenciación se detectó utilizando un espectrofotómetro NanoDrop ND-2000. El ARN total que pasó el control de calidad se utilizó en experimentos de secuenciación posteriores. Se realizó una comparación de la expresión génica mediante secuenciación de próxima generación. Todos los programas de secuenciación fueron realizados por BGI-Shenzhen Corporation (Shenzhen, China) utilizando la plataforma BGISEQ-500. Los datos brutos obtenidos por secuenciación se llevan a cabo mediante un control de calidad para determinar si los datos de secuenciación son adecuados para un análisis posterior. Si se aprueba, realice un análisis cuantitativo de genes según los niveles de expresión génica y realice un análisis de enriquecimiento significativo de las funciones de ontología génica (GO) en los genes expresados diferencialmente entre las muestras seleccionadas.
Ensayo de reacción cuantitativa en cadena de la polimerasa con transcripción inversa (RT-qPCR) para confirmar la activación de la red de genes TFEB-CLEAR
El ARN total se extrajo de las células cultivadas HepG-2 de los grupos de control y tratados con BPSi usando el reactivo Trizol y se transcribió de forma inversa a ADNc usando el kit de síntesis de primera hebra de ADNc BioRT Master HiSensi (Hangzhou Bioer Technology Co., Ltd .) con cebadores aleatorios. Se utilizó ADNc para amplificar la red de genes TFEB-CLEAR mediante PCR cuantitativa con el sistema de PCR en tiempo real Applied Biosystems ™ 7500 (Applied Biosystems, Life Technologies, Carlsbad, CA) con actina como control de referencia. Los cebadores utilizados para la RT-PCR cuantitativa se enumeraron en la Tabla S4.
Ensayo de transferencia Western para confirmar que la autofagia se activa después de la alimentación con BPSi
Las proteínas celulares del grupo de control y los grupos tratados con BPSi de diferente concentración se extrajeron mediante lisado de RIPA (Heart Biological Technology Co., Ltd.). El inhibidor de proteasa se añadió al lisado de RIPA y se enfrió previamente en hielo. Se lavaron las células 3 veces con PBS preenfriado. Se vertió el líquido por completo y se colocó el plato en hielo durante 2 min. Se agregaron cuatrocientos microlitros de lisado de RIPA a la superficie de toda la placa, se pipetearon varias veces con una pipeta y se incubaron en hielo durante 30 minutos, durante los cuales la placa se agitó varias veces para lisar completamente las células. El fluido celular lisado se transfirió a tubos Eppendorf de 1 ml y se centrifugó a 13.000 rpm durante 10 min, 4ºC. El sobrenadante obtenido se hirvió en agua durante 10 min y se colocó a -20 ° C para su uso posterior. La concentración de proteínas se cuantificó utilizando el kit de ensayo de proteínas Pierce® BCA (Thermo Scientific).
Los extractos de células que contenían 25 µg de proteína total se sometieron directamente a SDS-PAGE y se transfirieron. Las membranas se bloquearon con leche desnatada al 5% y se probaron con anticuerpos primarios que reconocen P62 (Proteintech # 18420–1-AP), TFEB (Proteintech # 13372–1-AP), LC 3B (Abcam # ab192890) y β-actina (Proteintech # 20536–1-AP). Los anticuerpos secundarios se eligieron de acuerdo con la especie de origen de los anticuerpos primarios y se detectaron mediante quimioluminiscencia mejorada (Pierce) o utilizando el sistema de imágenes en gel Bio-Rad ChemiDoc XRS + (Bio-Rad, EE. UU.). La intensidad de banda normalizada de P62, TFEB y LC 3B en relación con la β-actina se cuantificó mediante densitometría utilizando el software ImageJ en los grupos de BPSi, y los datos son la media ± S.D. a partir de tres experimentos independientes.
Medición del pH celular mediante PDMPO y el conjugado de transferrina roja pHrodo ™ mediante microscopía confocal
Estudio PDMPO
1 × 10 5 Las células HepG-2 se cultivaron en placas confocales estériles durante la noche y las nanopartículas de BPSi se alimentaron con una concentración de 100 µg / ml. Al día siguiente, antes de la tinción por inmunofluorescencia, los portaobjetos se lavaron tres veces con solución salina tamponada con fosfato (PBS) 0,01 M, pH 7,4, luego se añadió colorante PDMPO 1 µM (Ex / Em =329/440). Después de lavar con PBS tres veces, las células se incubaron con medio de cultivo RPMI-1640 fresco y se observaron bajo un microscopio de fluorescencia (Nikon A1R, Japón) con una cámara CCD y se tomaron fotografías en 5 minutos, y la proporción de la intensidad de fluorescencia azul y verde. en los lisosomas se calculó luego de acuerdo con el procedimiento de Chen et al. [19].
Estudio de conjugado de transferrina roja pHrodo ™
Las células HepG-2 se sembraron en placas confocales de la misma manera para la unión celular durante 24 h, luego se mantuvieron las placas en hielo durante 10 min. Las células se lavaron con una solución de imágenes de células vivas fría que contenía glucosa 20 mM y BSA al 1%. Se agregó el conjugado pHrodo ™ Red Transferrina (Ex / Em =560/585 nm) a 25 μg / mL en solución de imágenes de células vivas e incubar a 37 ° C durante 20 min, luego se lavaron las células en solución de imágenes de células vivas. La observación también se realizó mediante microscopía confocal. El análisis cuantitativo de la intensidad de las imágenes microscópicas se realizó mediante el software ImageJ.
Detección de pH lisosómico celular
Las células A549, HepG-2, HCT8, HCT15, HCT116 y B16 se cultivaron en un 2 de 75 cm matraz de cultivo y se alimentó con NP cuando las células proliferaron hasta el 70% del matraz de cultivo, y 6 h más tarde, se recogieron las células. Las células se congelaron y descongelaron repetidamente para liberar completamente el MB en las células. Los lisados celulares se centrifugaron a 12000 r / min durante 10 min. Se obtuvo el sobrenadante y se midió su absorbancia a 660 nm para calcular la cantidad total de MB liberado. La absorbancia de los NP 6.0 / 100 a un pH estándar se comparó para obtener el valor de pH de cada celda.
Análisis estadístico
El análisis estadístico se realizó con el software SPSS15.0 utilizando un análisis de varianza de dos vías (ANOVA) para grupos independientes y utilizando el método Tukey HSD para la prueba de comparaciones múltiples. La significancia estadística se basó en un valor de P <0.05.
Resultados y discusión
En primer lugar, detectamos la expresión génica diferencial cuando las células se alimentaban con nanopartículas de sílice porosa negra funcional PEG dual (NP BPSi) (ofrecidas por nuestro laboratorio colaborador [12]). El cambio del potencial zeta de -18,5 a + 2,8 mV también indicó que la PEGilación dual de superficie fue exitosa (Fig. S1a). El diámetro medio de las nanopartículas de BPSi fue de 156 nm (Fig. S1b). Basándonos en el mapa de calor de grupos de expresión génica diferencial (Fig. S2), seleccionamos más de 2 veces las expresiones génicas diferenciales para una investigación adicional. Se introdujeron Go y KEGG para analizar los genes diferenciales. A partir de los mapas de burbujas de enriquecimiento de Go (Fig. 1a), se seleccionaron los genes metabólicos y asociados a lisosomas, incluidos el ensamblaje de fagolisosomas, la fagocitosis y el proceso metabólico xenobiótico para su análisis adicional. En particular, las expresiones génicas asociadas a TFEB-CLEAR [17] aumentaron significativamente. Los resultados de RT-PCR (Fig.1b) también verificaron los resultados de secuenciación de genes, los genes en TFEB, la expresión lisosomal coordinada, y la vía de regulación (CLEAR) aumentaron significativamente, como CTSD, CTSF, TFEB, MFN1, LAMP2 y TPP1. . Estos genes se han marcado en la ruta del lisosoma, como se muestra en la Fig. S3. Su expresión es superior a la del grupo de control y tiene significación estadística ( P Control BPSi VS <0,05). Y TFEB regula positivamente la expresión de genes lisosomales, controla la población de lisosomas y promueve la degradación celular de sustratos lisosomales.
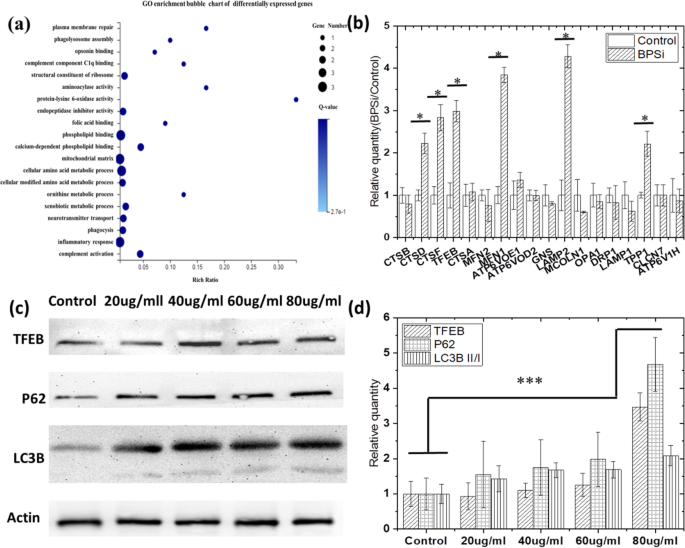
La autofagia se activa en las células HepG-2 después de la alimentación con BPSi. un GO mapa de burbujas de enriquecimiento de los genes expresados diferencialmente encontrados por secuenciación del transcriptoma. b Verificación de cambios genéticos en la red de genes TFEB-CLEAR después de BPSi tratado mediante experimentos RT-qPCT. c Expresión de la proteína P62, TFEB, LC3B II / I después del tratamiento con BPSi. d Intensidad gris de banda normalizada de proteína P62, TFEB, LC3B II / I en grupos tratados con BPSi según el grupo de control. Los datos se presentaron como media ± DE.
Además, TFEB regula la autofagia, y su sobreexpresión conduce a un aumento significativo en la producción de autofagosomas en células cultivadas debido al hecho de que la función principal del gen TFEB es inducir la biosíntesis de lisosoma y promover la aparición de autofagia [20]. También se empleó el análisis de transferencia Western para atestiguar si la autofagia ocurrió cuando las células se alimentaron con NP de BPSi. El propósito de la prueba de Western fue confirmar además que la expresión de TFEB aumentó y la aparición de autofagia celular después de la alimentación con BPSi. Las proteínas LC3B y P62 son ambas marcadores de autofagia. Cuando se produce la autofagia, aumenta la expresión de la proteína asociada a microtúbulos 1A / 1B-cadena ligera 3B (LC3B) II / I. p62 es un receptor de vesículas que se degradarán por autofagia y también es un receptor de agregados de proteínas ubiquitinados que se eliminarán, y su expresión disminuye cuando ocurre la autofagia. Entonces medimos la expresión de estas proteínas en el experimento occidental.
A partir de los resultados de la transferencia Western que se muestran en la Fig. 1c, d, TFEB ( P 80 μg / ml de control VS =0,000008), LC3B II / I ( P 80 μg / ml de control VS =0.000297) y p62 ( P 80 μg / ml de control VS =0,000016) proteínas todas significativamente reguladas positivamente. Como la regulación positiva de las proteínas TFEB y LC3B II / I indica la activación de la autofagia [18], sospechamos que la endocitosis BPSi promueve la aparición de autofagia. Sin embargo, se supone que la proteína p62 está regulada negativamente durante el proceso de autofagia, debido a la naturaleza de la proteína transportadora que llevó los endosomas a los lisosomas y finalmente se degradaron. En nuestro estudio, la importante regulación positiva de p62 indicó la terminación de la degradación durante el proceso de fusión de endolisosoma [21], que probablemente se debe al aumento del pH en las vesículas de endosomas. Por lo tanto, la endocitosis de BPSi puede primero inducir la aparición de autofagia, luego inhibir el proceso de autofagia al aumentar los valores de pH del endo / lisosoma, debido a su alcalinidad de amida.
Para atestiguar las características de aumento de pH en endo / lisosomas por endocitosis BPSi, se emplearon dos sondas fluorescentes de pH comerciales en nuestro estudio, pHrodo ™ Red Transferrin Conjugate (Thermo Fisher # P35376) y RatioWorks ™ PDMPO.
pHrodo ™ Red, como indicador comercial de pH intracelular, generalmente presenta una fluorescencia débil a pH neutro, pero aumenta la fluorescencia a medida que desciende el pH. Se suponía que debía cuantificar el pH citosólico celular en el rango de 9–4 con un pKa de ~ 6,5 con excitación / emisión de 560/585 nm. Pudimos obtener una conclusión de análisis cualitativo a partir de 6 determinaciones de líneas celulares de que la endocitosis de NP de BPSi tiene la capacidad de aumentar los valores de pH en endo / lisosomas, debido al debilitamiento de las señales fluorescentes rojas (Fig. S4 y S5). Sin embargo, después de repetir los experimentos varias veces siguiendo los protocolos de operación del producto, apenas analizamos cuantitativamente el valor exacto del pH que disminuyó entre las diferentes líneas celulares antes o después de la alimentación con NP BPSi debido a que no había correlación entre la intensidad y los valores de pH establecidos.
Luego se empleó PDMPO como una mejor solución para indicar los cambios del valor de pH después de la endocitosis de BPSi, que introduce técnicas de formación de imágenes de relación en la medición cuantitativa del pH. PDMPO [2- (4-piridil) -5 - ((4- (2-dimetilaminoetilaminocarbamoil) metoxi) fenil) oxazol] se caracteriza como sonda de pH acidotrópica de doble excitación y doble emisión. Emite una fluorescencia verde intensa a un pH más bajo y da una fluorescencia azul intensa a un pH más alto. Esta fluorescencia única dependiente del pH hace que la PDMPO sea una sonda de pH ideal para orgánulos ácidos con pKa =4,47. PDMPO marca selectivamente los orgánulos ácidos (como los lisosomas) de las células vivas y los dos picos de emisión distintos se pueden utilizar para controlar las fluctuaciones del pH de las células vivas en las mediciones de proporción. Sin embargo, todavía fallamos en medir los valores de pH en 6 líneas celulares antes y después de la alimentación con BPSi. Como se muestran los resultados en la Fig. S6, no se observaron diferencias significativas en las 6 líneas celulares antes y después de la alimentación con BPSi. Aunque se ha establecido una correlación entre la relación azul / verde y los valores de pH (Fig. S7), la correlación no lineal de pH 4-5 hace que el método PDMPO falle en el análisis cuantitativo de endo / lisosomas antes y después de las alimentaciones con BPSi.
A partir de los datos de dos indicadores de pH comerciales anteriores, demostramos en primer lugar nuestro sospecha de que las nanopartículas decoradas con PEG con amida en la cadena de PEG podrían hacer que el pH del endo / lisosoma aumente debido a la naturaleza alcalina de la amida. Sin embargo, sin el análisis cuantitativo de los cambios precisos del pH (rango de pH 0,1), todavía no podemos establecer las correlaciones entre el estado de autofagia y los valores de pH del endo / lisosoma, por lo que no se pudo predecir la autofagia.
Basándonos en nuestro estudio anterior sobre nanopartículas autodescomponibles [13, 14] [15, 22], mantuvimos la misma concentración de hidróxido de amonio en etanol al 75%, pero ajustamos las concentraciones de MB y TEOS. Se han establecido dos series de cantidades de TEOS en 100 μL y 80 μL, para obtener diferentes espesores de capa y tamaño de poro. Se han establecido diez series de cantidad de MB para obtener diferentes tamaños de la estructura central hueca y la eficacia de carga de MB.
Las cantidades de MB y TEOS agregadas en los protocolos se describen en la Tabla 1 a continuación.
Como se muestra en la Fig. 2a, b, el tamaño de las nanopartículas aumentó con el aumento de la cantidad de MB, en ambas concentraciones de TEOS (100 μL y 80 μL). A la misma concentración de MB, el tamaño de partícula aumentó con el aumento de la cantidad de TEOS. Moreover, with the increase of the TEOS amount, the shell thickness grew up, which has been proved by the element mapping (shown as Fig. 2c). The polydispersity index (PDI) and surface charge of the nanoparticles are shown in Fig. S8 and Table S1. The morphology studies predicted that with the increase of MB amount, the loading efficiency will grow up, leading to the faster release profile, while with the increase of TEOS amount, the release will slow down. And we need to find out the appropriate MB and TEOS concentration, with which we could obtain the optimized nanoparticle systems, that we may be able to make the MB release profile linear correlated with the pH changes.

Morphology characterization of 10 different self-decomposable nanoparticles with specific MB or TEOS amount. un STEM figures. b Nanoparticle size distribution analysis. c Si mapping of 10 self-decomposable nanoparticles. Scale bars in all figures are 100 nm. The size distribution analysis was performed by randomly chosen 100 nanoparticles from STEM figures and measured by ImageJ software. Data was presented as mean ± S.D.
The MB loading efficiency was determined by UV-Vis spectrum. The standard curve (Fig. S9) of MB was firstly drawn using series concentrations of MB solution (from 6.25 to 46.88 μg/mL), with the equation as y = 67.63x + 0.10919, R 2 = 0.9987. As calculated with the equation above, we obtain MB loading efficiency of 10 self-decomposable nanoparticles with specific parameters, detailed data shown in Fig. S10.
Before the study of the MB release profiles in different pH solutions, the release profiles in pure water have been studied. As shown in Fig. S11 and Fig. S12, all the nanoparticles with TEOS amount of 80 μL presented increased MB release along with the duration increase, which was reflected by the UV–Vis absorption. Moreover, with the MB encapsulated amount increase, the growth trend of MB release becomes more significant. Also, the release velocity grows faster. However, as the TEOS amount increase to 100 μL, the particle surface became more densed and the release becomes slower when the MB amount below 3.0 mg; almost no increase trend could be observed in the MB release in water during 14 days of release. As long as the MB amount increases to above 4.0 mg, an obvious increase trend of MB release could be observed. One thing to be noticed is that the nanoparticle parameter of both NPs7.5/80 and NPs6.0/100 presented solid growth as the time prolongs, almost showed a linear increase trend during the first 7 days, and then reached the platform.
Then, we focused on the MB release behavior in different pH buffers to figure out whether self-decomposable nanoparticles with specific parameters could have the linear pH-dependent MB release.
Firstly, we carried out the MB release experiments at pH 4.0 buffer solution. From Fig. S13, we could easily reach the conclusion that with the same TEOS amount of 100 μL, the MB release velocity presented a similar trend was observed in the 5 nanoparticle systems of TEOS at 80 μL (Fig. S14), the center positive correlation with the MB encapsulated amount.
Concentrated MB diffuses into the surrounding solution via diffusion due to concentration difference. The bigger concentration gradient makes the faster MB release. Compared with the MB release in pure water, we found that the acidic environment speeded up the release of MB (Fig. S13 and S14 compared with Fig. S11 and S12), indicating that the MB release is not only driven by diffusion; however, in acidic solutions, electrostatic repulsion is also an important driven force due to the positive charge nature of MB. We then calculated the release percentage of each nanoparticle parameter according to the MB loading efficiency, MB standard curve, and the dilution ratio at measurements. The release percentage reflected the release speed of MB in pH 4.0 acidic solution, and the results (Fig. 3) showed that only the release percentage of NPs7.5/80 presented linear release in pH 4.0 solution. Other nanoparticle systems with specific MB and TEOS parameters showed similar release trends, and the release percentage did not have a linear growth. One exception is NPs6/100 , and the MB release reached the platform in only 72 h; thus, it was hard to tell whether the MB release could grow linear before that duration at this stage.
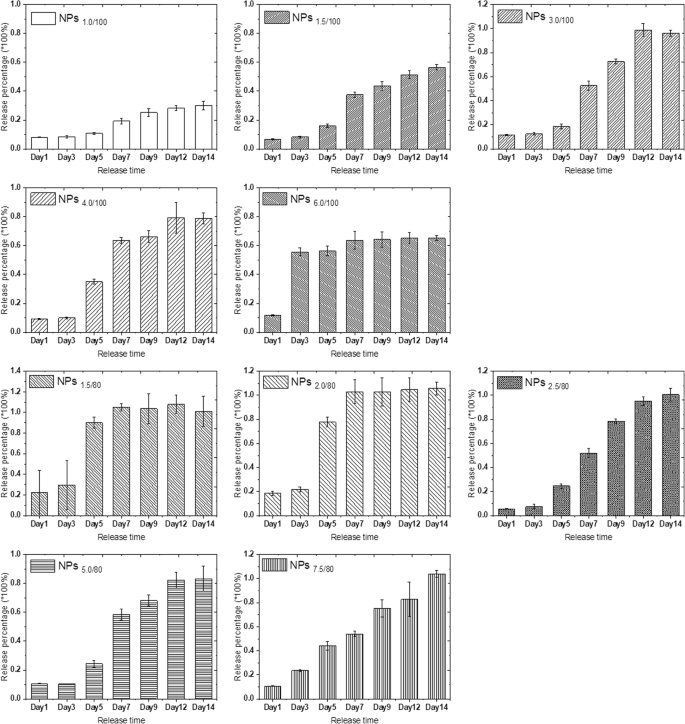
MB release percentage of 10 series self-decomposable nanoparticles after a specific duration in pH 4.0 buffer. All experiments were triple repeated, and the data were shown as mean ± S.D.
Meanwhile, we tested the MB release profiles in near-neutral and alkali buffers (pH 6.86 and pH 9.18). The results in both Fig. S15, S16, Fig. S17, and S18 demonstrated that the MB release slowed down with the solution pH increase to 6.86; moreover, with the central MB concentration increased, the MB release percentage decreased. At pH 9.18, all nanoparticles with 10 specific parameters presented a very slow MB release (Fig. S19 and S20); no matter in UV–Vis absorption or the release percentage, the trend was similar with the one in pH 6.86 buffer, but with even lower release percentage. So, it was clear that the self-decomposable nanoparticles only presented MB release linear growth in acidic solutions. We thought back to the endo/lysosomes pH, from 4 to 5, which is exactly the pH range of MB linear growth as a function of time in a specific MB/TEOS parameter. Thus, we get more confident that the self-decomposable nanoparticle system may be an accurate measuring tool for quantitative determining the endo/lysosome average pH, then provide evidence on the exact pH value of autophagy status.
The precondition of using the specific self-decomposable nanoparticles as an endo/lysosome pH indicator is that the nanoparticles stay stable in the endo/lysosome during the whole measurement process. Secondly, the MB release in endo/lysosome should occur smoothly when the measurement carried out.
The colocalization of the nanoparticle in the endo/lysosomes by cell TEM study and the MB release in 6 different cell lines were studied. From the cell TEM results, all of the 10 series nanoparticles stayed in the endo/lysosomes without escaping, after 24-h incubation with the HepG-2 cells (Fig. 4). Since the diameter of HepG2 cells used in Fig. 4 is about 10–20 μm and the diameter of MB@SiO2 nanoparticles is between 75 and 200 nm, it will be very difficult to clarify the nanoparticle morphologies using the images with low magnification (as shown in Fig. S21). We also investigated the intracellular location of the nanoparticles in the other 5 cell lines, and 4 nanoparticles were randomly selected to demonstrate the nanoparticles were trapped in the endo/lysosomes (Fig. S22). The nanoparticles with all parameters showed a central hollow structure in all other 5 cell lines after 24-h incubation, indicating the MB release. Moreover, under more precise observation, we noticed the MB release may be different due to different hollow sizes, pointing to the fact that (1) the endo/lysosome pH in different cells is different and (2) the MB release from the nanoparticles is very sensitive to the endo/lysosomes pH, especially for the NPs6/100 and NPs7.5/80 . From Fig. S23, we can find that nanoparticles have already realized the endocytosis and stayed in the vesicles 2 h after nanoparticle feeding, then nanoparticles gradually accumulated in lysosomes.
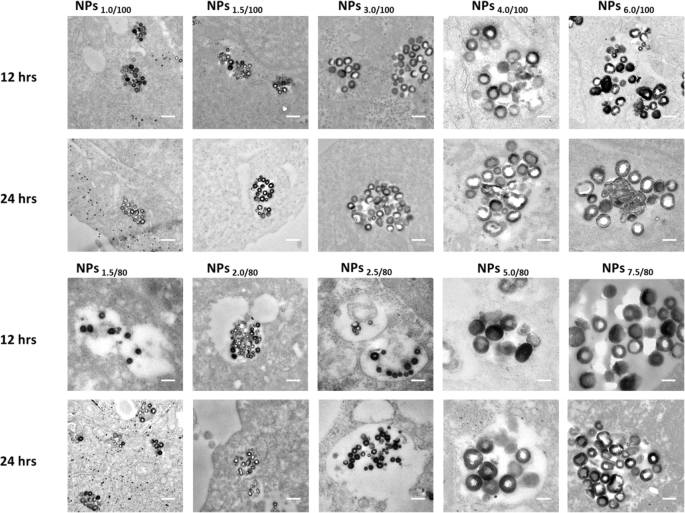
The colocalization of the nanoparticle in the endo/lysosomes by cell TEM study after 12 and 24 h incubation with the 10 series nanoparticles. The scale bar is 200 nm in the TEM images
We then evaluated the correlation between the pH values and the OD values in pH 4.0–4.8. From the results in Fig. 5 and Fig. S24, for NPs6/100 and NPs7.5/80 nanoparticle systems, the MB release presented a linear decrease as a function of pH in the pH range from 4.0 to 4.8.
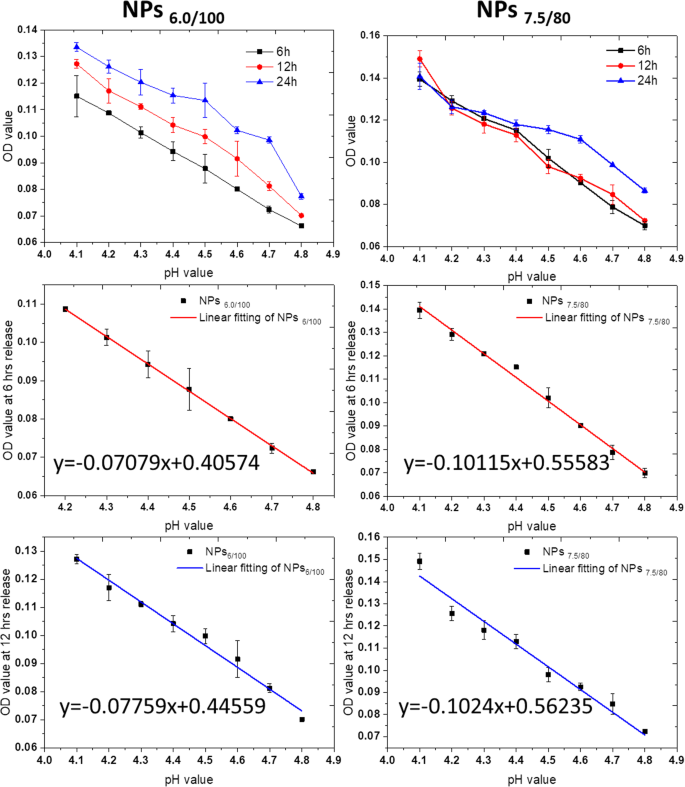
MB release as a function of pH values in NPs6/100 and NPs7.5/80 after specific incubation duration, 6 h, 12 h, and 24 h. The linear correlation equations were also calculated for 6 h and 12 h for MB release from both NPs6/100 and NPs 7.5/80 as a function of pH values. All experiments were carried out triplicated, and the data were shown as mean ± S.D.
We then converted the OD value to the MB release percentage according to the MB loading efficiency and feeding amount. As shown in Fig. S25, in the first 6 h, the MB release percentage in NPs6/100 and NPs7.5/80 nanoparticle systems also presented as a function of pH values. We then calculated the residual sum of squares and Pearson’s related coefficient at 6 h and 12 h release duration, respectively, as the residual sum of squares present a negative correlation with closeness of linear fitting, while the closer the absolute value of Pearson’s related coefficient to 1, the more linear it is. As shown in Table S2 and S3, the highest degree of linearity is the fitting of NPs6.0/100 nanoparticle systems, followed by the one of NPs7.5/80 at 6 h release.
Till then, we were so excited by the results that the method for precisely monitoring the pH values has been established, especially with the accuracy less than or equal to 0.1 pH value interval. That means, we have great possibilities to figure out the correlation between endo/lysosome pH values and the autophagy status, which is of great significance for better studying the autophagy mechanism and predicting the autophagy process. As we can see in Fig. S26, the MB release in HepG-2 cells have already reached the plateau after incubation for 4 h. Thus, we chose 6 h as the observation time point.
We then carefully investigated the MB release of NPs6.0/100 in 6 cell lines in the nanoparticle cell interaction duration of 6 h, including liver cancer HepG-2 cell line, colon cancer HCT8, HCT 15, and HCT 116 cell lines, lung cancer A549 cell line, and myomelanocytic cancer B16 cell line.
As shown in Table 2, we clearly differentiate the endo/lysosomes in 6 cancer cell lines, with the accuracy at 0.01 pH values, which is impossible to be done with the commercial intraocular pH indicator kits.
Moreover, we re-evaluated pH values in endo/lysosomes of the HepG2 cells before and after cultured with BPSi nanoparticles. We reached the conclusion that BPSi uptake significantly increases the endo/lysosome pH values, from 4.70 ± 0.09 to 5.59 ± 0.05, perfectly illustrating the reason for BPsi uptaken induced the autophagy initially then terminated the autophagy flux. The intracellular uptake of BPSi makes the quantities of the endo/lysosomes increased, which was consistent with the results of gene sequencing, that autophagy-related genes (TFEB-CLEAR) were activated. Meanwhile, the autophagy termination by the increased pH values in endo/lysosomes also coincide with the results of p62 proteins upregulation in Western blot study.
Discusión
Nanoparticles can generally cause autophagy in cells [23], and studies have shown that the autophagic response to nanoparticles presenting a neutral or anionic surface involves enhanced clearance of autophagic cargo. Cell exposure to nanoparticles presenting a cationic surface, on the other hand, results in transcriptional upregulation of the TFEB pathway, but also causes lysosomal dysfunction, ultimately resulting in blockage of autophagic flux [7]. And our results are in consistent with these previous conclusions. In our study, we found that the expression of autophagy-related genes and proteins in HepG2 cells has been increased after feeding of BPSi nanoparticles through transcriptome sequencing, RT-qPCR, and Western experiments. However, the expression level of autophagy-related P62 protein does not decrease as the autophagy is activated. We suspect that the PEG-amine on the surface of BPSi nanoparticles raises the pH value of the lysosome, resulting in inhibition of P62 degradation. Existing lysosomal pH indicators cannot verify our guess. To accurately measure the lysosomal pH of living cells, we established a new method for endo/lysosomes pH qualitative determination based on self-decomposable nanoparticle systems. Ten nanoparticle systems with specific MB/TEOS parameters were employed for obtaining optimized pH sensitively responsive measurement method. The radial MB concentration gradient from inner out served as a major driving force for MB release. The drug release proceeded with simultaneously carrier decomposition, which was driven by a diffusion-controlled mechanism. Moreover, as the pH value decreases, the hydrogen ion concentration increases, and the enhanced electrostatic interaction promotes inner MB to release faster than in neutral solution [24]. The optimized central hollow nanoparticle system could release the central concentrated MB as a linear function of precise pH values in the range of pH 4.0–4.8, which is exactly the pH of lysosomes. Finally, by this qualitative pH indicator based on self-decomposable nanoparticles, we have succeeded in the detection of the average pH values of lysosomes in 6 cell lines. Moreover, by this system, we can qualitatively differentiate the pH changes of lysosomes before and after BPSi nanoparticle endocytosis by HepG-2 cells, clarifying the mechanism of the autophagy occurrence and then termination after BPSi endocytosis. The self-decomposable nanoparticle systems pave a brand new way for studying the luminal pH values, providing new tools to know better of the cell signaling and metabolism, and then providing new ways and methods for the treatment of cancer [25, 26].
Conclusión
In this study, we found that BPSi can promote cell autophagy through transcriptome sequencing, but the amino groups on the surface of the nanoparticles can increase the pH of the lysosome and inhibit the degradation of autophagic flow. Thus, the lysosome pH significantly influences the autophagy stages. And precisely acquiring the information of lysosome pH will promote the perceiving of autophagy. However, the existing fluorescent lysosomal pH indicators could only determine a wide range of lysosomal pH; thus, we established a precise lysosomal pH indicator based on the self-dissociation system. By adjusting the synthesis parameters of MB@SiO2 , the release of MB loaded on the nanoparticles was linearly and negatively correlated with pH. And the nanoparticles mainly stay in the lysosome after entering the cell. By measuring the amount of MB released in the cells, the pH value of the lysosome can be calculated exactly according to the linear function. The established precise pH indicator provided a brand new tool and methodology to precisely study the lysosome pH values and further acquire more information on autophagy.
Disponibilidad de datos y materiales
Todos los datos generados o analizados durante este estudio se incluyen en este artículo publicado y sus archivos de información complementaria.
Abreviaturas
- BPSi:
-
Black mesoporous silicon
- FBS:
-
Suero fetal bovino
- GO:
-
Gene ontology
- HCl:
-
Ácido clorhídrico
- KEGG:
-
Kyoto Encyclopedia of Genes and Genomes
- MB:
-
Azul de metileno
- NaBr:
-
Sodium bromide
- NaSi:
-
Sodium silicide
- NH4Br:
-
Ammonium bromide
- NP:
-
Nanopartículas
- OD:
-
Densidad óptica
- PBS:
-
Solución salina tamponada con fosfato
- PDMPO:
-
2-(4-Pyridyl)-5-((4-(2-dimethylaminoethy- laminocarbamoyl) methoxy) phenyl) oxazole
- RT-qPCR:
-
Reverse transcription quantitative polymerase chain reaction
- TEOS:
-
Ortosilicato de tetraetilo
Nanomateriales
- Monitoreo de aplicaciones SaaS:Ver el bosque y los árboles
- El desarrollo y la aplicación de la tela de fibra de vidrio
- Preparación de nanopartículas de mPEG-ICA cargadas con ICA y su aplicación en el tratamiento del daño celular H9c2 inducido por LPS
- Absorbedor perfecto de banda ultra estrecha y su aplicación como sensor plasmónico en la región visible
- La posible toxicidad hepática, cerebral y embrionaria de las nanopartículas de dióxido de titanio en ratones
- Formación y propiedades luminiscentes de Al2O3:nanocompuestos de SiOC en la base de nanopartículas de alúmina modificadas por feniltrimetoxisilano
- La preparación de la nanoestructura yema-cáscara de Au @ TiO2 y sus aplicaciones para la degradación y detección del azul de metileno
- Aptitud ecológica en la síntesis y estabilización de nanopartículas de cobre:actividades catalíticas, antibacterianas, citotóxicas y antioxidantes
- Estudios TEM y STEM sobre las morfologías transversales de películas antirreflectantes de SiO2 de banda ancha de doble / triple capa
- Saponinas Platycodon de Platycodi Radix (Platycodon grandiflorum) para la síntesis verde de nanopartículas de oro y plata
- Película de condensador:propiedades, construcción y aplicación



