Los tetraedros de ADN modificados con Gint4.T cargados con doxorrubicina inhiben la proliferación de células de glioma al dirigirse a PDGFRβ
Resumen
El glioma es uno de los tumores cerebrales intrínsecos más letales debido a su crecimiento invasivo. El efecto del tratamiento del glioma es deficiente debido a la presencia de la barrera hematoencefálica y la barrera hematoencefálica y la barrera contra los tumores sanguíneos y la focalización insuficiente del fármaco. Los tetraedros de ADN (TDN) muestran un gran potencial para la administración de fármacos y pueden ser una nueva estrategia terapéutica para el glioma. En este estudio, usamos TDN para administrar doxorrubicina (DOX) para la terapia del glioma. Se usó Gint4.T, un aptámero que podría reconocer el receptor β del factor de crecimiento derivado de plaquetas en la célula tumoral, para modificar TDN (Apt-TDN) para la administración de fármacos dirigida. Los TDN se autoensamblaron mediante síntesis en un solo paso, que mostró un tamaño pequeño (10 nm) y carga negativa. La prueba de suero bovino fetal mostró su estabilidad como vehículo de administración de fármacos. Apt-TDN podría ser absorbido eficazmente por células U87MG. En comparación con DOX y DOX @ TDN (TDN cargado con DOX), el DOX @ Apt-TDN (TDN modificado con Gint4.T cargado con DOX) mostró una tasa de apoptosis más temprana, una detención del ciclo celular más alta y una mayor citotoxicidad hacia las células U87MG. En conclusión, nuestros hallazgos indicaron que DOX @ Apt-TDN proporciona una terapia novedosa con una aplicación clínica prometedora para los pacientes con gliomas.
Introducción
El glioma, un tumor derivado del neuroepitelio, es la neoplasia intracraneal maligna más común. Casi 1/3 de todos los tumores cerebrales son gliomas y aproximadamente 4/5 de los tumores cerebrales malignos primarios son gliomas [1, 2, 3, 4]. Actualmente, el tratamiento más eficaz para el glioma es la resección quirúrgica y la quimiorradioterapia posoperatoria concurrente, pero, lamentablemente, el pronóstico para los pacientes sigue siendo malo. La quimioterapia tradicional para el glioma no muestra un buen resultado debido a la focalización deficiente del tumor, las complicaciones debidas a la barrera hematoencefálica (BBB) y la barrera tumoral sanguínea (BTB) y la focalización insuficiente del fármaco. La BBB es el factor más importante que previene la entrega de casi todas las macromoléculas (incluidos fármacos y genes) al parénquima cerebral y su función adecuada. Los fármacos que no pueden atravesar la BHE deben administrarse en dosis suficientemente altas para lograr una concentración de tratamiento eficaz en el área de interés. Sin embargo, el exceso de fármacos puede provocar efectos secundarios sistémicos graves y una acumulación de fármaco no deseada en los tejidos no afectados. Además, los fármacos antiglioma convencionales existentes tienen una capacidad de focalización insuficiente [3, 4]. Las nanopartículas se han convertido en la herramienta portadora de fármacos más prometedora. Debido a su ventaja de tamaño, las nanopartículas pueden atravesar la BBB y ejercer un efecto antitumoral. Kafa y col. [5] diseñaron nanotubos de carbono de paredes múltiples funcionalizados químicamente (f-MWNT) dirigidos a ANG y confirmaron su capacidad para cruzar la BBB a través de experimentos in vivo e in vitro. Sin embargo, estos nanomateriales pueden distribuirse a varios órganos de todo el cuerpo o incluso ingresar al sistema nervioso central (SNC), donde pueden causar neurotoxicidad [6].
El ADN es un material ideal para la construcción de nanoestructuras porque su ensamblaje se puede controlar con precisión mediante el emparejamiento de bases Watson-Crick [7]. Hasta la fecha, se han diseñado y demostrado una serie de nanoestructuras de ADN bidimensionales (2D) y tridimensionales (3D) [8,9,10]. Las nanoestructuras tetraédricas de ADN (TDN) han atraído una atención significativa debido a su biocompatibilidad, estabilidad, abundantes sitios de modificación funcionalizada y baja inmunogenicidad [11,12,13]. Turberfield y col. nanoestructuras tetraédricas de ADN sintetizado con un alto rendimiento utilizando un método de síntesis de un solo paso [14]. Walsh y col. encontraron que un tetraedro de ADN que contiene una sonda de ácido nucleico podría ingresar a las células de mamíferos sin la necesidad de un reactivo de transfección [15]. Lee y col. demostraron que las nanopartículas tetraédricas autoensambladas se pueden utilizar para la entrega de ARNip dirigida in vivo [16]. TDN ha mostrado excelentes perspectivas de aplicación en diagnóstico molecular, administración molecular y farmacoterapia dirigida. También se utilizan ampliamente en la investigación de tumores en varios órganos, como el cáncer de mama [17]. De manera similar, TDN también se ha utilizado en el estudio de enfermedades del sistema nervioso. Tian y col. [18] utilizó tetraedros de ADN como base y los modificó con angiopep-2 (ANG) para construir con éxito la nanoonda ANG-TDN, que podría apuntar a la proteína 1 relacionada con el receptor de lipoproteínas de baja densidad (LRP-1) para obtener imágenes específicas. La investigación ha demostrado que ANG-TDN puede cruzar la BBB. Ma et al. [19] sugirió que las nanoestructuras de ADN tetraédrico pueden ingresar a las células madre neurales (NSC) sin la necesidad de agentes de transfección, donde promueven la migración, proliferación y diferenciación de las células madre neurales [20], y tienen un gran potencial para la reparación y regeneración del tejido neural . Por lo tanto, investigamos la posibilidad del uso de TDN en la terapia del glioma y la mejora de su capacidad de focalización.
El receptor β del factor de crecimiento derivado de plaquetas (PDGFRβ) es un miembro importante de la familia de las tirosina proteína quinasa que participa en la proliferación celular, la migración y la angiogénesis. Varios estudios han demostrado que el PDGFβ es un objetivo prometedor para las terapias antitumorales debido a su papel en la angiogénesis [21, 22]. Los aptámeros son oligonucleótidos cortos de ADN o ARN monocatenarios que se producen mediante la evolución sistemática de ligandos mediante el método de enriquecimiento exponencial (SELEX). Los aptámeros son similares a los anticuerpos, que tienen una alta afinidad y especificidad hacia sus objetivos [23]. Debido a sus propiedades únicas, los aptámeros desempeñan un papel importante en la administración dirigida de agentes quimioterapéuticos, ARNip y nanopartículas cargadas con fármacos. Gint4.T, un aptámero de ARN que puede unirse específicamente a PDGFRβ, también es un antagonista específico de PDGFRβ [24]. Monaco et al. [25] sugirió que los aptámeros de Gint4.T pueden cruzar la barrera hematoencefálica (BBB) y reconocer específicamente PDGFRβ. Las nanopartículas poliméricas conjugadas con Gint4.T (PNP) pueden ser absorbidas fácilmente por las células de glioblastoma (GBM). En este estudio, presentamos un nuevo sistema cargado de fármacos que combina el aptámero Gint4.T y TDN. El TDN modificado con Gint4.T (Apt-TDN) cargado con DOX (DOX @ Apt-TDN) exhibió una captación celular específica mejorada y citotoxicidad contra las células U87MG.
Métodos
Materiales
Todos los oligonucleótidos de ADN y oligonucleótidos de ARN 2'F-Py se adquirieron de Sangon Biotech (Shanghai, China), y todas las secuencias de oligonucleótidos se enumeran en la Tabla 1. La solución de tinción de gel de ADN GelRed se adquirió de Sangon Biotech. Tanto el suero fetal bovino (FBS) como el medio Eagle modificado de Dulbecco (DMEM) se adquirieron de Thermo Fisher (Nueva York, EE. UU.). La doxorrubicina (DOX) se adquirió de Mengbio Technology (Chongqing, China). Las células U87MG se adquirieron en la Biblioteca de Células de la Academia de Ciencias de la Vida de Shanghai (Shanghai, China). DAPI se compró a Zhongshan Golden Bridge Biotechnology (Beijing, China).
Preparación de nanoestructuras de ADN
Para ensamblar el ADN tetraedro (Tabla 1), se agregaron 2 μL de cada oligonucleótido (S1, S2, S3 y S4) a 42 μL de tampón TM (Tris-HCl 10 mM, MgCl 2 , pH =8). A continuación, la solución de ADN se calentó a 95 ° C durante 5 min y posteriormente se enfrió a 4 ° C durante 2 min utilizando una máquina de PCR Bio-Rad (California, EE. UU.) [26, 27]. La concentración final de TDN fue de 2 µM. TDN 'se preparó de la misma manera, excepto que S1 se reemplazó por S1'. Para sintetizar el Apt-TDN, se añadió el aptámero Gint4.T a TDN en una proporción molar igual y la mezcla se incubó a 37 ° C durante 60 min. Antes de la síntesis, el aptámero se sometió a un breve paso de desnaturalización-renaturalización (85 ° C durante 5 min, enfriamiento rápido durante 2 min y posteriormente calentado a 37 ° C durante 10 min) [25].
Electroforesis en gel de agarosa
Se corrió un gel de agarosa (3%) en tampón TEB 0,5 x a 100 V durante 30 min. La temperatura del aparato de electroforesis se mantuvo a 0 ° C colocando el aparato en un baño de hielo. Antes de la electroforesis, se añadió GelRed al gel de agarosa para teñir las hebras de ADN. Cuando finalizó el proceso, se utilizó un escáner de fluorescencia Bio-Rad (California, EE. UU.) Para capturar una imagen del gel.
Dispersión de luz dinámica
Se utilizó un Malvern Zetasizer ZS90 (Malvern, Reino Unido) para medir el tamaño hidrodinámico y el potencial zeta de la TDN. Se sometió un total de 1 ml de TDN (100 nM) a análisis de dispersión dinámica de luz (DLS).
Imágenes de microscopía de fuerza atómica
Los TDN se diluyeron a 100 nM con tampón TM (tampón Tris-HCl que contiene MgCl 2 ). Luego, se agregaron 10 μl de cada muestra de TDN a mica recién escindida y se incubó durante 10 min. Posteriormente, se obtuvieron imágenes de las muestras en un instrumento de microscopía de fuerza atómica (AFM) en modo AC (Agilent 5500, EE. UU.).
Medición de la capacidad de carga de fármaco del TDN preparado
La doxorrubicina se disolvió en agua desionizada para preparar una solución de almacenamiento de 500 µM. Se mezcló doxorrubicina a diferentes concentraciones (1 a 20 μM) con TDN (100 nM) o Apt-TDN (100 nM) durante 6 horas a temperatura ambiente (24-26 ° C). A continuación, las soluciones mezcladas se centrifugaron a 12.000 x g durante 10 min para obtener la TDN cargada con fármaco. Luego, se retiraron 50 μL de los sobrenadantes y se mezclaron con PBS en una proporción de 1:1. Se utilizó un lector de microplacas Varioskan LUX (California, EE. UU.) Para medir la intensidad de fluorescencia de la doxorrubicina ( λ ex =480 nm y λ em =590 nm) para determinar la cantidad de doxorrubicina en los sobrenadantes [28]. La concentración de doxorrubicina cargada en el TDN se calculó mediante la curva estándar y la intensidad de fluorescencia. También mezclamos doxorrubicina con TDN en proporciones molares crecientes.
Estabilidad sérica de TDN in vitro
Los TDN se mezclaron con medio completo y se incubaron a 37 ° C durante 0, 2, 4, 6, 8, 10, 12 o 24 h. Las soluciones de TDN se mezclaron con FBS en una proporción de 1:1 y se incubaron a 37 ° C durante 1, 3, 5 o 7 h. Después de la incubación, las mezclas se procesaron en un gel de agarosa al 3%.
Citotoxicidad de la TDN in vitro
Para determinar la citotoxicidad de las células TDN, U87MG a una concentración de 1 × 10 4 células / pocillo se sembraron en una placa de 96 pocillos. Se retiró el medio de cultivo celular y se agregaron medios frescos que contenían TDN 0-500 nM y se incubaron durante otras 24 hy 48 h después de la incubación durante la noche. Luego, se agregaron 10 μL de solución de CCK-8 a cada pocillo y la mezcla se incubó durante 1 h. Luego se midió la absorbancia a 450 nm usando un lector de microplacas.
Imágenes de fluorescencia
La captación celular de DOX y TDN se estudió mediante microscopía de fluorescencia (Olympus, Tokio, Japón). Se sembraron células U87MG en cubreobjetos en placas de 24 pocillos con medio que contenía suero bovino fetal inactivado por calor al 10% y penicilina y estreptomicina al 1% y se cultivaron durante al menos 1 día a 37 ° C en una atmósfera humidificada con 5% de CO 2 hasta que las células alcanzaron al menos el 75% de confluencia. Después de la incubación, se retiraron los medios de cultivo. Se añadieron medios completos que contenían Cy3-TDN y Cy3-Apt-TDN 100 nM y se incubaron durante 3 h. TDN y Apt-TDN se marcaron con Cy3 para detectar la captación intercelular de nanopartículas. Para evaluar la captación celular de DOX, se agregaron DOX (DOX 2 μM), DOX @ TDN (DOX 2 μM) y DOX @ Apt-TDN (DOX 2 μM) a las células U87MG y se incubaron durante 3 h. Después de 3 h de tratamiento, las células se fijaron con paraformaldehído al 4% durante 20 min en la oscuridad y posteriormente se tiñeron con 4 ', 6-diamidino-2-fenilindol (DAPI) durante 5 min. Las células se lavaron con PBS tres veces y se observaron bajo un microscopio de fluorescencia.
Citometría de flujo
Un total de 1 × 10 6 Se implantaron células U87MG en placas de 6 pocillos. Después de la incubación durante la noche, se retiraron los medios de cultivo y se añadieron medios suplementados con Cy3-TDN 100 nM, Cy3-Apt-TDN 100 nM o Cy3-Apt-TDN 100 nM + Apt libre 1 μM y se incubaron durante 3 h. Luego, las células se fijaron con paraformaldehído al 4% durante 20 min y se utilizó citometría de flujo para analizar los porcentajes de células positivas para Cy-3.
Ciclo celular y apoptosis
Después del tratamiento con DOX, DOX @ TDN o DOX @ Apt-TDN durante 24 h, 5 × 10 5 las células se recogieron y fijaron en etanol helado al 75% durante la noche. Luego, las células se incubaron con RNasa y yoduro de propidio durante 30 min a 37 ° C en la oscuridad. El ciclo celular se investigó mediante citometría de flujo. Además, después de los diferentes tratamientos, las células se tiñeron con Anexina V-FITC / DAPI y se exploró la apoptosis temprana.
Ensayos CCK-8
Para determinar la viabilidad celular, las células U87MG (5 × 10 3 ) se sembraron en placas de 96 pocillos con 100 μL de medio y se cultivaron durante la noche a 37 ° C en una atmósfera que contenía un 5% de CO 2 . Posteriormente se retiró el medio y se añadió medio fresco que contenía DOX, DOX-TDN o DOX-Apt-TDN. Después de 24 h de incubación, se agregaron 10 μL de solución de CCK-8 y las células se cultivaron durante 1 h más. Se utilizó un lector de microplacas para medir la absorbancia a 450 nm.
Análisis estadístico
Todos los experimentos de este estudio se realizaron por triplicado y todos los datos se presentan como el valor medio con su desviación estándar (media ± DE). El análisis estadístico se realizó mediante el programa SPSS 24.0 (IBM, EE. UU.). Las diferencias significativas se determinaron utilizando la t de Student prueba, con P <0.05 indica diferencias significativas entre grupos.
Resultados
Síntesis y caracterización de TDN y Apt-TDN
El TDN fue autoensamblado a partir de cuatro oligonucleótidos (Tabla 1) mediante síntesis en un solo paso, como se informó anteriormente [18, 29]. El aptámero dirigido a tumores Gint4.T se utilizó para modificar TDN mediante el emparejamiento de bases Watson-Crick. El tetraedro de ADN contiene cuatro caras, cada una de las cuales está formada por un oligonucleótido. Por tanto, cuatro oligonucleótidos se hibridaron entre sí para formar un tetraedro de ADN (Fig. 1a). El análisis de electroforesis en gel mostró una única banda prominente en los carriles 4 y 5, lo que sugiere que TDN y Apt-TDN se construyeron con éxito. La movilidad del Apt-TDN se redujo en comparación con la del TDN, lo que sugiere que el aptámero Gint4.T modificó con éxito el TDN.
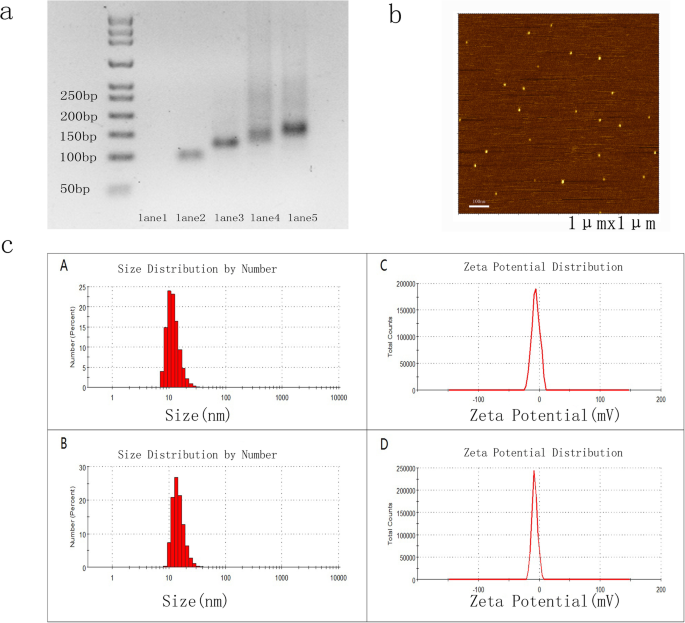
un Síntesis del ADN tetraedro y Gint4.T-TDN. Carril 1:S1; carril 2:S1 + S2; carril 3:S1 + S2 + S3; carril 4:S1 + S2 + S3 + S4 (TDN); carril 5:TDN mezclado con Apt-tail (Gint4.T); Apt-TDN. El carril 1 no era visible porque los colorantes de ácido nucleico no pueden teñir adecuadamente el ADN monocatenario. b Las imágenes AFM mostraron que las alturas de TDN y Apt-TDN eran ~ 2 nm. c Determinación del tamaño de partícula y potencial zeta de TDN y Apt-TDN por dispersión dinámica de luz (DLS). Los tamaños de partícula promedio de TDN y Apt-TDN fueron 10,10 nm (A) y 13,54 nm (B), respectivamente. Los potenciales zeta promedio de TDN y Apt-TDN fueron - 5,69 mV (C) y - 7,3 mV (D), respectivamente
Los tamaños de TDN y Apt-TDN fueron determinados por DLS y AFM. TDN y Apt-TDN mostraron tamaños de partículas de 10,1 nm y 13,5 nm, respectivamente, lo que refleja la adición del ligando Gint4.T (Fig. 1c (A), (B)). Debido a que el diámetro hidrodinámico incluye moléculas de agua, las partículas eran más grandes que sus tamaños teóricos. Las alturas de TDN y Apt-TDN determinadas por imágenes AFM fueron ~ 2 nm (Fig. 1b), lo que indica que la modificación del aptámero no cambió la estructura 3D. Los potenciales zeta promedio de TDN y Apt-TDN fueron -5,69 mV (C) y -7,3 mV (D), respectivamente (Fig. 1c (C) (D)). Basándonos en estos parámetros, llegamos a la conclusión de que TDN y Apt-TDN se habían ensamblado con éxito.
Estabilidad y citotoxicidad de la TDN in vitro
El análisis de electroforesis en gel mostró que el TDN permaneció intacto cuando se incubó en medio completo durante 24 ha 37 ° C (Fig. 2a (A)). Además, cuando la concentración de suero bovino fetal aumentó al 50%, la TDN permaneció estable durante al menos 7 h (Fig. 2a (B)), lo que coincide con informes anteriores [18, 26]. Para determinar la citotoxicidad de la nanoestructura, se utilizó el ensayo CCK-8 para evaluar la viabilidad celular de las células U87MG después del tratamiento con TDN a varias concentraciones. Como se muestra en la Fig. 2b, no se observó citotoxicidad significativa en las células U87MG tratadas con TDN a 0-500 nM durante 24 hy 48 h. Por lo tanto, las nanopartículas de ADN se pueden utilizar como vehículos estables y bioseguros para la administración de fármacos.
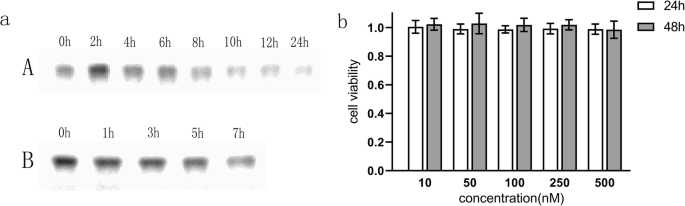
un La electroforesis en gel mostró que el TDN permaneció estable durante 24 h en medio completo a 37 ° C (A); el TDN permaneció estable durante 7 h cuando la concentración de suero bovino fetal se incrementó al 50% (B). b Se cocultivaron células U87MG con TDN a diferentes concentraciones (10-500 nM) durante 24 hy 48 h. El ensayo CCK-8 mostró que la actividad de las células U87MG no se vio afectada, lo que indicó la bioseguridad de la TDN
Capacidad de carga de fármacos de TDN y Apt-TDN
La doxorrubicina es un fármaco quimioterapéutico de amplio espectro que puede intercalarse en cadenas dobles de ADN. Calculamos una curva estándar de doxorrubicina (Fig. 3a) y luego investigamos la intercalación de la doxorrubicina en la TDN. La cantidad de doxorrubicina intercalada en TDN y Apt-TDN aumentó gradualmente al aumentar la concentración de doxorrubicina. Cuando la concentración de doxorrubicina fue de 14 μM, la cantidad de doxorrubicina intercalada en el TDN y Apt-TDN alcanzó un máximo de 5,5 μM y 6,0 μM, respectivamente, y posteriormente se estabilizó (Fig. 3b), lo que indica que las hebras de ADN estaban completamente ocupadas. Mientras tanto, mezclamos doxorrubicina con TDN en proporciones molares crecientes. Se escaneó el espectro de fluorescencia de la doxorrubicina para su análisis. Como se muestra en la Fig. 3c, el espectro de fluorescencia de la doxorrubicina se inactivó con doxorrubicina en una relación molar de 0,05. Con base en estos hallazgos, llegamos a la conclusión de que aproximadamente 55 moléculas de doxorrubicina estaban contenidas dentro de un solo TDN, mientras que 60 moléculas estaban contenidas dentro de un solo Apt-TDN.
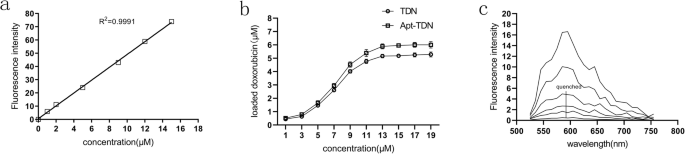
un Una curva estándar de concentraciones de DOX en tampón PBS; λex =480 nm y λem =590 nm. La cantidad de DOX transportada por TDN y Apt-TDN. b DOX intercalado en el ADN de doble hebra de TDN y Apt-TDN. Cuando la concentración de DOX alcanzó 14 μM y la concentración de DOX intercalada en TDN y Apt-TDN alcanzó un máximo de 5,5 μM y 6,0 μM, respectivamente, un solo tetraedro de ADN podría transportar 55 moléculas de Dox, mientras que un solo tetraedro de ADN modificado por aptámero transportó 60 DOX moléculas. c Espectros de fluorescencia de DOX en el sobrenadante. Se mezcló doxorrubicina con el TDN en proporciones molares crecientes (0, 0,0005, 0,001, 0,005, 0,01 y 0,05 de arriba a abajo). Cuando la relación molar era 1:20, la fluorescencia se apagó
Captación celular dirigida de Apt-TDN
El ADN es una macromolécula cargada negativamente, lo que dificulta su entrada en la membrana celular cargada negativamente. Normalmente, las moléculas de ADN individuales deben acceder a las células con la ayuda del reactivo de transfección. Aquí, etiquetamos TDN y Apt-TDN con Cy3 para monitorear la captación intracelular de las nanopartículas. Después de la incubación con células U87MG durante 3 h, la señal de fluorescencia roja Cy3 emergió en el citoplasma celular, lo que indica que el TDN se unió a la citomembrana y se absorbió en la célula sin la ayuda de agentes de transfección (Fig. 4a). El Apt-TDN mostró una mayor fluorescencia roja, lo que sugiere que la presencia del aptámero Gint4.T aumentó significativamente la captación del tetraedro de ADN por las células U87MG. Sin embargo, cuando se añadió aptámero libre, la fluorescencia de Cy3 disminuyó hasta el nivel observado para el TDN solo. Debido a la inhibición competitiva por parte del aptámero libre, inferimos que el aptámero en la TDN no podría facilitar la captación de la TDN. Basándonos en esta inhibición competitiva, demostramos que el Apt-TDN puede dirigirse a las células U87MG. La citometría de flujo demostró además que el porcentaje de células U87MG positivas para Cy3 era mayor en el grupo Apt-TDN que en el grupo TDN. Free Apt disminuyó el porcentaje de células U87MG positivas para Cy3 en el grupo Apt-TDN (Fig. 4b).
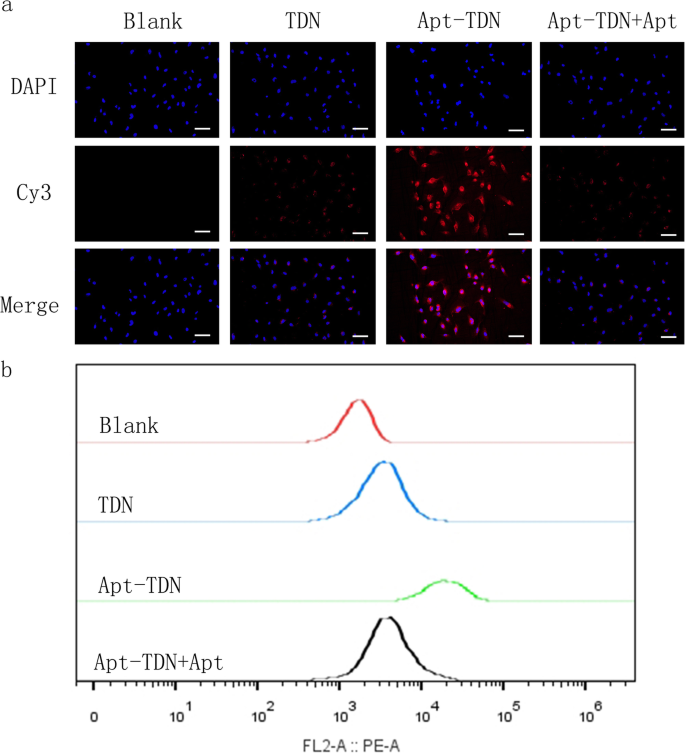
un Captación de células U87MG de TDN y Apt-TDN (TDN-Gint4.T). El TDN entró en las células U87MG directamente sin agentes de transfección, y la captación de Apt-TDN (ligada al aptámero Gint4.T) aumentó significativamente y fue inhibida competitivamente por el Apt libre (Gint4.T), lo que indica que el aptámero Gint4.T juega un papel importante. papel importante en la focalización celular. La barra de escala representa 50 μm. b Las curvas de citometría de flujo muestran la captación intracelular de TDN, Apt-TDN y Apt-TDN + Apt después de la incubación durante 3 h
Captación celular de DOX @ TDN y DOX @ Apt-TDN
Utilizamos el espectro de fluorescencia característico de la doxorrubicina para evaluar la eficiencia de absorción del fármaco. Después de 3 h de tratamiento, se obtuvieron imágenes de doxorrubicina intracelular mediante microscopía de fluorescencia (Fig. 5a). La doxorrubicina libre podía entrar en las células U87MG y estaba ubicada en el núcleo. Con la adición de DOX @ TDN, la fluorescencia fue mayor que la de la doxorrubicina libre. Este resultado sugirió que las nanopartículas de ADN mejoraron la captación celular de doxorrubicina. Cuando se agregó DOX @ Apt-TDN, la señal roja en el núcleo era incluso más alta que la de las células a las que se había agregado DOX @ TDN. El análisis semicuantitativo de la captación intracelular de DOX confirmó además que el Apt-TDN facilitó un aumento de más del doble en la captación intracelular de DOX en comparación con el del fármaco único. Inferimos que esto se debe a la unión específica de Gin4.T a los receptores, después de lo cual pueden ingresar más nanopartículas a la célula. Después de la digestión en lisosomas, la doxorrubicina puede liberarse en el citoplasma y realizar otras funciones en el núcleo.
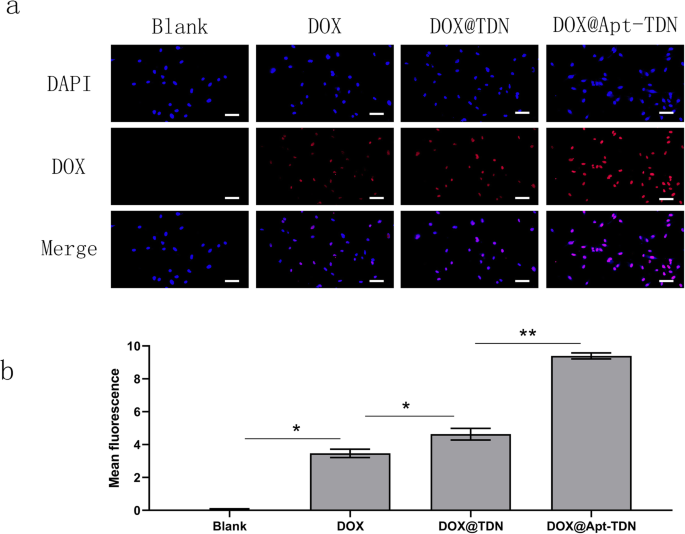
un Captación celular de DOX, DOX @ TDN y DOX @ Apt-TDN. Modificado con el aptámero Gint4.T, el Apt-TDN podría administrar más doxorrubicina a las células U87MG que el TDN. Además, el TDN podría transportar más fármaco a las células que el fármaco solo. Las barras de escala indican 50 μm. b Análisis semicuantitativo de la intensidad de fluorescencia de la doxorrubicina con tratamiento con PBS, DOX, DOX @ TDN y DOX @ Apt-TDN (en comparación con el blanco:* p <0.05, ** p <0.01)
Citotoxicidad de DOX, DOX @ TDN y DOX @ Apt-TDN
Para el estudio de citotoxicidad, se trataron tres grupos de células U87MG con diferentes concentraciones de doxorrubicina (Fig. 6a). El IC 50 los valores de doxorrubicina fueron 13,39 μM para el tratamiento con DOX, 7,826 μM para el tratamiento con DOX @ TDN y 4,205 μM para el tratamiento con DOX @ Apt-TDN. Entre los tres tratamientos, el DOX @ Apt-TDN mostró la mayor citotoxicidad a las 24 h, lo que indicó la especificidad del Apt-TDN para las células U87MG. Después de 24 h, las células de los grupos DOX, DOX @ TDN y DOX @ Apt-TDN se recolectaron y usaron para explorar la apoptosis temprana. Nuestros datos demostraron que la tasa de apoptosis temprana fue mayor en el grupo DOX @ Apt-TDN que en los otros dos grupos (Fig. 6b). Además, la proporción de células en la fase G0 / G1 aumentó en el grupo DOX-Apt-TDN en comparación con los grupos DOX y DOX-TDN ( p <0.01), y la proporción de células en la fase S disminuyó en el grupo DOX-Apt-TDN ( p <0,01) (figura 6c). El porcentaje de células en la fase G2 no se modificó (no se muestran los datos).
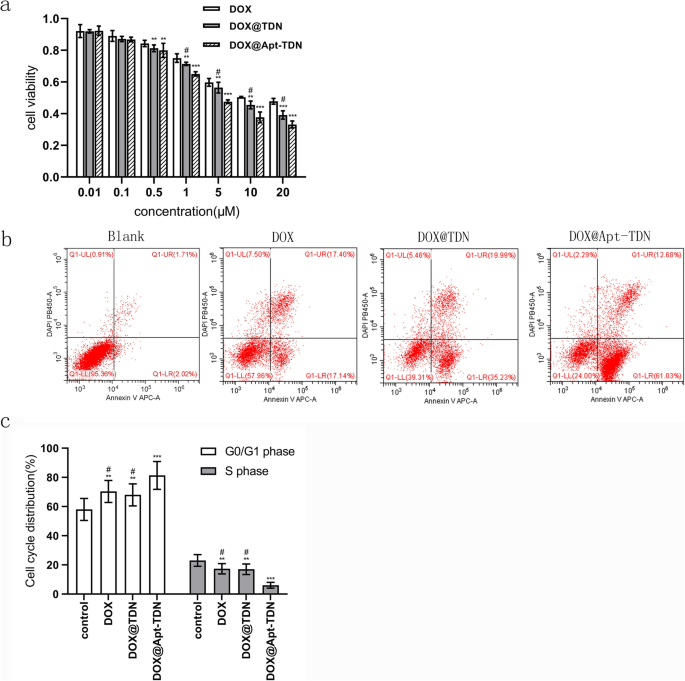
un Citotoxicidad de DOX, DOX @ TDN y DOX @ Apt-TDN a diversas concentraciones. La tasa de inhibición de las células U87MG aumentó significativamente con el aumento de la concentración de DOX, pero los grupos DOX @ TDN y DOX @ Apt-TDN exhibieron una citotoxicidad significativamente mayor en comparación con el grupo DOX. La tasa de inhibición celular del grupo DOX @ Apt-TDN también fue significativamente mayor que la del grupo DOX @ TDN (en comparación con DOX, ** p < 0,05; en comparación con DOX, *** p <0,01; en comparación con DOX @ Apt-TDN, # p <0,05). b Apoptosis de células U87MG después de incubación con PBS, DOX, DOX @ TDN y DOX @ Apt-TDN durante 24 h. c Histogramas de citometría de flujo del ciclo celular U87MG después de la incubación con PBS, DOX, DOX @ TDN y DOX @ Apt-TDN durante 24 h (en comparación con el control, ** p < 0,05; en comparación con el control, *** p <0,01; en comparación con DOX @ Apt-TDN, # p <0.01)
Discusión
Ya sea mediante experimentos in vitro o in vivo, se debe determinar la estabilidad de un fármaco y su portador. Después del ensamblaje del TDN, se determinó primero su estabilidad in vitro. Este estudio demostró que la estructura 3D de las nanopartículas de ADN puede mejorar su estabilidad en el suero al inhibir la unión de enzimas. La seguridad biológica de la nanoestructura relevante es el requisito más importante para su aplicación. No se observó citotoxicidad significativa en células U87MG cocultivadas con diversas concentraciones de TDN durante 24 ho 48 h. Ninguna de las secuencias de ADN utilizadas en este estudio codifica información genética y no se informaron efectos secundarios en la prueba de citotoxicidad. Por lo tanto, la TDN puede servir como un portador de medicamentos seguro y estable.
La eficacia de focalización de una nanoestructura modificada por aptámeros es crucial para la administración selectiva de fármacos a las células cancerosas. A diferencia de los anticuerpos, los aptámeros son químicamente estables, económicos y pueden producirse en masa. Además, a diferencia de otros materiales, los aptámeros pueden unirse fácilmente a un tetraedro de ADN utilizando el principio de complementariedad de bases. Por lo tanto, la combinación de aptámeros y tetraedros de ADN sienta las bases para la administración de fármacos dirigida y los tratamientos de próxima generación. Nuestros resultados mostraron que el TDN podría ingresar a las células sin agentes de transfección, lo que es similar a los resultados de Walsh et al. y Ma et al. [15, 19]. En comparación con la de TDN, la captación de Apt-TDN por las células U87MG aumentó significativamente. Después de la adición de aptámero libre, este aumento desapareció. Este resultado sugiere que Gint4.T es un antagonista específico de PDGFRβ [25]. Nuestro estudio demostró que el aptámero Gint4.T podría apuntar a las células U87MG debido a la alta expresión de PDGGRβ en la superficie de las células de glioma. Camorani y col. [24] también mostró que el aptámero Gint4.T podría apuntar a las células tumorales interactuando específicamente con el dominio extracelular de PDGFRβ. Las ventajas de la selección de aptámeros Gint4.T se han demostrado hasta cierto punto a través de estudios in vitro, pero se requieren más pruebas in vivo para confirmar estos hallazgos. Este estudio también confirmó que DOX @ Apt-TDN es más citotóxico que DOX o DOX @ TDN, lo que probablemente se origina a partir de dos factores. En primer lugar, el aptámero Gint4.T puede unirse específicamente a dominios extracelulares de PDGFR, bloqueando la proliferación de células tumorales e inhibiendo el crecimiento de las células tumorales [24]. Esto es consistente con nuestros resultados experimentales. En comparación con el grupo de control, el ciclo celular U87MG cambia después del tratamiento con DOX, DOX @ TDN y DOX @ Apt-TDN aumentó significativamente en las células tumorales de la fase G0 / G1, disminuyó en las células de la fase S y bloqueó las células tumorales en G0. / Fase G1, que indica que pueden inhibir el ciclo celular de las células U87MG e inhibir su proliferación. En comparación con los grupos DOX y DOX @ TDN, el grupo DOX @ Apt-TDN tenía una capacidad significativamente mayor para inhibir la proliferación de células U87MG. Además, Gint4.T se une específicamente a las células tumorales y mejora la orientación celular del complejo DOX @ Apt-TDN. Por lo tanto, podemos aumentar la eficacia de la focalización del fármaco y reducir la dosis de administración sistémica de fármacos antitumorales para prevenir sus efectos secundarios sistémicos.
Conclusiones
La modificación con Gint4.T aumentó la especificidad y la eficiencia de TDN en el tratamiento del glioma al dirigirse al PDGFRβ que se expresa en grandes cantidades en las células del glioma. Además, cuando se carga con Apt-TDN, podría promover significativamente los efectos anti-glioma de DOX. Por lo tanto, DOX @ Apt-TDN puede servir como una estrategia terapéutica prometedora contra el glioma para los pacientes. La deficiencia de este estudio es que solo se ha verificado in vitro. En el estudio posterior, exploraremos más en modelos animales.
Disponibilidad de datos y materiales
Los datos relevantes se incluyen en el artículo.
Abreviaturas
- BBB:
-
Barrera hematoencefálica
- BTB:
-
Barrera contra tumores sanguíneos
- TDN:
-
Nanoestructuras de ADN tetraédrico / tetraedros de ADN
- DOX:
-
Doxorrubicina
- PDGFRβ:
-
Receptor β del factor de crecimiento derivado de plaquetas
- Apt-TDN:
-
TDN modificado por Gint4.T
- DOX @ TDN:
-
TDN cargado con DOX
- DOX @ Apt-TDN:
-
TDN modificado con Gint4.T cargado con DOX
- CNS:
-
Sistema nervioso central
- ANG:
-
Angiopep-2
- LRP-1:
-
Low-density lipoprotein receptor-related protein 1
- SELEX:
-
Exponential enrichment
- PNPs:
-
Polymeric nanoparticles
- GBM:
-
Glioblastoma
- FBS:
-
Foetal bovine serum
- DMEM:
-
Medio Eagle modificado de Dulbecco
- DLS:
-
Dispersión de luz dinámica
- AFM:
-
Microscopía de fuerza atómica
- CCK-8:
-
Cell Counting Kit-8
- Cy3:
-
Sulfo-Cyanine3
- DAPI:
-
4′,6-Diamidino-2-phenylindole
Nanomateriales
- Optimización de la eficiencia de transferencia con una celda robótica
- Dirigirse a las células endoteliales con nanopartículas multifuncionales de GaN / Fe
- Biosensor ultrasensible para la detección de ADN de Vibrio cholerae con nanoesferas compuestas de poliestireno-co-ácido acrílico
- Promoción del crecimiento celular SH-SY5Y mediante nanopartículas de oro modificadas con 6-mercaptopurina y un péptido penetrante de neuronas
- Una investigación sobre una célula solar de silicio cristalino con una capa de silicio negro en la parte posterior
- Nanocomposites a base de óxido de grafeno decorados con nanopartículas de plata como agente antibacteriano
- Fabricación de una célula solar de silicio monocristalino eficiente al 20,19% con microestructura piramidal invertida
- Efectos del fullereno C60 sobre la interacción del difenil-N- (tricloroacetil) amidofosfato con el ADN in silico y su actividad citotóxica contra la línea celular leucémica humana in vitro
- Propiedades fotovoltaicas mejoradas en la celda solar de heterounión plana Sb2S3 con un enfoque de selección rápida
- El efecto de apoptosis en las células de cáncer de hígado de nanopartículas de oro modificadas con ácido litocólico
- Optimización de su aplicación de soldadura con una celda de soldadura RW950 personalizada



