Análisis de actina y organización de la adhesión focal en células U2OS en nanoestructuras poliméricas
Resumen
Antecedentes
En este trabajo, exploramos cómo las células U2OS se ven afectadas por matrices de nanopilares de polímero fabricados en superficies de vidrio planas. Nos centramos en describir los cambios en la organización del citoesqueleto de actina y en la ubicación, el número y la forma de las adherencias focales. A partir de nuestros hallazgos, identificamos que las células se pueden clasificar en diferentes regímenes en función de su comportamiento de propagación y adhesión en nanopilares. Un análisis cuantitativo sugiere que las células sembradas en matrices de nanopilares densos están suspendidas en la parte superior de los pilares con adherencias focales que se forman más cerca de la periferia celular en comparación con superficies planas o matrices de pilares dispersos. Este cambio es análogo a respuestas similares para células sembradas en sustratos blandos.
Resultados
En este trabajo, exploramos cómo las células U2OS se ven afectadas por matrices de nanopilares de polímero fabricados en superficies de vidrio planas. Nos centramos en describir los cambios en la organización del citoesqueleto de actina y en la ubicación, el número y la forma de las adherencias focales. A partir de nuestros hallazgos, identificamos que las células se pueden clasificar en diferentes regímenes en función de su comportamiento de propagación y adhesión en nanopilares. Un análisis cuantitativo sugiere que las células sembradas en matrices de nanopilares densos están suspendidas en la parte superior de los pilares con adherencias focales que se forman más cerca de la periferia celular en comparación con superficies planas o matrices de pilares dispersos. Este cambio es análogo a respuestas similares para células sembradas en sustratos blandos.
Conclusión
En general, mostramos que la combinación de nanofabricación de alto rendimiento, microscopía óptica avanzada, herramientas de biología molecular para visualizar procesos celulares y análisis de datos se puede utilizar para investigar cómo las células interactúan con superficies nanoestructuradas y ayudará en el futuro a crear sustratos de cultivo que inducen función celular.
Resumen gráfico
Antecedentes
In vivo, las células normalmente residen en un entorno 3D complejo llamado matriz extracelular (ECM). El ECM no solo sirve como un andamio estructural para las células, también es un transportador de señales biomecánicas y bioquímicas y, por lo tanto, regula una variedad de procesos como la morfogénesis tisular, la homeostatis y la diferenciación. Está compuesto de agua, polisacáridos y proteínas [1, 2, 3, 4] y la composición varía entre los tipos de tejido.
Motivados por la necesidad de crear modelos de cultivo celular que representen mejor las condiciones in vivo, los investigadores han comenzado cada vez más a estudiar el comportamiento celular también en matrices 3D y en sistemas “semi-3D”. Se han identificado una serie de diferencias en los fenotipos celulares entre sustratos planos y sistemas con mayor dimensionalidad [5, 6]. Por ejemplo, se sabe que características como la viabilidad, la proliferación, la diferenciación y la morfología difieren entre las células en superficies planas y las células incrustadas en matrices 3D [3, 7].
Los sustratos de tipo in vivo varían desde sustratos “semi-3D” /2.5D, como superficies planas decoradas con diversas nanoestructuras hasta sistemas “3D real”, como geles de colágeno o matrices de matrigel [8,9,10,11]. Además, el posicionamiento controlado de los ligandos en las superficies puede proporcionar nuevos conocimientos sobre cómo las células interactúan con varios patrones químicos [12, 13, 14]. También se ha demostrado que factores mecánicos como la rigidez de la estructura o incluso la química de la superficie influyen en la función celular [15,16,17]. Con este fin, se han desarrollado una gran cantidad de sustratos diferentes para estudios celulares [3, 18, 19, 20, 21, 22].
También se ha sugerido que los sistemas de cultivo 3D con mayor precisión podrían predecir el efecto in vivo de un fármaco y, por tanto, estos sistemas podrían encontrar aplicaciones en el descubrimiento de fármacos [16, 23, 24]. También se pueden utilizar patrones topográficos a nanoescala controlados con precisión para regular la morfología celular. Por ejemplo, las arrugas y los surcos se pueden utilizar para recrear la alineación estriada de los cardiomiocitos y así representar mejor las condiciones fisiológicamente relevantes para modelar diversas enfermedades [25, 26].
El citoesqueleto de la célula está conectado a la ECM y se facilita mediante adherencias focales (FA), un complejo multiproteico que incluye integrinas de la superficie celular y proteínas de andamio. Dependiendo de un conjunto complejo de mecanismos reguladores, los FA se forman y se desmontan a una tasa de rotación necesaria para el movimiento hacia adelante, por ejemplo, en la migración celular. Se sabe que los FA ejercen fuerza mecánica sobre el ECM y, a la inversa, el ECM que ejerce fuerza sobre las células influye en la afinidad y la avidez de la integrina en la membrana [27].
Una de las proteínas que se sabe que es parte integral de los ácidos grasos es la vinculina. Es una de las proteínas enlazadoras implicadas en el anclaje de F-Actina al complejo de integrina. La falta de vinculina altera la morfología celular, la adhesión y la motilidad [28], y altera la capacidad de las células para transducir la fuerza al sustrato [29, 30, 31]. La vinculina no solo participa en la conexión mecánica del citoesqueleto de actina con los complejos de integrina, sino que también tiene la capacidad de reticular y agrupar filamentos de actina [32,33,34], modificar los haces de actina existentes [35], captar los filamentos de actina, nuclear nuevos sitios de polimerización de actina [36] y reclutar modificadores de actina [37].
Las células responden a las matrices 3D cambiando el número y el tipo de adhesión célula-sustrato e inducen cambios en la organización espacial del citoesqueleto. Estos cambios, a su vez, influyen en la distribución, el tamaño y la dinámica de las adherencias formadas [4, 38, 39, 40, 41]. Este reordenamiento puede conducir a cambios en la proliferación, morfología y motilidad celular [42].
Para comprender la influencia de los entornos 3D complejos en las células, es necesario desarrollar nuevos sistemas modelo en los que se puedan estudiar los procesos celulares y compararlos con controles planos. Como se sabe que la respuesta celular depende de las características físicas, mecánicas y químicas del sustrato de cultivo, es deseable fabricar sustratos celulares con propiedades controladas con precisión [43, 44, 45]. Además, es muy ventajoso si las células y el sustrato se pueden estudiar fácilmente utilizando técnicas de análisis ya establecidas, como la microscopía óptica.
Un tipo de sustrato que ha llamado la atención recientemente son las superficies planas decoradas con nanopilares o nanoalambres [18, 21, 46,47,48,49,50,51,52,53]. En comparación con, por ejemplo, los hidrogeles, estas superficies estructuradas no imitan el verdadero entorno 3D, pero tienen una topografía superficial bien definida. Estos sustratos se denominan típicamente 2.5D. Estos sistemas ya se han aplicado para facilitar la entrega de moléculas biológicamente relevantes en las células [54, 55], para monitorear la actividad enzimática [56], para probar la mecánica nuclear [57] y para estudiar cómo la sintonización de la curvatura de la membrana influye en varios factores relacionados con la membrana celular. procesos [58, 59, 60]. Al fabricar nanoestructuras sobre sustratos transparentes, es posible integrar este enfoque con la microscopía óptica.
El número de combinaciones posibles de diferentes líneas celulares, tipo de nanoestructura y geometría es alto, y abundan los ejemplos de la literatura. Li y col. describió el comportamiento de las células en superficies decoradas con nanoestructuras de fosfuro de galio colocadas al azar y cuantificó la fracción de células con grandes AF [61]. Se investigó la morfología celular y de FA en la superficie con varias densidades de área de nanocables. Los resultados indicaron que las células sembradas en superficies de baja densidad estaban en contacto con el sustrato y formaban grandes AF alrededor de los bordes de las células. Se detectaron FA grandes en una gran fracción de células en estas matrices. Para densidades de área de nanocables altas, las células se suspendieron en la parte superior de las matrices de nanocables y se observaron FA en forma de puntos debajo de las células. Una fracción menor de células en estos arreglos mostró grandes AF en comparación con las células en la superficie con baja densidad de área de nanocables.
Buch-Månson y col. estudiaron las interacciones de la superficie nanoestructurada entre células para matrices de nanocolumnas de silicio colocadas al azar en un sustrato de Si [62]. En el proceso de fabricación utilizado, se controló la densidad del área pero no la distancia pilar-pilar. La investigación de los FA mostró que las células en las matrices con la densidad de área intermedia tenían el mayor número de FA que también tenían la forma más asimétrica. Se sugirió que algunos de estos FA se formaron en las paredes laterales de las nanocolumnas. Esto no se observó en superficies con densidad de nanocolumnas de área alta y baja.
En trabajos anteriores hemos descrito protocolos detallados para la fabricación de nanoestructuras de polímero SU-8 en superficies planas de vidrio [63], y exploramos el comportamiento celular de dos líneas celulares diferentes en estas superficies [45, 48]. En este trabajo, utilizamos la litografía por haz de electrones (EBL) para fabricar superficies decoradas con estructuras de polímero SU-8 alineadas verticalmente para estudiar los cambios en la organización de FA y citoesqueleto de actina en la línea de células epiteliales de osteosarcoma U2OS. Realizamos análisis tanto cualitativos como cuantitativos de los cambios inducidos por la superficie con diferentes claves topológicas.
Resultados
Utilizando protocolos previamente establecidos, hemos fabricado cubreobjetos de vidrio decorados con matrices definidas con precisión de nanopilares (NP) SU-8 orientados verticalmente con separación variable y geometría definida [63]. Superficies con densidades de área NP de 456, 205, 115 y 29 NP / 100 μm 2 (correspondientes a pasos de 500 nm, 750 nm, 1000 nm y 2000 nm). Primero examinamos las tendencias generales en la morfología celular, la estructura del citoesqueleto de actina y las interacciones célula-sustrato. Seguimos esto mediante una comparación cuantitativa de la morfología de células y FA en varios sustratos nanoestructurados y controles de vidrio plano. Combinamos microscopía de alta resolución con fabricación de alto rendimiento para realizar análisis cualitativos de al menos \ ({\ approx 100} \) células para cada tipo de superficie, con imágenes después de 24 hy 48 h. En total, analizamos> 400 imágenes de alta resolución y 20 conjuntos de datos 3D.
La Figura 7 muestra una representación esquemática de las matrices NP (A, B) y las imágenes de microscopía electrónica de sustratos fabricados (C, D). Se montaron portaobjetos de vidrio que contenían estructuras nanofabricadas usando parafina bajo un fondo hueco, cultivo de 35 mm con las estructuras apuntando hacia arriba. Las imágenes de microscopía electrónica de vista lateral de arriba hacia abajo y con título que se muestran en la Fig. 1C, D muestran matrices de nanopilares con paso y altura de 1000 nm. La Tabla 1 muestra los parámetros geométricos de las matrices utilizadas en este trabajo, su clasificación, así como la densidad numérica del área NP correspondiente. Clasificamos las matrices NP en densas y escasas según el comportamiento de adhesión celular observado (ver más abajo).
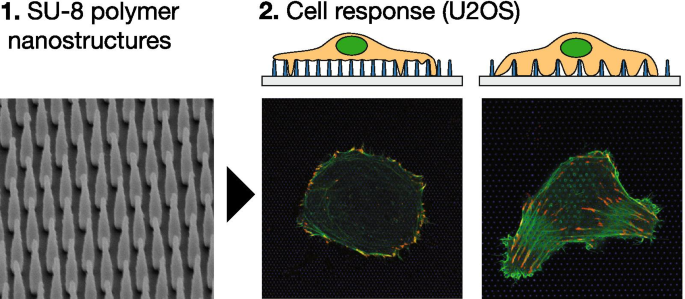
La Figura 1 muestra células U2OS representativas cultivadas en vidrio (A) y superficie nanoestructurada (B – F) durante 24 h. Las células se han cotransfectado con pCMV-LifeAct-GFP y pTAG-RFP-Vinculin, que permite la visualización de F-Actin y vinculina a través de la producción de proteínas de fusión LifeAct-TagGFP2 fluorescentes (en adelante:LifeActGFP) y TagRFP-vinculina, respectivamente. La red de F-actina y las áreas ricas en vinculina en los AF se detectan claramente. En la periferia celular, la señal LifeActGFP está muy cerca de la membrana y, por lo tanto, usamos esta señal para visualizar la morfología celular. La señal de los SU-8 NP se muestra en azul (consulte la sección "Experimental").
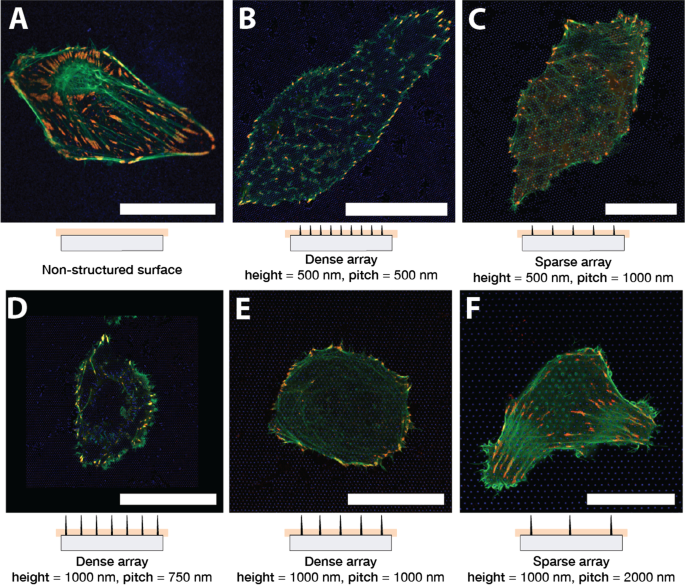
U2OS que expresa LifeActGFP fluorescente (verde) y TagRFP-vinculina (rojo) en diferentes tipos de superficies. La coloración amarilla indica señales superpuestas de los canales LifeActGFP y TagRFP-vinculina. Debajo de cada micrografía se muestra una vista lateral esquemática de la matriz NP correspondiente, junto con la posición aproximada del plano de adquisición. Imágenes de celdas en A superficie de vidrio plano no estructurado y en arreglos de pilares con diferentes pasos de arreglo y alturas de estructura: B Paso de 500 nm y altura de 500 nm, C Paso de 1000 nm y altura de 500 nm, D Paso de 750 nm y altura de 1000 nm, E Paso de 1000 nm y altura de 1000 nm, y F Paso de 2000 nm y altura de 1000 nm. Todas las imágenes presentadas son de células representativas. Barra de escala de 25 μm. Tenga en cuenta que C tiene una escala diferente a las otras imágenes
Las pruebas preliminares que utilizaron células transfectadas mostraron que las células sembradas en superficies de vidrio y estructuradas parecían estar completamente extendidas después de aproximadamente 6 h. No se observaron diferencias claras en la propagación celular entre las diferentes superficies, y la inspección visual no reveló signos de disminución de la viabilidad celular debido a las matrices de pilares en este punto o en experimentos posteriores. En los siguientes experimentos, las células se transfectaron 6 h después de la siembra, y luego se obtuvieron imágenes de 24 h, 48 h después de la transfección, correspondientes a la transfección 30 h, 54 h después de la siembra. A continuación, estos dos puntos de tiempo serán referidos por el tiempo de observación después de la siembra, es decir, 24 h, 48 h.
Las células se sembraron en matrices NP con una altura de 500 nm, 1000 nm y un tono de 750 nm, 1000 nm, 2000 nm. Después de la propagación inicial, se observó que las células eran redondas o alargadas, similar a la situación en superficies planas. Se encontró que esta morfología general era consistente en múltiples experimentos. Las células sembradas en matrices de NP dispersas generalmente tenían una forma similar a las células en la superficie del vidrio, ver la Fig. 1F que representa una celda representativa en una matriz con paso de 2000 nm. Las fibras de F-actina también estaban presentes en la base de las NP y en la proximidad de la superficie del vidrio, lo que indica que las células pudieron acceder al área cercana al sustrato. Como se observó anteriormente [45, 62, 64], las células en matrices densas típicamente parecían estar suspendidas en la parte superior de las NP (Fig. 1B, D, E). Las células en matrices densas parecen tener F-actina menos prominente cerca de la superficie del vidrio, lo que indica que las fibras de actina no se formaron entre los pilares en las proximidades del sustrato. La relación entre la altura de NP y la separación determinó si las células se adhirieron al sustrato o estaban suspendidas en la parte superior de la matriz de NP. Esto se ilustra, por ejemplo, en la Fig. 1C, donde los NP más cortos hacen que la célula entre en contacto con el sustrato, mientras que los NP más largos dificultan el contacto, Fig. 1E. Estas observaciones de fibras de actina fueron corroboradas adicionalmente por pilas z realizadas para algunas de las superficies, como se presenta en la Fig. 2.
Para obtener una comprensión más detallada de cómo las células se adhieren a la superficie estructurada y no estructurada, evaluamos la distribución de los FA según se visualiza mediante la presencia de la proteína de fusión TagRFP-vinculina. Las células en superficies planas típicamente formaron FA alargados distribuidos debajo de todo el cuerpo celular, como se muestra en la Fig. 1A.
En matrices dispersas, U2OS pudo entrar en contacto con la superficie del vidrio entre NP y se adhirió de manera similar a las células en el vidrio, como se muestra en la Fig.1C, F.Para estas matrices NP, los FA se formaron en el vidrio entre NP y la señal de actina F También se detectó en la imagen adquirida cerca de la base de los NP. Esto indicó que las células pudieron doblar la membrana alrededor de las nanoestructuras. Sin embargo, las células en matrices más densas, como 750 nm, separación de 1000 nm nm y 1000 nm de altura, se vieron claramente impedidas de adherirse al sustrato entre las nanoestructuras, como se muestra en las imágenes Fig.1D, E que se adquirieron cerca de la base de la NP. Sin embargo, alrededor de la periferia, las células normalmente podían unirse al sustrato entre las nanoestructuras que forman FA, a menudo dirigidas por la simetría de la matriz de pilares subyacente.
Las células que se extendían en matrices de NP con una longitud más corta y con un espaciado entre pilares de 1000 nm formaron adherencias tanto hacia la periferia como debajo del cuerpo celular. La orientación de la fibra de F-actina fue dirigida por la simetría de la matriz subyacente, como se muestra en la Fig. 1C. Sin embargo, la ubicación y la orientación de los AF que contienen vinculina no mostraron ningún patrón claro, y el AF formó NP intermedias.
Las células U2OS en pilares de 500 nm con una distancia entre pilares de 500 nm generalmente formaron menos adherencias y más pequeñas en comparación con la superficie plana como se muestra en la Fig. 1B. Para las células sembradas en esta matriz, las fibras de actina se observaron principalmente en las proximidades de la superficie del vidrio en las posiciones en las que terminaban en FA. Nuevamente, esta es una señal de que la red de actina no pudo entrar en contacto con la superficie y, por lo tanto, se asumió que las células estaban suspendidas en la parte superior de la matriz. Sin embargo, las células parecían tener una red de F-actina más intacta que se formaba por encima de los pilares. Partes de la red de actina que se observó entre los pilares, parecían alinearse con estructuras en la matriz NP subyacente. Esto se puede ver en la Fig. 1B, ya que las fibras de actina F y los FA se forman predominantemente a lo largo de una de las direcciones de la red, es decir, paralelas a las "líneas" abiertas, de la matriz de pilares.
Tanto en matrices densas como dispersas, observamos estructuras de actina F "en forma de anillo" que se formaron alrededor de NP que sobresale hacia arriba en el cuerpo celular. La estructura del anillo de actina F pareció ser más prominente en matrices dispersas, como se muestra en la Fig. 1E, F.
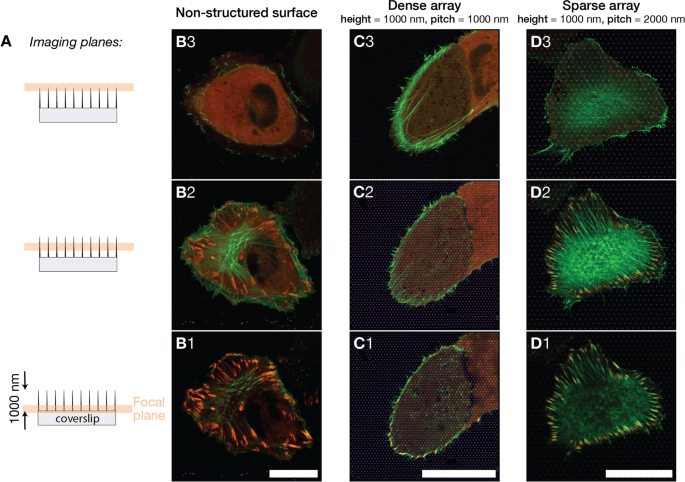
A Se adquirieron imágenes de células U2OS en diferentes superficies en tres planos focales indicados. Las imágenes se obtuvieron cerca de la base del pilar, aproximadamente a la altura de medio pilar y cerca del vértice del pilar. Los planos de imagen se ubicaron a 0.0 μm, 0.4 μm, 0.8 μm de la superficie plana del vidrio. B - D ) Imágenes de fluorescencia fusionadas que muestran U2OS que expresa LifeActGFP fluorescente (verde) y TagRFP-vinculina (rojo) en diferentes tipos de superficies, el color amarillo indica la superposición de LifeActGFP y TagRFP-vinculina. Las superficies con imágenes eran B superficie de vidrio no estructurado, C Matriz de pilares inclinados de 1000 nm, D Matriz de pilares con tono de 2000 nm. Las imágenes presentadas son representativas de las células de cada tipo de superficie. La distancia vertical entre planos focales para cada tipo de superficie es de aproximadamente 400 nm. Barras de escala de 25 μm
Basándonos en los resultados presentados anteriormente, hemos seleccionado tres superficies para una descripción más detallada y cuantitativa de la morfología celular y los FA. Estudiamos células en matrices densas (tono 1000 nm, longitud 1000 nm), matrices dispersas (tono 2000 nm, longitud 1000 nm) y comparamos los resultados con células en superficies de vidrio planas utilizadas como control.
Al emplear el detector Airyscan junto con el posprocesamiento de imágenes dedicado, pudimos realizar imágenes con una resolución xy de aproximadamente 140 nm y una resolución z de aproximadamente 400 nm [65]. La Figura 2 muestra imágenes de células en las tres superficies, con planos de imágenes separados por aproximadamente 400 nm. La investigación de los haces de F-actina en diferentes z . Las células de la superficie plana tenían una red de F-actina claramente visible en el mismo plano focal o justo encima de los FA (ver Fig. 2-B2). Para las células en matrices densas, la red de actina F se encontró en un plano focal más alto dentro de la célula en comparación con el plano FA, que estaba en contacto con el soporte de vidrio (Fig. 2-C1 y C2 / C3). Para las células en matrices dispersas, la situación fue similar a la célula en los controles de vidrio y la red de actina y los FA se detectaron a la misma altura (Fig. 2-D2). Estos datos apoyan la observación inicial de que las células en matrices dispersas unían la superficie entre las estructuras, mientras que las células en matrices densas podían adherirse principalmente a la superficie alrededor de la periferia celular.
Para analizar y cuantificar las diferencias en FA y morfología celular para las tres superficies seleccionadas, se utilizó un script de análisis de imágenes basado en Python (ver "Experimental"). Para el análisis cuantitativo se analizaron más de 300 imágenes de alta resolución. En estas imágenes, se identificaron> 400 células y> 7700 FA, la Tabla 2 enumera el número de células detectadas y FA para los tres tipos de superficie incluidos en el análisis. Para todas las superficies, se obtuvieron imágenes de las células tanto 24 h, 48 h después de la transfección. En el siguiente análisis, se comparan parámetros geométricos como el área de la superficie, la circularidad y la relación de aspecto para las celdas en 3 tipos de superficies y después de 24 h, 48 h. Se pueden encontrar análisis adicionales en la información complementaria. El área de superficie, circularidad y relación de aspecto de las celdas se muestran en la Fig. 3 y para el número de FA, el área combinada de FA por celda y la fracción de área de FA a celda en la Fig. 4. Los parámetros geométricos se definieron como se describe en el "Experimental" sección.
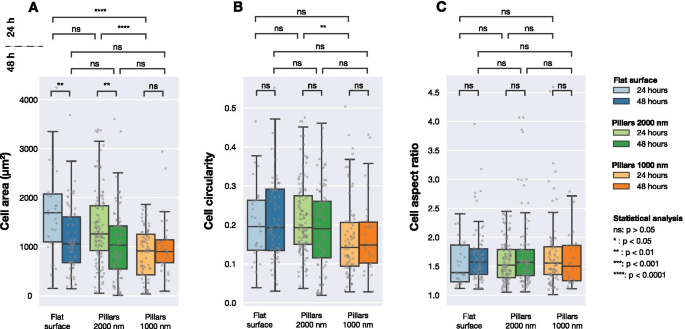
Área celular calculada, circularidad celular y relación de aspecto celular para células U2OS que expresan proteínas de fusión LifeActGFP y TagRFP-vinculina fluorescentes en diferentes superficies obtenidas en imágenes 24 hy 48 h después de la transfección. La detección de imágenes se realizó en función de la intensidad de la señal LifeActGFP. Cada punto gris corresponde a una celda y los diagramas de caja muestran valores medianos ( Q 2) así como el primero ( Q 1) y tercer cuartil ( Q 3). Las diferencias estadísticas entre las distribuciones se evaluaron mediante la prueba no paramétrica de Mann-Whitney
La Figura 3 resume los datos recopilados para superficies planas y estructuradas. Como se muestra en la Fig. 3A, se observaron diferencias significativas en el área celular después de 24 h de cultivo celular. Sin embargo, después de 48 h, no hubo diferencia significativa entre el área celular promedio en las superficies estudiadas. Al considerar la circularidad celular (Fig. 3B), no se detectaron diferencias significativas entre las diferentes superficies, excepto entre las células sembradas en pilares densos y dispersos fotografiados después de 24 h. Las celdas en las tres superficies tenían las mismas relaciones de aspecto promedio, como se presenta en la Fig. 3C.
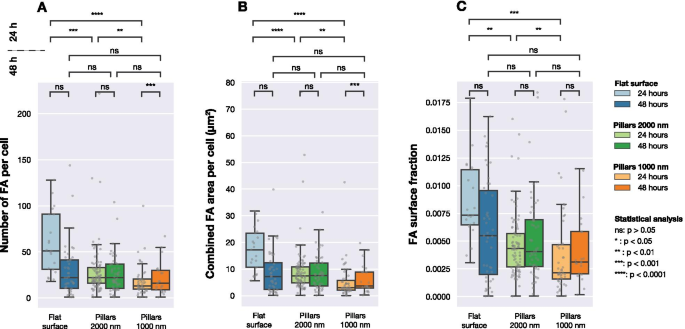
Número de FA, área de FA combinada por celda y fracción de área de FA a área de celda para las células fotografiadas en tres tipos de superficies diferentes (matrices planas y de pilares con pasos de 1000 nm y 2000 nm). Cada punto gris corresponde a una observación de una celda. La significancia estadística entre las distribuciones se determinó mediante la prueba no paramétrica de Mann-Whitneys
La Figura 4 muestra la distribución del número de FA detectados por celda, el área de superficie total de FA en cada celda y la relación entre el área de FA y el área de celda. Después de 24 h, el número de FA formados por las células en las tres superficies diferentes fue significativamente diferente. Como se muestra en la Fig. 4B, el área de superficie total de FA por celda fue diferente para las celdas sembradas en superficies planas y estructuradas. Lo mismo se puede ver al comparar la cantidad relativa de FA (el área total de FA detectados dividida por el área total de la celda) para las diferentes superficies, como se muestra en la Fig. 4C.
Sin embargo, después de 48 h de cultivo, ya no se observaron diferencias significativas entre las poblaciones de células. Al considerar el número de FA por celda, el área de FA combinada por celda o la fracción de superficie de FA, no se encontraron diferencias entre las tres superficies.
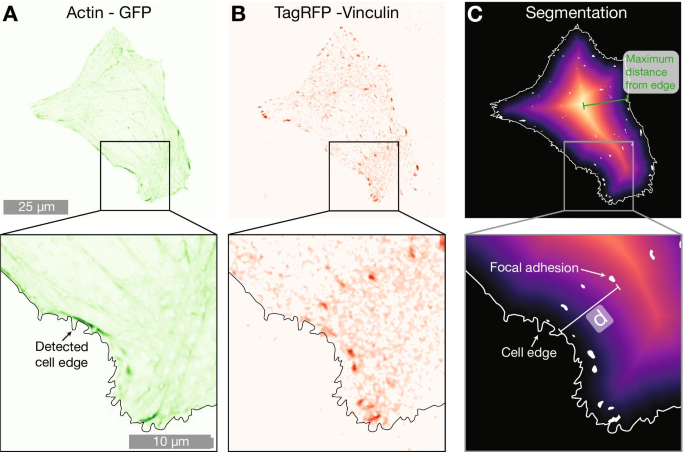
Celda de ejemplo en una matriz de pilares de 2000 nm que expresa A LifeActGFP (verde) y B TagRFP-vinculina (rojo). Superpuesto a ambas figuras está el borde de la celda detectado determinado mediante el uso de la señal de la expresión de LifeActGFP (como se muestra en A ). C Muestra un mapa de distancia desde el borde de la celda detectado, así como los FA detectados. La distancia más corta (indicada con d en la figura) desde cada punto de vinculina segmentado (áreas blancas) hasta la periferia celular (indicada con una línea blanca continua) se calculó para todos los FA en las imágenes
Para comprender si la presencia de NP influye en la localización de los FA en la célula, realizamos un análisis adicional utilizando la ubicación de los FA. Los datos de microscopía indicaron que los FA en células en matrices de NP densas se ubicaron más cerca de la periferia celular, como se indica en las Figs. 1 y 2. Para cuantificar esta tendencia, calculamos la distancia más corta desde cada FA hasta el borde de la celda. Esto se realizó como se ilustra en la Fig. 5. Se usó actina F para determinar la ubicación de la periferia y, mediante la construcción de mapas de distancia, se calculó la distancia entre cada centro de un FA detectado y la periferia de la celda. Para tener en cuenta las diferencias en el tamaño de las celdas, normalizamos las distancias entre el borde de la celda detectado y FA por la distancia máxima desde el borde al centro geométrico para cada celda (una distancia que es equivalente al radio para las celdas que tienen una forma circular). Los datos normalizados por la distancia máxima se presentan en la Fig. 6 y la Tabla 3. Para las celdas en superficies planas, los FA se distribuyen más hacia el centro de la celda, mientras que tanto en matrices dispersas de 2000 nm como en matrices densas de 1000 nm, las FA se encuentran más cerca de la periferia de la celda. Este efecto es más notable para matrices densas con un tono de 1000 nm. Los resultados de un enfoque de normalización alternativo, donde las ubicaciones de FA se normalizaron por área de superficie celular, se incluyen en la Información complementaria. Estos datos muestran las mismas tendencias cualitativas que los datos normalizados por la distancia máxima al borde en cada celda presentada en la Fig. 6.
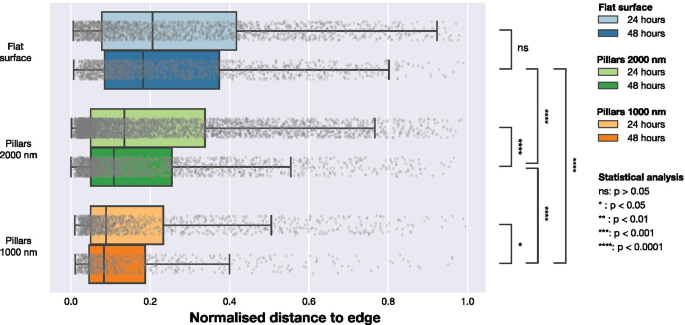
Distribución de las posiciones de los FA en relación con el borde de celda más cercano normalizado por la distancia máxima desde el centro geométrico de la celda al borde. Las distancias se obtuvieron calculando la distancia desde cada FA observado hasta el borde de la celda definido por la señal LifeActGFP y representado para los tres tipos de superficies a las 24 hy 48 h. Los puntos grises representan observaciones individuales de AF y sus distribuciones se resumen en los diagramas de caja. Para probar la probabilidad de que los FA de las diferentes superficies y puntos de tiempo fueran de la misma distribución, se realizaron pruebas de Mann-Whitney en todas las distribuciones con niveles de significancia indicados en la figura
Discusión
La organización del citoesqueleto de actina y la formación de adherencias son procesos ampliamente estudiados en superficies planas. El presente estudio fue diseñado para investigar y cuantificar los cambios en la organización del citoesqueleto de actina y las adherencias focales en matrices de nanopilares. En este estudio no hemos investigado cómo las matrices de nanopilares afectan la migración celular. Sin embargo, esperamos que las células de las matrices densas puedan tener una mayor motilidad, como se observó anteriormente en las mismas matrices de pilares para los fibroblastos embrionarios de ratón [45].
Permitir que las células se propaguen y se adhieran a la superficie durante un período prolongado de tiempo permite observar la organización del citoesqueleto de actina y la presencia de AG completamente maduros. 24 h después de la siembra, observamos diferencias significativas en el área celular, la circularidad y la relación de aspecto de las células sembradas en las superficies. Sin embargo, no se detectaron diferencias significativas después de 48 h, lo que indica que las células después de 24 h aún no se han adherido completamente a las superficies, y que las nanoestructuras influyen principalmente en la organización de los FA antes de que estén completamente maduros. Ni después de 24 h ni después de 48 h se observa formación de AF en la parte superior o en los lados de los NP.
Los cambios en la organización del citoesqueleto de actina también están relacionados con la forma en que una célula interactúa con el entorno. Por ejemplo, tanto las fibras de estrés como los FA crecen cuando se someten a estiramiento y parecen ser funcionales interdependientes [66]. Otros han informado de que las células más redondas y la localización de FA alrededor de los bordes se observan a menudo para las células sembradas en superficies blandas o adaptables [67]. En nuestros resultados, observamos una tendencia similar. Las celdas en matrices densas tienden a mostrar fibras alrededor del borde de la celda, como se muestra en las Figs. 1E o 2B. Los FA parecen formarse cerca del borde de la celda para estas celdas. Especulamos que cuando las células carecen de una superficie plana, como cuando las células están suspendidas en la parte superior de los pilares, la distribución de FA se asemeja a los FA en sustratos blandos, como la utilizada por Prager-Khoutorsky y colaboradores [67].
A partir de nuestros resultados, observamos que las células suspendidas en la parte superior de los pilares parecen tener una red de actina desarrollada por encima de los pilares, pero sin que se formen FA en los propios pilares. Sin embargo, para las células en matrices dispersas, las células parecen estar menos influenciadas por los NP y tanto la red de actina como los FA parecen más "como superficies planas".
La interacción entre los FA y el citoesqueleto de actina es compleja y aún no está completamente caracterizada. Se sabe que los FA que unen el citoesqueleto de actina al ECM actúan como puntos de tracción y promueven la formación de fibras de estrés en las células. Por el contrario, las fibras de actina están influyendo nuevamente en la organización y maduración de los AG. Numerosos estudios describen cómo las células tienden a estar suspendidas en la parte superior de matrices de NP densas [61, 62, 68] y cómo las membranas celulares interactúan con NP individuales [69, 70, 71]. Estas observaciones están corroboradas por estudios teóricos [64] y el comportamiento celular sobre pilares se comprende bastante bien.
El mecanismo detrás de la formación de FA y la unión al sustrato alrededor del borde de la célula en matrices densas sigue sin estar claro. A este respecto, la comparación con células sobre un sustrato blando es particularmente interesante. En el caso de sustratos blandos, las fibras de actina se organizan en forma de anillo cerca del borde de la célula y los FA se forman alrededor de la periferia de la célula [67]. On the nanopillar arrays similar type of architecture is observed, but the actin fibres are typically shorter. Similar qualitative trends in terms of actin organisation and FA formation were observed by Li et al. for cells seeded on random nanowire arrays made from gallium phosphide [61].
In our studies we also observed formation of F-actin rings around NPs. The formation of F-actin rings around NP has previously been described for fibroblasts on similar surfaces [45] and for U2OS cells on nanostructures with a range of structure sizes [58].
Contrasting our results to other studies highlight an important aspect of studies on cellular response to NP arrays:cellular response may vary considerably depending on cell type, NP material, NP geometry and as well as other parameters. For example, Buch-Månson et al. studied fibroblasts and investigations of FAs showed that cells suspended on arrays with intermediate NP density had the highest number of FAs. In our results we do not see a similar trend. However, these studies cannot be directly compared as Buch-Månson et al. studied another cell line using a system with different array geometry, surface porosity and NPs length [62].
There are also studies describing the effect FAs placement has on cells [41]. By modelling cells on planar substrates Stolarska et al. suggest that the cells can control intra-cellular stresses by three mechanisms:FA position, FA size and attachment strength. FAs around the periphery allows the cells to be more sensitive to changes in the micro-environment. This could also be an underlying mechanisms for cells on NPs. Yet, it is not obvious that the results for the planar substrate are directly transferable to NP decorated surfaces.
Cell-interactions with the surrounding environment, for flat substrate, NPs arrays or in vivo ECM, are regulated by a complex set of relations between actin organisation, membrane mechanics, cell dynamics and contact with FAs. To further explore these relations, applying flat surfaces structured with NPs could be one promising approach. Such surfaces may also aid in exploring discrepancies in the cellular response to environmental cues between different cell lines.
Conclusions
In order to create more physiologically relevant systems for cellular studies, a plethora of 3D and 2.5D approaches have been proposed. One approach is to use flat-surfaces decorated with vertically aligned nanostructures as a simple model system. High resolution live cell imaging of co-transfected U2OS cells expressing pCMV-LifeAct-GFP and pTAGRFP-Vinculin have been used to study the influence of nanopillar arrays on actin cytoskeleton focal adhesion organisation. Our present results indicate that the U2OS cells spreading on surfaces decorated with nanopillars can be categorised into three different regimes by how they respond to the nano-structures. These observed changes are quantified by analysing more than 400 high-resolution images, and indicate that tuning geometrical properties of the nanostructured surface can be used to direct cell behaviour.
More specifically, the U2OS cells were found to either contact the substrate, attach preferably around the cell edge, or be fully suspended on top of the vertical NP arrays. In the latter case, we hypothesise that the resulting reorganisation of FA and cytoskeleton is an effect analogous to what is seen for softer substrates.
Increased understanding of how cells behave on nano-structured surfaces, such as pillar arrays, could help us discover more details about complex cellular processes. For example, it is still poorly understood how changes in the actin cytoskeleton and its architecture influence cell signalling. By studying the cell response on nanostructured surfaces in a systematic way, the potential connection between actin cytoskeleton, cell adhesions and a plethora of biochemical signalling pathways could be further explored. We therefore envision that further development of the presented platform and analysis could have implications for advanced in vitro applications or for development of smarter in vivo biointerfaces.
Methods
Fabrication of Nanostructures and Sample Mounting
SU-8 nanostructures were fabricated as previously explained [63]. Briefly, 24 mm by 24 mm glass cover slips (#1.5, Menzel-Gläser, thickness 170 μm) were cleaned by immersion in acetone, isopropyl alcohol, rinsed in de-ionised water and dried. The cover slips were then oxygen plasma treated for 2 min (Diener Femto plasma cleaner, power 100 W, base pressure 0.3 torr), followed by dehydration for 10 min on a 150 °C hot plate. Samples were then placed in a desiccator containing an open vial of Hexamethyldisilazane (HMDS, Sigma Aldrich product no:440191). HMDS was applied by vapour deposition, the desiccator was pumped to low vacuum using a diaphragm pump for 5 min and the samples were kept in HMDS atmosphere for 60 min.
Substrates for EBL were prepared directly after HMDS treatment by spin coating SU-8 2001 (Microchem Corp.) to a desired thickness of 500 nm and 1000 nm. SU-8 was made fluorescent by adding either Oxazine 170 perchlorate, Rhodamine 800 or Coumarin 102 (all Sigma Aldrich) to a final concentration of 100 μg mL −1 resistir. After spin coating samples were dehydrated on a hot plate at 95 °C. To mitigate charging during EBL exposure samples were then covered by a layer of conductive polymer AR-PC 5091 Electra 92 (AllResist GmbH) by spin coating at 2000 rpm for 60 s to thickness of 50 nm.
An Elionix ELS-G100 100 kV EBL-system was used to fabricate SU-8 nanopillars (NPs) with processing parameters as described in our previous work [63]. Table 1 summarise the arrays fabricated for this work. Pillar arrays were exposed using the Elionix dot-pattern generator where each pillar is exposed in a single exposure. Arrays were exposed over an area of 2000 μm by 4000 μm, with a current of 500 pA in write fields of 500 μm by 500 μm. NPs had a tip diameter of about 100 nm as a base diameter of 150 nm and 200 nm for structures of length 500 nm and 1000 nm respectively.
After EBL exposure, the samples were rinsed in DI-water to remove the conductive polymer, then post exposure baked for 2.3 min at 95 °C and developed twice in mr-Dev 600 (Micro Resist Technology GmbH) developer for 20 s, rinsed in isopropyl alcohol and dried. Samples were then treated with oxygen plasma (Diener Femto plasma cleaner, power 50 W, base pressure 0.3 torr) for 30 s to render SU-8 hydrophilic and to give it similar surface chemistry as glass by oxidising surface epoxy-groups to hydroxyl.
Fabricated structures were imaged using Scanning electron microscopy (SEM) and samples sputter coated with 5 nm Platinum/Palladium alloy deposited with a 208 HR B sputter coater (Cressington Scientific Instruments UK). SEM was performed with a FEI Apreo SEM, at 5 kV and 0.2 nA with sample 45° pre-titled stage and with additional tilting of 30°.

A Side view schematic representation of nano-structured surface mounted in petri dish. Glass slides are mounted using paraffin such that structures are pointing upwards. B Tilted schematic representation of nano-pillar array on flat surface, and two important parameters for the nano-pillar arrays (height and pitch). These figures are not drawn to scale. C , D Overview of the nanopillar arrays employed in this work. Top-down and tilted side-view scanning electron micrographs of fabricated nano-pillar array with pillars of height 1000 nm and pitch 1000 nm. Scalebars 2000 nm
When exposing the pillars, an indexing system was also exposed to make navigation during live-cell imaging more reliable. Arrays were optically inspected after fabrication to ensure free and standing pillars. The short Oxygen plasma treatment to render the SU-8 structures did not lead to any optically visible change to the structures. Lastly, the samples were mounted underneath 35 mm diameter dishes (Cellvis, Mountain View, CA, USA) with 14 mm holes and nano-structures pointing upwards, as indicated schematically in Fig. 7. As flat surfaces, areas outside the structured part of the same samples were used. Before usage, all dishes were disinfected with 70% ethanol twice and dried.
Cell Culture and Transfection
U2OS-cells (ATCC) were cultivated in Dulbecco’s modified Eagle’s Medium (DMEM Prod. 41965039, Fischer Scientific) with 10% fetal bovine serum (FBS) and kept at 5% CO2 and 37 °C. Before detachment, cells were washed with PBS and detached with Trypsin-ethylenediaminetetraacetic acid (trypsin-EDTA) and seeded on nanostrucutred or flat surfaces. For the diameter 14 mm glass wells 15,000 cells were seeded.
For the standard transfection experiments, cells were allowed 6 h for adhering to surfaces before transfection. U2OS cells were transiently transfected using Lipofectamine 2000 (Invitrogen, Fischer Scientific) by adapting the manufacturer protocol to our system. Briefly, 2 μL Lipofectamine 2000 was added to 50 μL Opti-MEM I Reduced Serum Media (Prod. 11058021, Gibco , Fischer Scientific) and incubated for 5 min at room temperature. Plasmid DNA coding for fluorescent LifeAct-TagGFP2 and TagRFP-vinculin fusion proteins were co-transfected by using 0.5 μg plasmid DNA (vinculin-pTagRFP and pCMVLifeAct plasmids) was diluted in 50 μL Opti-MEM I and incubated at room temperature for 5 min. For co-transfection of TagRFP-vinculin and pCMVLifeAct 0.5 μg of each plasmid was used.
The diluted DNA was added to the diluted Lipofectamine 2000 in a 1:1 ratio, and left to incubate for 20 min at room temperature. 40 μL of the combined transfection complex was then added to each well. After 18 h, 1.5 mL DMEM (Prod. 41965039) supplemented with 10% FBS and 1% 10000U/mL Penicillin-Streptomycin was added to each dish.
For reverse transfection experiments, the same amounts of reactants were used, but the transfection complex was added to a suspension of U2OS cells, and the suspension was then added to the wells.
Microscopy
Live cell imaging was performed usin g a Zeiss LSM 800 Airyscan with an inverted Axio Observer Z1 stand connect to a PeCon compact incubator. Imaging was performed in an humidified environment at 37 °C, with 5% CO2 fluir. High resolution imaging was performed using a Zeiss Plan-Apochromat 63x/1.4NA DIC M27 oil objective with Cargille Immersion Oil Type 37 (n =1.51) suited for use at 37 °C. All images were taken using the system optimised pixel size both in-plane (typically 34 nm) and for stacks in the vertical axis (typically 180 nm).
To minimise imaging bias, imaging was performed in a standardised manner where each pillar array was raster scanned and cells expressing both LifeActGFP and Vinculin RFP were imaged. The high resolution images were then processed using a Zeiss algorithm for reconstruction of AiryScan images and exported as CZI-files for further manual and automatised image processing.
Image Analysis
For all cells, cell shape was based on the expression LifeActGFP fusion protein and expression of TagRFP-vinculin was used to identify FAs. Segmentation of images was performed using a script written in Python 3 [72] using CZIfile [73] (version 2017.09.12) for reading the microscopy images in Zeiss-format. The python packages Scipy [74] and Scikit-image [75] were used for multi-dimensional image processing and image segmentation respectively.
To reduce the influence from fluorescence cross-talk from pillars (due to Oxazine 170 perchlorate, Rhodamine 800 or Coumarin 102), the pillar/surface channel was used as a background and subtracted from the TagRFP-vinculin imaging channel. A median filter (size:10 pixels) was applied to remove noise from the TagRFP-vinculin channel, followed by classification of the image into regions based on their intensity value using a Multi-Otsu approach. Multi-Otsu thresholding with three classes was applied. The first class was typically the background, the second class constituted the cytosolic vinculin, whereas vinculin rich areas in FAs appeared brighter and could be classified into a third class. The quality of the image segmentation was briefly assessed by comparison to manual segmentation.
Area of cells and vinculin rich regions were described by counting pixel numbers and from this the actual area was found by correcting for the pixel size. Shape geometries were described by fitting each region with an ellipse with the same second-moment as the segmented region. In order to describe the cell area geometry, three measures were used:(1) Aspect ratio defined as the ratio of the ellipse major axis to the minor axis. (2) Circularity given as,
$$\begin{aligned} C =\frac{4\pi *\text{Area}}{\text{Perimeter}^2}, \end{aligned}$$ (1)and roundness given as,
$$\begin{aligned} R =\frac{4*\text{Area}}{\pi *\text{MajorAxis}^2}. \end{aligned}$$ (2)Segmented vinculin areas with a fitted ellipse that were too round (aspect ratio \(\le 1.5\)) or too elongated (aspect ratio \(\ge 8.5\)) were rejected. In addition, vinculin areas smaller than 0.05 μm 2 were filtered out. In order to find the distance between each vinculin area and the cell edge, the shortest euclidean distance between each centroid (the centre of the fitted ellipse for each vinculin area) and the cell edge was calculated.
Statistical Analysis
Statistical comparisons of distributions were performed by using the non-parametric two-tailed Mann-Whitney test neither assuming normal distribution nor equal standard deviation. P -values \(\ge {0.05}\) were considered to represent a non-significant (ns) difference between the two populations. Significant values were denoted with * for p in 0.01 to 0.05, ** for p in 0.001 to 0.01, *** for p in 0.0001 to 0.001 and lastly **** for p \(\le {0.0001}\).
Abreviaturas
- DMEM:
-
Dulbecco’s Modified Eagle’s Medium
- EBL:
-
Electron Beam Lithography
- ECM:
-
Extracellular Matrix
- EDTA:
-
Ethylenediaminetetraacetic Acid
- FA:
-
Focal Adhesion
- FBS:
-
Fetal Bovine Serum
- GFP:
-
Green Fluorescent Protein
- HMDS:
-
Hexamethyldisilazane
- NP:
-
Nanopillar
- RFP:
-
Red Fluorescent Protein
- SEM:
-
Scanning Electron Microscopy
Nanomateriales
- Ejemplos de circuitos y listas de red
- Baterías especiales
- Célula solar
- Compuestos de grafeno y polímeros para aplicaciones de supercapacitores:una revisión
- Estudio in vitro de la influencia de nanopartículas de Au en las líneas celulares HT29 y SPEV
- Síntesis de nanocristales de ZnO y aplicación en células solares de polímero invertido
- Estudio de la fuerza de adhesión y la transición vítrea de películas delgadas de poliestireno mediante microscopía de fuerza atómica
- Promoción del crecimiento celular SH-SY5Y mediante nanopartículas de oro modificadas con 6-mercaptopurina y un péptido penetrante de neuronas
- Influencia de la rigidez elástica y la adherencia de la superficie en el rebote de nanopartículas
- Avances recientes en métodos sintéticos y aplicaciones de nanoestructuras de plata
- Fabricación de nanofibras helicoidales CA / TPU y análisis de su mecanismo



