El microARN-326 exosómico derivado de macrófagos M1 suprime la progresión de las células del carcinoma hepatocelular a través de la vía de señalización NF-κB mediadora
Resumen
La evidencia acumulada ha demostrado que el microARN (miR) derivado de exosomas derivados de macrófagos M1 puede regular la progresión del carcinoma hepatocelular (CHC). Sin embargo, no se ha informado del efecto de miR-326 derivado de exosomas derivados de macrófagos M1 sobre el HCC. Por lo tanto, el objetivo del presente estudio fue explorar el mecanismo del miR-326 exosómico de los macrófagos M1 en la regulación de la progresión de las células de HCC. RT-qPCR detectó la expresión de miR-326 en líneas celulares de HCC. La expresión de miR-326 en HCC se alteró por transfección y se detectó el efecto de miR-326 sobre la expresión de CD206 y NF-κB, la proliferación celular, la formación de colonias, la migración, la apoptosis y la invasión. Posteriormente, se aislaron exosomas de macrófagos M1. RT-qPCR identificó la expresión de miR-326 en exosomas derivados de macrófagos M1. La expresión de miR-326 en exosomas derivados de macrófagos M1 se modificó mediante transfección. Se cocultivaron exosomas derivados de macrófagos M1 con células HCC para averiguar sus efectos sobre el progreso biológico de las células HCC. Finalmente, se realizaron experimentos in vivo para verificar los resultados in vitro. MiR-326 disminuyó en las células de HCC y se enriqueció en exosomas derivados de macrófagos M1. La regulación positiva de miR-326 inhibiría la proliferación de células de HCC, la formación de colonias, la migración, la invasión y la expresión de CD206 y NF-κB y promovería la apoptosis, e inhibiría el crecimiento de tumores de HCC in vivo , mientras que la regulación a la baja de miR-326 mostró efectos opuestos. Los exosomas derivados de macrófagos M1 inhibieron la proliferación de células de HCC, la formación de colonias, la migración, la invasión y la expresión de CD206 y NF-κB y mejoraron la apoptosis, mientras que la sobreexpresión de miR-326 aumentó el efecto de los exosomas derivados de macrófagos M1 en las células de HCC. Se revela que miR-326 exosómico derivado de macrófagos M1 suprime la proliferación, la migración y la invasión, así como promueve la apoptosis del HCC mediante la regulación negativa de la expresión de NF-κB.
Introducción
El carcinoma hepatocelular (CHC) es el quinto cáncer más común a nivel mundial y el cáncer primario de hígado más común [1]. Manifestado por los datos del Registro Nacional de Cáncer de China, la tasa de mortalidad del cáncer de hígado primario figura en el tercer lugar, mientras que la incidencia es la cuarta entre las neoplasias malignas comunes [2]. Los principales factores de riesgo de CHC son la infección crónica por el virus de la hepatitis C y el virus de la hepatitis B, alimentos contaminados con aflatoxinas, obesidad, tabaquismo, consumo excesivo de alcohol y diabetes tipo 2 [3]. La quimioembolización transarterial es un tratamiento establecido para el HCC en estadio intermedio, que mejora la supervivencia en la mayoría de los pacientes con HCC en estadios intermedios o avanzados [4]. Actualmente, el diagnóstico de CHC depende principalmente de biomarcadores séricos y técnicas de imagen [5]. La tasa de supervivencia a 5 años relacionada con el CHC es sólo de alrededor del 60%, y la incidencia aumenta año tras año en los últimos años [6]. Dado eso, la investigación de un objetivo terapéutico preciso es una prioridad en el tratamiento del HCC.
Los macrófagos son células efectoras y reguladores importantes del sistema inmunológico, ejercen funciones enormes en la remodelación y reparación de tejidos y realizan funciones metabólicas orquestadas en casi todos los tejidos in vivo [7]. Se revela que los macrófagos M1 ejercen efectos promotores de tumores y mejoran la motilidad de las células de HCC [8]. El exosoma es una vesícula discoide con un diámetro de 40-150 nm [9]. Según Xu et al., Los microARN exosómicos (miARN) tienen funciones sobre la proliferación, la invasividad, la metástasis y la resistencia a los fármacos del CHC mediante la modulación de la expresión génica en las células diana [10]. Un estudio ha demostrado que los exosomas que contienen miR-326 podrían ser un objetivo clínico potencial en la esclerosis múltiple [11]. Los miRNA podrían actuar como oncogenes o como inhibidores de tumores mediante la modulación de un gran número de genes codificados por proteínas, mediante la degradación del mRNA y el bloqueo de la traducción en una secuencia específica [12]. Un estudio ha discutido que miR-326 suprime el crecimiento de células de HCC al perturbar la progresión del ciclo celular, así como potenciar la apoptosis, además suprime la invasión celular al disminuir el fenotipo de transición epitelial-mesenquimal [13]. Otro estudio ha informado de que miR-326 podría ser un objetivo terapéutico potencial para el tratamiento de pacientes con CHC [14]. Por lo tanto, este estudio analizó el mecanismo del miR-326 exosómico que regula la invasión y migración de las células de HCC.
Materiales y métodos
Declaración de ética
Todos los experimentos con animales cumplieron con la Guía para el cuidado y uso de animales de laboratorio de los comités internacionales. El protocolo fue aprobado por el Comité Institucional de Uso del Cuidado Animal del Tercer Hospital de la Universidad de Jilin.
Inducción e identificación de macrófagos
La línea celular monocítica humana THP-1 (Instituto de Zoología de Kunming, CAS, Kunming, China) se cultivó en medio RPMI 1640 (Gibco, CA, EE. UU.; Thermo Fisher Scientific, MA, EE. UU.) Que contenía suero bovino fetal inactivado por calor al 10% ( FBS). Se hicieron reaccionar células THP-1 con 150 ng / ml de 12-miristato 13-acetato de forbol (PMA; P8139, Sigma-Aldrich, SF, CA, EE. UU.) Y se incubaron en medio RPMI durante 24 h para obtener macrófagos M0. Luego, se observó la morfología celular antes y después de la inducción mediante tinción de Wright. Las células THP-1 y los macrófagos inducidos se resuspendieron en 5 μL de PBS, se dejaron caer sobre el portaobjetos de vidrio, se tiñeron con la solución de tinte de Wright y se mezclaron con la solución tampón a 1:2. Teñidas durante 10 min y enjuagadas con agua corriente, las células se observaron al microscopio. Además, los marcadores de macrófagos M0 (CD68 y CD206) se midieron mediante la reacción en cadena de la polimerasa cuantitativa de transcripción inversa (RT-qPCR). A continuación, se indujeron macrófagos en macrófagos M1 mediante incubación con 20 ng / mL de interferón (IFN) -γ (# 285-IF; R&D Systems, MN, EE. UU.) Y 10 pg / mL de LPS (# 8630; Sigma-Aldrich) durante 18 h. Los marcadores de macrófagos M1 (IDO1 e IL-12 p35) fueron examinados por RT-qPCR [15].
Extracción de exosomas
Los exosomas se extrajeron mediante un kit de separación de exosomas (ExoEasy Maxi Kit, Qiagen, Hilden, Alemania). El sobrenadante de macrófagos se acumuló en un tubo de centrífuga de 15 ml en condiciones asépticas y se filtró mediante una película de filtrado de 0,8 µM. El sobrenadante celular de cada grupo se añadió con tampón XBP (1:1) y luego se centrifugó con una columna de centrifugación por afinidad de membrana exoEasy a 500 g . Las células se suplementaron con 10 ml de tampón XWP y se centrifugaron con 3000-5000 g . La columna de centrifugación por afinidad de membrana exoEasy se incubó con 400 μL de tampón de elución XE y se centrifugó a 500 g . El tampón de elución se transfirió a una columna de centrifugación por afinidad de membrana exoEasy y se centrifugó a 500 g . El tampón de elución se almacenó durante 24 ha 4 ° C y luego se usó para la identificación. El resto se almacenó a -80 ° C.
Análisis de seguimiento de nanopartículas y observación TEM (NTA)
Los exosomas obtenidos anteriormente se dejaron caer en la malla de cobre de la membrana soportada por carbono y se les añadió ácido fosfotúngstico al 2%. La muestra se observó bajo un TEM y se recopiló y analizó la imagen óptima.
Las impurezas y partículas en PBS se eliminaron mediante un filtro microporoso de 0,22 µM. Según la densidad de partículas de los exosomas, el PBS filtrado se diluyó a la concentración adecuada y se detectó con un detector de nanopartículas Nanosight NS300 (Malvern, Westborough, MA, EE. UU.).
Después de la identificación, los exosomas derivados de macrófagos transfectados con el inhibidor de miR-326 y el control negativo (NC) del inhibidor de miR-326, el imitador de miR-326 y el imitador de miR-326 se extrajeron mediante un kit de separación de exosomas (Invitrogen).
RT-qPCR
El ARN total se extrajo con Trizol Reagent (Thermo Fisher) y la PCR en tiempo real se realizó utilizando SYBR-Green PCR Master Mix (Roche) y el ABI 7500 Real-Time PCR System (Life Technologies, Grand Island, NY, EE. UU. ). Las secuencias de los cebadores se muestran en la Tabla 1. El análisis cuantitativo se llevó a cabo utilizando el método de 2 - △△ Ct .
Análisis de Western Blot
Se extrajo la proteína total de células y exosomas. La concentración de proteína se determinó mediante el kit de ácido bicinconínico (BCA) (Boster Biological Technology Co. Ltd., Wuhan, Hubei, China). La proteína se añadió con tampón de muestra y se hirvió a 95 ° C, y cada pocillo se cargó con 30 µg. La proteína se separó con electroforesis en gel de poliacrilamida al 10% (Boster Biological Technology) y se sometió a electrotransferencia sobre una membrana de fluoruro de polivinilideno, a lo que siguió el sellado en albúmina de suero bovino (BSA) al 5%. La membrana se incubó con anticuerpo primario contra CD63 (1:1000, Developmental Studies Hybridoma Bank, University of Iowa, Ames, IA, EE. UU.), CD181 (1:1000, R&D Systems), GAPDH (1:2000, Jackson ImmunoResearch Laboratories, PA, EE. UU.) Y con el anticuerpo secundario (1:500, Jackson ImmunoResearch Laboratories) marcado con peroxidasa de rábano picante. Las imágenes se obtuvieron con el sistema de escaneo de fluorescencia de infrarrojos de dos colores Odyssey, y los valores de gris de las bandas se midieron con el software de análisis de imágenes Cantidad Uno.
Cultivo celular y detección
La línea de células hepáticas normales HL-7702 y la línea celular de HCC humano BEL-7404, HepG2, SMMC-7721 y QGY-7703 se seleccionaron y cultivaron en Gibco RPMI Media 1640 con suero fetal bovino (FBS) al 10%, penicilina (100 U / ml ) y estreptomicina (100 mg / mL). La expresión de MiR-326 se detectó mediante RT-qPCR y se examinaron las líneas celulares adecuadas.
Etiquetado de exosomas y absorción de exosomas
Los exosomas se resuspendieron con 250 μL de Diluyente C y se trituraron suavemente para evitar dañar la membrana del exosoma. Se añadió el colorante PKH67 (1 μL, Sigma-Aldrich) a 250 μL de Diluyente C para alcanzar 500 μL y se incubó. La solución se añadió con 500 μL de BSA al 1% y se incubó a 37 ° C durante 1 min. Los exosomas se obtuvieron por centrifugación a 120.000 g 4 ° C durante 2 h. Los exosomas marcados con PKH67 se obtuvieron mediante centrifugación a 120.000 g 4 ° C durante 2 h. Los exosomas se resuspendieron con 6 mL de medio RPMI-1640 evitando la luz. Luego, los exosomas marcados se cultivaron conjuntamente con células HCC durante 12 h. Después de eso, el medio de cultivo se retiró y se lavó con PBS durante 3 veces, 5 min / vez, y los exosomas marcados con fluorescencia que no fueron absorbidos internamente por las células HCC se lavaron minuciosamente. Los exosomas se fijaron con paraformaldehído al 4% y se tiñeron con 4'-6-diamidino-2-fenilindol. Después del sellado, se observó la distribución de fluorescencia mediante un microscopio confocal láser.
Agrupación y tratamiento de células
Se sembraron células HepG2 y células SMMC-7721 en la placa de 12 pocillos a 0,5-1 × 10 6 células / pocillo. Con una confluencia del 50 al 60%, las células se transfectaron con Lipofectamine 2000 (Invitrogen, Carlsbad, CA). Las células HepG2 se distribuyeron en un grupo imitador de miR-326 (transfectado con un imitador de miR-326) y un grupo imitador de NC (transfectado con un imitador de miR-326 NC). Las células SMMC-7721 se asignaron al grupo inhibidor de miR-326 (transfectadas con inhibidor de miR-326) y grupo inhibidor de NC (transfectadas con inhibidor de miR-326 NC). Se mezclaron miR-326-mimic, miR-326-inhibitor y sus NCs con Lipofectamine 2000 para la transfección. Las células HepG2 y las células SMMC-7721 sin ningún tratamiento se establecieron como grupo en blanco. miR-326-mimic, miR-326-inhibitor y su NC fueron ideados y compuestos por Guangzhou RibBio Co., Ltd. (Guangzhou, China) (Tabla 1).
Cocultivo de exosomas derivados de macrófagos M1 con células de HCC
La concentración de proteína de la suspensión de exosomas derivados de macrófagos M1 se detectó mediante el método BCA y se calculó el volumen de la suspensión de exosomas correspondiente con 50 μg de proteína. Las células HepG2 y las células SMMC-7721 se sembraron en una placa de 12 pocillos a 1 × 10 5 células / mL por pocillo. Las células HepG2 se distribuyeron en 4 grupos:grupo de control (células HepG2 no cocultivadas con exosomas), grupo exosomas (Exo) (células HepG2 cocultivadas con exosomas derivados de macrófagos M1), grupo mímico Exo-miR-326 (HepG2 células cocultivadas con exosomas derivados de macrófagos M1 que se transfectaron con imitador de miR-326), grupo imitador Exo-NC (células HepG2 cocultivadas con exosomas derivados de macrófagos M1 que se transfectaron con NC imitador de miR-326). Las células SMMC-7721 también se asignaron a 4 grupos:grupo en blanco (células SMMC-7721 no cocultivadas con exosomas), grupo Exo (células SMMC-7721 cocultivadas con exosomas derivados de macrófagos M1), Exo-miR-326- grupo inhibidor (células SMMC-7721 cocultivadas con exosomas derivados de macrófagos M1 que se transfectaron con el inhibidor de miR-326), grupo inhibidor Exo-NC (células SMMC-7721 cocultivadas con exosomas derivados de macrófagos M1 que se transfectaron con miR- 326 inhibidor NC).
Ensayo de bromuro de 3- (4, 5-dimetiltiazol-2-il) -2, 5-difeniltetrazolio (MTT)
Las células se separaron con tripsina y se sembraron en una placa de 96 pocillos con una densidad celular de 4 × 10 4 células por pocillo. El medio de cultivo se abandonó después de cultivar 12, 24, 36, 48, 60 h, respectivamente. Se incubaron con 500 μL de solución de MTT 0,5 g / L, las células se añadieron a 200 μL de solución de dimetilsulfóxido, se trituraron y se incubaron. Los valores de densidad óptica (OD, 490 nm) se midieron con un lector de microplacas.
Ensayo de formación de colonias
Cultivadas durante 24 h y separadas con tripsina, las células se sembraron en una placa pequeña de 35 mm con 300 células por placa. La solución se reemplazó cada 3 d. Después de 10 d de cultivo, las células se fijaron con 40 g / L −1 paraformaldehído y teñido con 1 g / L −1 solución de violeta cristal y se seca. El número de colonias (más de 50 células) se calculó con un microscopio.
Ensayo Transwell
Celdas (1 × 10 5 ) se suspendieron con 200 μL de medio de cultivo en blanco. Los experimentos se realizaron de acuerdo con las instrucciones de la cámara Transwell (Corning Glass Works, Corning, N.Y., EE. UU.) (Se necesitaba matrigel para el experimento de invasión, pero no para el experimento de migración). Se añadió RPIM 1640 (10% FBS, 600 μL) en la cámara inferior. Las cámaras superior e inferior se separaron mediante una membrana Transwell prerrevestida con matrigel (BD Biosciences, Franklin Lakes, Nueva Jersey, EE. UU.). Cultivada durante 24 h, la cámara se fijó con alcohol al 95%. Después de teñir con una solución de violeta cristal, las células se observaron en cinco campos visuales bajo el microscopio.
Citometría de flujo
Ciclo celular:las células se desprendieron mediante tripsina. Celdas (1 × 10 6 ) se suspendieron con 0,5 ml de PBS y se trituraron en una única suspensión. Mezclado con 4,5 ml de etanol al 70% preenfriado en hielo, las células se centrifugaron a 3000 g , se enjuaga con 5 mL de PBS y se centrifuga nuevamente a 3000 g . Seguido de eso, las células se suspendieron con 1 ml de solución de tinción de PI / Triton X-100 (20 µg de PI / Triton X-100 al 0,1%) que contenía 0,2 mg de RnasaA. El ciclo celular se detectó mediante citometría de flujo.
Apoptosis celular:células tripsinizadas (1 × 10 6 ) se suspendieron con 1 ml de PBS, se trituraron y se centrifugaron a 3000 g . Las células se enjuagaron a su vez con tampón de incubación (10 mmol / L de Hepes / NaOH, pH 7,4, 140 mmol / L de NaCl, 5 mmol / L de CaCl 2 ) y centrifugado a 3000 g . A continuación, las células se incubaron con 100 μL de solución de marcado (se agregaron FITC-Anexina V y PI al tampón de incubación para alcanzar 1 μg / mL), se centrifugaron a 3000 g , se lavó una vez con tampón de incubación y se incubó con una solución fluorescente (SA-FLOUS). La apoptosis celular se detectó mediante citometría de flujo. La longitud de onda de la citometría de flujo fue de 488 nm y la fluorescencia FITC se detectó mediante un filtro de banda a 515 nm, mientras que el PI con una longitud de onda superior a 560 nm. Los resultados fueron analizados automáticamente por computadora.
Xenoinjerto de tumor en ratones desnudos
Se distribuyeron aleatoriamente cuarenta ratones (Facultad de Ciencias Animales, Universidad de Jilin, Jilin, China) de 4 a 6 semanas de edad en 8 grupos, con 5 ratones en cada grupo. Los ratones se alimentaron en un laboratorio de animales de grado libre de patógenos específicos durante 1 semana, y el alimento, el cojín y la botella de agua se reemplazaron a tiempo. El estado de salud de los ratones debe observarse todos los días. Una semana después, las células de HCC se prepararon en suspensión celular y se inyectaron por vía subcutánea en el cuello y la espalda a 0,1 ml de suspensión celular (1 x 10 6 ). El crecimiento del tumor se observó después de 3-5 d. Los ratones desnudos se pesaron cada 4 días y se midió el volumen del tumor. Los ratones desnudos fueron sacrificados 20 días después de la inyección.
Análisis estadístico
Todos los datos fueron interpretados por el software SPSS 17.0 (SPSS Statistics, Chicago, IL, EE. UU.). Los datos de medición se indicaron como media ± desviación estándar. Las comparaciones entre dos grupos fueron formuladas por t -prueba, mientras que las comparaciones entre múltiples grupos se evaluaron mediante análisis de varianza unidireccional (ANOVA). P valor <0.05 fue indicativo de una diferencia estadísticamente significativa.
Resultados
Identificación de macrófagos y exosomas M1
Se utilizó la tinción de Wright para observar la morfología de las células THP-1 inducidas por PMA. Se imaginó que el volumen de células THP-1 antes de la inducción era pequeño y la proporción de carioplasma era mayor; la morfología de las células después de la inducción fue irregular, el volumen se hizo más grande y la proporción de carioplasma disminuyó; el citoplasma era más rico y de color celeste, con abundantes partículas y pocas vacuolas; el núcleo era de color rojo violáceo y a menudo se inclinaba hacia un lado, mostrando que las células tenían las características morfológicas típicas de los macrófagos (Fig. 1a).
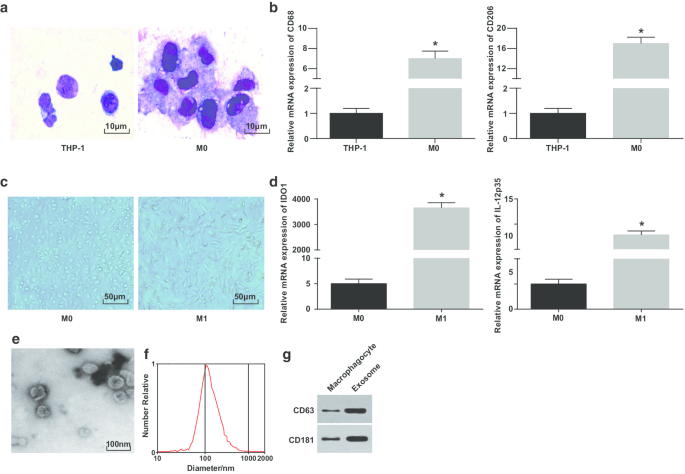
Identificación de macrófagos y exosomas M1. un Tinción de Wright para observar la morfología de las células THP-1 antes y después del tratamiento con PMA. b Expresión de CD68 y CD206 en células THP-1 y células THP-1 tratadas con PMA detectada por RT-qPCR; c morfología de macrófagos y macrófagos M1. d Expresión de IDO1 e IL-12 p35 en macrófagos M0 y macrófagos M0 tratados con LPS e INF-γ detectados por RT-qPCR. e TEM para la observación de exosomas. f Detección de la distribución del tamaño de partícula de exosomas por NTA. g Bandas de proteínas de CD63 y CD181. En el panel b, * P <0,05 frente a células THP-1; En el panel d, * P <0,05 frente a macrófagos M0. Los datos de medición se representaron como media ± desviación estándar ( N =3), las comparaciones entre dos grupos se realizaron mediante la prueba t
Para verificar aún más la inducción exitosa de macrófagos, se probó la expresión de CD68 y CD206 antes y después de la inducción mediante RT-qPCR. Se manifestó que la expresión de CD68 y CD206 estaba elevada desde la inducción de PMA, lo que indica que PMA indujo con éxito células THP-1 en macrófagos M0 (Fig. 1b). Luego, los macrófagos M0 se polarizaron en macrófagos M1 por inducción de LPS e INF-γ. Se observaron y ensayaron la morfología y la expresión del marcador de superficie de macrófagos de tipo M1 IDO1 e IL-12 p35 de los macrófagos. Los macrófagos M0 mostraron morfologías adherentes variadas e irregulares, mostrando forma redonda, elíptica o fusiforme. La morfología de los macrófagos estimulados por IFN-γ se demostró con más pseudópodos y protuberancias y fusiformes (Fig. 1c). RT-qPCR destacó (Fig. 1d) que después del tratamiento con LPS e INF-γ, los macrófagos M1 mostraron un aumento en sus marcadores (IDO1 e IL-12p35).
Posteriormente, los exosomas derivados de macrófagos fueron observados por el TEM. Se reveló que los exosomas derivados de los macrófagos eran ricos y la forma era redonda u ovalada, con estructura membranosa, tamaño uniforme y menos contaminantes (Fig. 1e). NTA mostró que los exosomas con un pico centralizado de curva MODE y lineal suave tenían un diámetro más concentrado y menos impurezas (Fig. 1f). El análisis de transferencia de Western informó que frente a los macrófagos, la expresión de la proteína marcadora específica CD63 y CD181 aumentó en exosomas derivados de macrófagos (Fig. 1g). Estos resultados indican que inducimos con éxito a los monocitos para que se diferenciaran en macrófagos y los polarizamos en macrófagos M1.
Exosomas derivados de macrófagos M1 Entregan miR-326 a las células de HCC y afectan la expresión de miR-326 en las células de HCC
Para verificar si los exosomas derivados de macrófagos M1 transportaban miR-326 a células HCC, se cultivaron conjuntamente células HepG2 y SMMC-7721 con exosomas. Se pudo reconocer que una gran cantidad de exosomas fueron asimilados por células HepG2 y SMMC-7721 después de una transfección de 4 h bajo el microscopio de fluorescencia (Fig. 2a, b).
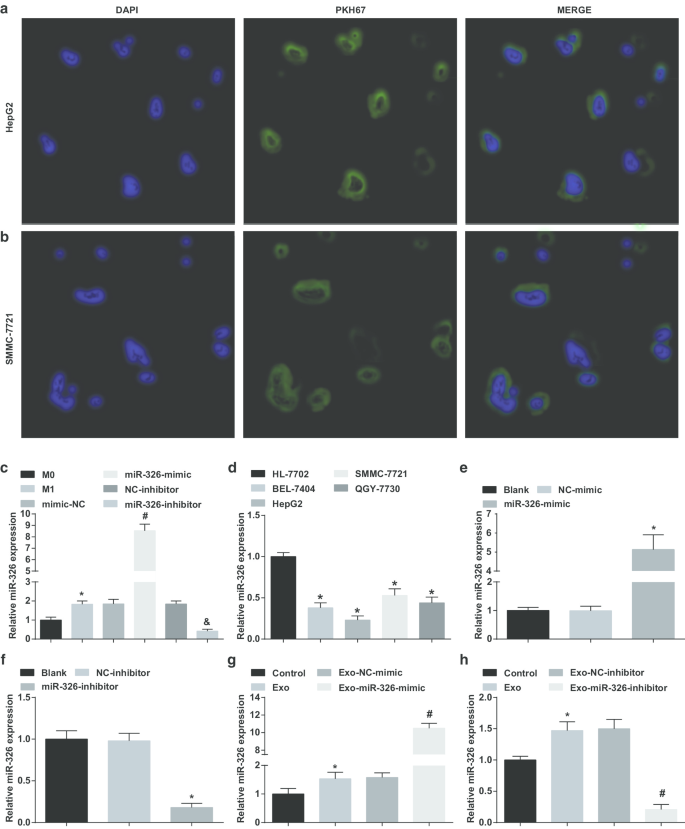
Los exosomas derivados de macrófagos M1 entregan miR-326 a las células de HCC y afectan la expresión de miR-326 en las células de HCC. un Captación de exosomas derivados de macrófagos M1 por células HepG2. b Captación de exosomas derivados de macrófagos M1 por células SMMC-7721. c Comparación de la expresión de miR-326 de exosomas de macrófagos en cada grupo antes y después de la inducción de INF-γ y LPS. d Expresión de miR-326 en líneas celulares de HCC (BEL-7404, HepG2, SMMC-7721, QGY-7703) y línea celular de hepatocitos humanos normales HL-7702 detectada por RT-qPCR. e RT-qPCR detectó el efecto del imitador de miR-326 sobre la expresión de miR-326 en células HepG2. f RT-qPCR detectó el efecto del inhibidor de miR-326 sobre la expresión de miR-326 en células SMMC-7721. g RT-qPCR detectó los efectos de exosomas derivados de macrófagos M1 transfectados con míR-326 sobre la expresión de miR-326 en células HepG2. h RT-qPCR detectó los efectos de exosomas derivados de macrófagos M1 transfectados con inhibidor de miR-326 sobre la expresión de miR-326 en células SMMC-7721. En el panel c, * P <0,05 frente al macrófago M0; En el panel d, * P <0,05 frente a células HL-7702; En el panel e, * P <0,05 frente al grupo imitador de NC; En el panel f, * P <0,05 frente al grupo inhibidor de NC; En el panel g, * P <0.05 versus el grupo de control, # P <0,05 frente al grupo que imita Exo-NC; En el panel h, * P <0.05 versus el grupo de control, # P <0,05 frente al grupo inhibidor de Exo-NC. Los datos de medición se representaron como media ± desviación estándar ( N =3), las comparaciones entre varios grupos se realizaron mediante análisis de varianza unidireccional
Posteriormente, el imitador / inhibidor de miR-326 se transfectó en macrófagos y se comprobó la expresión de miR-326 en sus exosomas antes y después de la transfección. Se demostró que frente a los exosomas de macrófagos M0, la expresión de miR-326 aumentó en exosomas derivados de macrófagos M1. Se observó una expresión elevada de miR-326 en los exosomas derivados de macrófagos M1 con un tratamiento imitador de miR-326. La expresión de MiR-326 se redujo en exosomas derivados de macrófagos M1 con el tratamiento con inhibidor de miR-326 (Fig. 2c).
Luego, se probó la expresión de miR-326 en líneas celulares de HCC. Como se manifestó, la expresión de miR-326 disminuyó en las células BEL-7404, HepG2, SMMC-7721 y QGY-7703 en comparación con las células HL-7702, entre las cuales las células HepG2 se manifestaron con la expresión más baja, mientras que las células SMMC-7721 con la expresión más alta (Figura 2d).
Seguido de eso, miR-326-mimic y miR-326-inhibitor se transfectaron en células HepG2 y SSMC-7721, respectivamente, y examinaron sus efectos sobre la expresión de miR-326. MiR-326 imita la expresión elevada de miR-326 en células HepG2, mientras que el inhibidor de miR-326 redujo la expresión de miR-326 en células SSMC-7721 (Fig. 2e, f).
A continuación, los exosomas de macrófagos M1 transfectados con miR-326-mimic y miR-326-inhibitor se cocultivaron con células HepG2 y SSMC-7721, respectivamente. Se destacó que el cocultivo con exosomas aumentó la expresión de miR-326 en células de HCC, los exosomas derivados de macrófagos M1 transfectados con míR-326 aumentaron aún más la expresión de miR-326 en células HepG2, mientras que los macrófagos M1 transfectados con inhibidor de miR-326 Los exosomas derivados degradaron la expresión de miR-326 en células SSMC-7721 (Fig. 2g, h). Se sugiere que los exosomas derivados de macrófagos M1 entregan miR-326 a las células de HCC y afectan la expresión de miR-326 en las células de HCC.
El miR-326 exosómico derivado de macrófagos M1 reduce la proliferación celular y la capacidad de formación de colonias en células de HCC
Al explorar el efecto del miR-326 exosómico sobre la proliferación de células de HCC, se llevaron a cabo ensayos de formación de colonias y MTT para examinar la proliferación de células de HCC. Se sugirió que en las células HepG2, la restauración de miR-326 alteraba la proliferación celular y la capacidad de formación de colonias (Fig. 3a, e, f). Los exosomas derivados de macrófagos M1 obstruyeron las células HepG2 para proliferar y formar colonias. Los exosomas derivados de macrófagos M1 transfectados con mimetismo MiR-326 afectaron aún más la proliferación celular y la capacidad de formación de colonias (Fig. 3c, i, j).
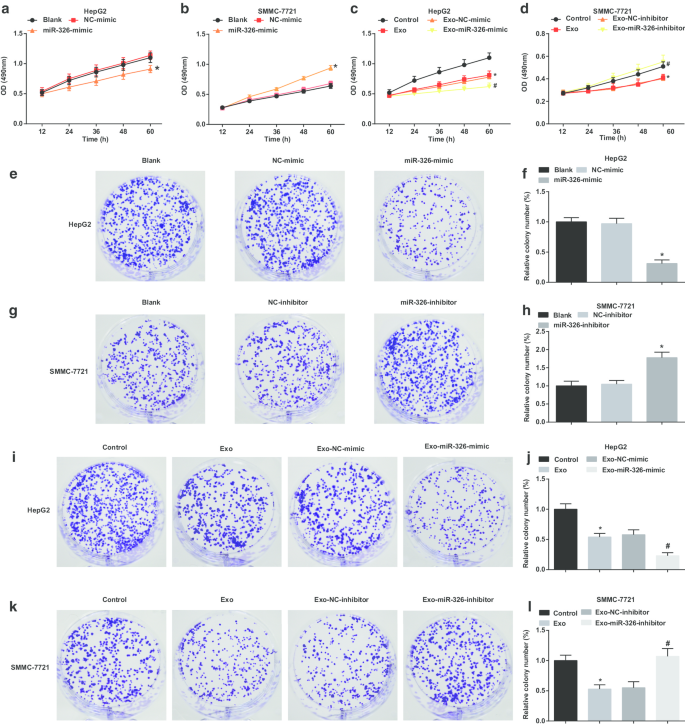
El miR-326 exosómico del macrófago M1 inhibe la proliferación celular y la formación de colonias en las células de HCC. un El ensayo MTT detectó el efecto de la transfección del imitador de miR-326 sobre la proliferación de células HepG2. b El ensayo MTT detectó el efecto de la transfección del inhibidor de miR-326 sobre la proliferación de células SMMC-7721. c El ensayo MTT detectó el efecto del cocultivo con exosomas derivados de macrófagos M1 transfectados con míR-326 sobre la proliferación de células HepG2. d El ensayo MTT detectó el efecto del cocultivo con exosomas derivados de macrófagos M1 transfectados con inhibidor de miR-326 sobre la proliferación de células SMMC-7721. e El ensayo de formación de colonias detectó el efecto de la transfección del imitador de miR-326 sobre la capacidad de formación de colonias de las células HepG2. f Número de colonias de células HepG2. g El ensayo de formación de colonias detectó el efecto de la transfección del inhibidor de miR-326 sobre la capacidad de formación de colonias de las células SMMC-7721. h Número de colonias de células SMMC-7721. yo El ensayo de formación de colonias detectó el efecto del cocultivo con exosomas derivados de macrófagos M1 transfectados con míR-326 sobre la capacidad de formación de colonias de las células HepG2. j Número de colonias de células HepG2 tratadas con exosomas. k El ensayo de formación de colonias detectó el efecto del cocultivo de exosomas derivados de macrófagos M1 transfectados con inhibidor de miR-326 sobre la capacidad de formación de colonias de las células SMMC-7721. l Número de colonias de células SMMC-7721 tratadas con exosomas. En el panel ayf, * P <0,05 frente al grupo imitador de NC; En el panel byh, * P <0,05 frente al grupo inhibidor de NC; En el panel cyj, * P <0.05 versus el grupo de control, # P <0,05 frente al grupo que imita Exo-NC; En el panel d y l, * P <0.05 versus el grupo de control, # P <0,05 frente al grupo inhibidor de Exo-NC. Los datos de medición se representaron como media ± desviación estándar ( N =3), las comparaciones entre varios grupos se realizaron mediante análisis de varianza unidireccional
En las células SMMC-7721, la caída de miR-326 mejoró la capacidad de proliferación celular y formación de colonias (Fig. 3b, g, h). En las células SMMC-7721 tratadas con exosomas derivados de macrófagos M1, la capacidad de proliferación celular y formación de colonias disminuyó. Los exosomas derivados de macrófagos M1 transfectados con inhibidor de MiR-326 estimularon aún más la proliferación celular y la capacidad de formación de colonias (Fig. 3d, k, l). Se insinúa que el miR-326 derivado de exosomas derivados de macrófagos M1 impide la proliferación de células de HCC.
El miR-326 exosómico derivado de macrófagos M1 suprime la migración y la invasión de células de HCC
Luego, se examinó el efecto del miR-326 exosómico sobre la invasión y migración de células de HCC. Se demostró que en las células HepG2, la restauración del miR-326 restringió la invasión y la migración (Fig. 4a-c). Los exosomas derivados de macrófagos M1 perturbaron a las células HepG2 para invadir y migrar. La invasión y la migración se degradaron aún más cuando las células HepG2 se cultivaron conjuntamente con exosomas derivados de macrófagos M1 transfectados con míR-326 (fig. 4g-i).
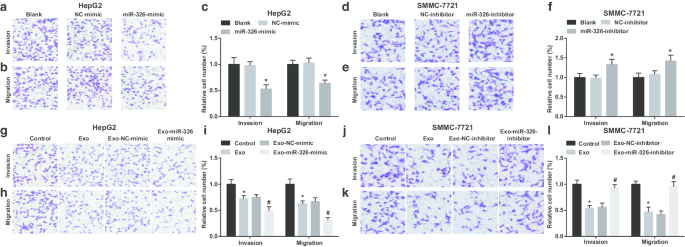
El miR-326 exosómico del macrófago M1 inhibe la migración y la invasión de las células de HCC. un El ensayo Transwell detectó el efecto de la transfección del imitador de miR-326 sobre la invasión de células HepG2. b El ensayo Transwell detectó que el efecto de la transfección de miR-326 imita la migración de las células HepG2. c El número de invasión y migración de células HepG2. d El ensayo Transwell detectó el efecto de la transfección del inhibidor de miR-326 sobre la invasión de células SMMC-7721. e El ensayo Transwell detectó el efecto de la transfección del inhibidor de miR-326 sobre la migración de células SMMC-7721. f El número de invasión y migración de células SMMC-7721. g El ensayo Transwell detectó el efecto del cocultivo con exosomas derivados de macrófagos M1 transfectados con míR-326 sobre la invasión de células HepG2. h El ensayo Transwell detectó el efecto del cocultivo con exosomas derivados de macrófagos M1 transfectados con míR-326 sobre la migración de células HepG2. yo El número de invasión y migración de células HepG2 cocultivadas con exosomas derivados de macrófagos M1. j El ensayo Transwell detectó el efecto del cocultivo con exosomas derivados de macrófagos M1 transfectados con inhibidor de miR-326 sobre la invasión de células SMMC-7721. k El ensayo Transwell detectó el efecto del cocultivo con exosomas derivados de macrófagos M1 transfectados con inhibidor de miR-326 sobre la migración de células SMMC-7721. l El número de invasión y migración de células SMMC-7721 cocultivadas con exosomas derivados de macrófagos M1. En el panel c, * P <0,05 frente al grupo imitador de NC; En el panel f, * P <0,05 frente al grupo inhibidor de NC; En el panel i, * P <0.05 versus el grupo de control, # P <0,05 frente al grupo que imita Exo-NC; En el panel l, * P <0.05 versus el grupo de control, # P <0,05 frente al grupo inhibidor de Exo-NC. Los datos de medición se representaron como media ± desviación estándar ( N =3), las comparaciones entre varios grupos se realizaron mediante análisis de varianza unidireccional
La caída de MiR-326 dio como resultado mejoras en la invasión y migración de células SMMC-7721 (Fig. 4d-f). When treated with M1 macrophages-derived exosomes, SMMC-7721 cells were exhibited with decreased invasion and migration. However, SMMC-7721 cell invasion and migration were boosted upon co-culture with miR-326-inhibitor-transfected M1 macrophages-derived exosomes (Fig. 4j–l). It is implied that miR-326 derived from M1 macrophage exosomes impedes the invasion and migration of HCC cells.
M1 Macrophage-Derived Exosomal miR-326 Promotes Apoptosis of HCC Cells
When examining the effect of exosomal miR-326 on the cell cycle and apoptosis of HCC cells, PI single staining and Annexin V-FITC/PI double staining were applied. It was illustrated that miR-326 overexpression increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases and raised apoptosis in HepG2 cells (Fig. 5a–d). Co-culturing with M1 macrophages-derived exosomes increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases and raised cell apoptosis of HepG2 cells. Co-cultivation with exosomes from M1 macrophages transfected with miR-326-mimic further enhanced these effects (Fig. 5i–l).
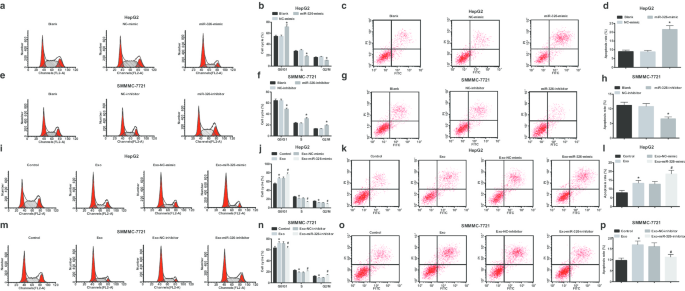
M1 macrophage exosomal miR-326 promotes apoptosis of HCC cells. un , b Flow cytometry detected the effect of transfection of miR-326 mimic on HepG2 cell cycle; c , d flow cytometry detected the effect of transfection of miR-326 mimic on HepG2 cell apoptosis; e , f flow cytometry detected the effect of transfection of miR-326 inhibitor on SMMC-7721 cell cycle; g , h flow cytometry detected the effect of transfection of miR-326 inhibitor on SMMC-7721 cell apoptosis; yo , j flow cytometry detected the effects of co-culture of with miR-326 mimic-transfected M1 macrophage-derived exosomes on HepG2 cell cycle; k , l flow cytometry detected the effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on HepG2 cell apoptosis; m , n flow cytometry detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on SMMC-7721 cell cycle; o , p , flow cytometry detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on SMMC-7721 cell apoptosis. In panel b and d, *P <0.05 versus the NC-mimic group; In panel f and h, *P <0.05 versus the NC-inhibitor group; In panel j and l, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel n and p, *P <0.05 versus the controlgroup, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (N = 3), comparisons among multiple groups were conducted by one-way analysis of variance
In SMMC-7721 cells, miR-326 down-regulation reduced cells arrested at G0/G1 phase, elevated cells arrested at S and G2/M phases, and declined cell apoptosis (Fig. 5e–h). Untransfected M1 macrophages-derived exosomes increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases, and heightened cell apoptosis. MiR-326-inhibitor-transfected M1 macrophages-derived exosomes degraded cells arrested at G0/G1 phase, elevated cells arrested at S and G2/M phases, and decreased cell apoptosis (Fig. 5m–p). Briefly, it is summarized that miR-326 derived from M1 macrophage exosomes arrests cell cycle in G0/G1 phase and induces cell apoptosis in HCC.
M1 Macrophage-Derived Exosomal miR-326 Declines CD206 and NF-κB Expression in HCC Cells
Next, the potential mechanism of miR-326 derived from M1 macrophage exosomes in the biological progress of HCC cells was explored. NF-κB is the key link between inflammation and cancer. Many regulatory proteins and miRNAs could inhibit the excessively activated NF-κB signaling to suppress cancer [16]. Such a beneficial effect may include the polarization of M2 macrophages into M1 macrophages. CD206 and NF-κB expression in HepG2 and SMMC-7721 cells was tested by RT-qPCR. It was suggested that miR-326 restoration decreased CD206 and NF-κB expression in HepG2 cells, while miR-326 knockdown enhanced CD206 and NF-κB expression in SMMC-7721 cells (Fig. 6a, b). Moreover, co-culture with M1 macrophage exosomes significantly reduced CD206 and NF-κB expression in HepG2 cells, while co-culture with M1 macrophage exosomes-overexpressing miR-326 further decreased CD206 and NF-κB expression. Treated with untransfected M1 macrophages-derived exosomes, CD206 and NF-κB expression was decreased in SMMC-7721 cells. Co-cultured with miR-326-inhibitor-transfected M1 macrophages-derived exosomes, SMMC-7721 cells were featured by heightened CD206 and NF-κB expression (Fig. 6c, d). It was concluded that miR-326 from M1 macrophage exosomes played a tumor suppressor by inhibiting NF-κB in HCC cells.
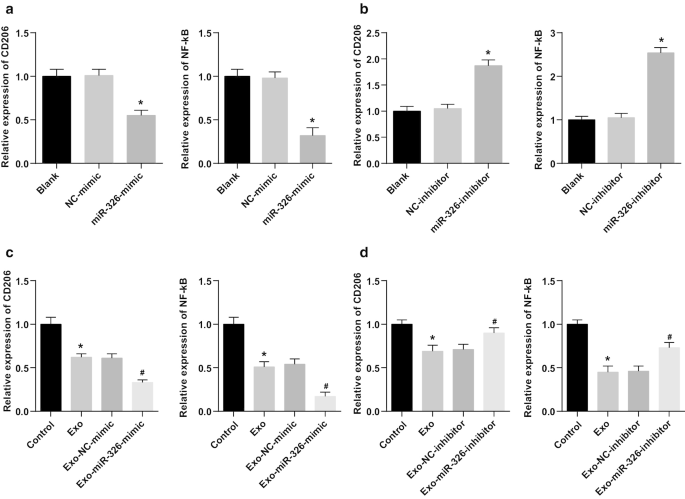
M1 macrophage exosomal miR-326 declines CD206 and NF-κB expression in HCC cells. un RT-qPCR detected the effect of transfection of miR-326 mimic on the expression of CD206 and NF-κB in HepG2 cells; b RT-qPCR detected the effect of transfection of miR-326 inhibitor on the expression of CD206 and NF-κB in SMMC-7721 cells. c RT-qPCR detected the effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on the expression of CD206 and NF-κB in HepG2 cells; d RT-qPCR detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on the expression of CD206 and NF-κB in SMMC-7721 cells. In panel a, *P <0.05 versus the NC-mimic group; In panel b, *P <0.05 versus the NC-inhibitor group; In panel c, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel d, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (N = 3), comparisons among multiple groups were conducted by one-way analysis of variance
miR-326 from M1 Macrophage Exosomes Inhibits HCC Tumor Growth In Vivo
Finally, the in vivo results were validated through tumor xenografts. As displayed, miR-326 overexpression decreased volume and weight of tumors in HepG2 cells (Fig. 7a–c). In mice transplanted with HepG2 cells co-cultured with exosomes, the treatment with M1 macrophage exosomes significantly reduced the tumor volume and weight of HepG2 cells, while treatment with M1 macrophages-overexpressing miR-326 further reduced the tumor volume and weight of HepG2 cells (Fig. 7g–i).
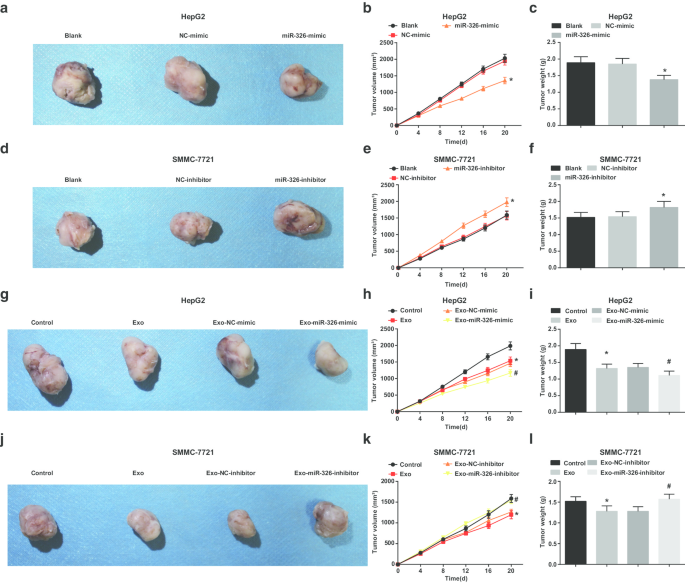
M1 macrophage exosomal miR-326 reduces the volume and weight of HCC tumor in vivo. un - c The effect of transfection of miR-326 mimic on the tumor volume and tumor of nude mice xenografted with HepG2 cells. d - f The effect of transfection of miR-326 inhibitor on the tumor volume and tumors of nude mice xenografted with SMMC-7721 cells. g - yo The effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on the tumor volume and tumor of nude mice xenografted with HepG2 cells. j - l The effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on the tumor volume and tumor of nude mice xenografted with SMMC-7721 cells. In panel b and c, *P <0.05 versus the NC-mimic group; In panel e and f, *P <0.05 versus the NC-inhibitor group; In panel h and i, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel k and l, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (n = 5), comparisons among multiple groups were conducted by one-way analysis of variance
In SMMC-7721 cells, miR-326 suppression increased volume and weight of tumors (Fig. 7d–f). Untransfected M1 macrophages-derived exosomes obstructed tumor growth in volume and weight of SMMC-7721 cells. Co-cultured with miR-326 inhibitor-transfected M1 macrophages-derived exosomes, SMMC-7721 cells were injected into mice and caused elevations in tumor volume and weight (Fig. 7j–l). Summarily, miR-326 derived from M1 macrophage exosomes depressed tumor growth of HCC in vivo.
Discussion
HCC is a common cancer that is characterized with high morbidity and mortality, difficult early diagnosis and treatment, poor prognosis and 5-year survival rate [17]. Recently, a study has highlighted that the lowly expressed lncRNA cox-2 declines the ability of M1 macrophages to suppress HCC cell growth, invasiveness, angiogenesis migration and promote apoptosis [18]. Hu et al. have discussed that miR-326 is obviously degraded in HCC tissues and cell lines, while down-regulated miR-326 is connected to the TNM stage, lymph node metastasis and differentiation of HCC patients [14]. It is customarily considered that HCC cells-derived exosomes can form a fertile environment to facilitate HCC cells growth, invasiveness and metastasis as well as development of drug resistance [19]. The current study was designed to explore the mechanism of exosomal miR-326 in regulating invasion and migration of HCC cells.
Our results indicated that miR-326 expression decreased in HCC cells but increased in exosomes. A recent study has pointed out that miR-326 expression is declined in HCC tissues [20]. Another study has presented miR-326 expression is notably reduced in HCC cell lines and tissues and its down-regulation predicts a poor prognosis in HCC [13]. It is reported that miR-326 acts a tumor-suppression role and is greatly depressed in HCC cells [21]. All these aforementioned evidences are in line with our findings. A study has purported that in comparison with the controls, miR-326 expression is raised in Tconv-derived exosomes which is observed in relapsing–remitting multiple sclerosis patients [11].
Other results emerge from our data that highly expressed exosomal miR-326 reduced cell proliferation, colony formation, migration and invasion as well as facilitated apoptosis of HCC cells in vitro and reduced the volume and weight of HCC tumor in vivo. It has been suggested previously that HCC cell growth can be suppressed via overexpression of miR-326, and HCC cell migration and invasion ability are markedly attenuated through elevating miR-326 [21]. It is reported that the up-regulated miR-326 expression suppresses HCC cell growth and invasiveness as well as stimulates cell apoptosis in vitro [14]. Besides that, a prior study has verified that overexpression of miR-326 declined tumor growth in vivo [13]. A study has revealed that ectopic expressed miR-326 markedly attenuates cell growth, and suppresses cellular migration and invasiveness in non-small cell lung cancer cell lines [22]. Moreover, it is found that miR-326 decreases profibrotic genes like MMP-9, implying its repressive function in cancer cell proliferation [23]. Also, it is presented that miR-326 represses Bcl-2 protein expression and elevates Bax expression so as to affect the apoptosis [24]. Similar to our findings, there are some miRNAs interacting with exosomes to play a role in HCC development. It is displayed that highly expressed exosomal miR-638 can repress the proliferation of HCC cells, involving the potential impact on carcinogenesis [25]. Another study also proves that HCC cells-derived exosomal miR-451a suppresses tumor angiogenesis via disrupting endothelial functions as apoptosis, tube formation, migration and permeability [26]. A prior research generally confirms that when treated with the overexpression of miR-744 exosomes, the proliferation of HCC cells is dramatically suppressed [27].
Conclusion
To briefly conclude, our study provides evidence that M1 macrophage-derived exosomal miR-326 suppresses proliferation, migration and invasion as well as advances apoptosis of HCC cells, supplying a new insight in a novel target therapy for HCC. Due to the limited sample size and limited known researches, the exact mechanism of miR-326 is not fully elucidated, and therefore, further large-scale studies are required to illustrate the underlying mechanism.
Abreviaturas
- HCC:
-
Carcinoma hepatocelular
- miRNAs:
-
MicroRNAs
- FBS:
-
Suero fetal bovino
- M/CSF:
-
Macrophage colony-stimulating factor
- PBS:
-
Phosphate buffered saline
- DMEM:
-
Dulbecco’s modified eagle medium
- PPAR:
-
Peroxisome proliferator-activated receptor
- TEM:
-
Microscopio electrónico de transmisión
- NTA:
-
Nanoparticle tracking analysis
- NC:
-
Negative control
- RT-qPCR:
-
Reverse transcription quantitative polymerase chain reaction
- BCA:
-
Bicinchoninic acid
- BSA:
-
Albúmina de suero bovino
- Exo:
-
Exosomes
- ANOVA:
-
Análisis de varianza unidireccional
Nanomateriales
- Control completo de polarización de terahercios con ancho de banda ampliado a través de metauperficies dieléctricas
- Estudio in vitro de la influencia de nanopartículas de Au en las líneas celulares HT29 y SPEV
- Efecto del electrodo ITO pulverizado de ángulo oblicuo en estructuras de células solares de perovskita MAPbI3
- Fabricación y caracterización de un nuevo catalizador anódico compuesto de nanofibras de carbono Tio2 para celdas de combustible de metanol directo mediante el método de electrohilado
- Eficacia antitumoral y farmacocinética mejoradas de la bufalina mediante liposomas pegilados
- Ajuste del modo de movimiento deslizante de los nanotubos de carbono mediante grupos de hidroxilo
- Evaluación de la actividad del citocromo P450 3A4 inhibido por nanopartículas de oro y los mecanismos moleculares que subyacen a su toxicidad celular en la línea celular de carcinoma hepatocelular …
- Diminutas nanopartículas de fluoruro de tierras raras activan el crecimiento de las células tumorales mediante interacciones polares eléctricas
- Mecanismo de falla de conmutación en una celda de metalización programable basada en peróxido de zinc
- Inhibidor de la autofagia (LY294002) y nanoliposoma a base de combinación de 5-fluorouracilo (5-FU) para una eficacia mejorada contra el carcinoma de células escamosas de esófago
- Secreción de células parietales gástricas y células caliciformes intestinales:una nueva vía metabólica de nanopartículas metálicas in vivo mediada por células mejorada con diarrea a través d…



