Identificación de macromoléculas características de genotipos de Escherichia coli mediante microscopio de fuerza atómica Mapeo mecánico a nanoescala
Resumen
La categorización de las cepas microbianas se basa convencionalmente en el método molecular, y rara vez se estudian las características morfológicas de las cepas bacterianas. En esta investigación, revelamos las estructuras macromoleculares de la superficie bacteriana a través del mapeo mecánico AFM, cuya resolución no solo estaba determinada por el tamaño de la punta a nanoescala, sino también por las propiedades mecánicas de la muestra. Esta técnica permitió el estudio a nanoescala de estructuras membranosas de cepas microbianas con una preparación simple de muestras y entornos de trabajo flexibles, que superó las múltiples restricciones en la microscopía electrónica y los métodos analíticos bioquímicos habilitados para etiquetas. Las macromoléculas características ubicadas entre la superficie celular se consideraron proteínas de la capa superficial y se encontró que eran específicas de Escherichia coli genotipos, a partir de los cuales se caracterizaron los tamaños moleculares promediados con diámetros que iban de 38 a 66 nm, y las formas moleculares eran parecidas a las de un riñón o redondas. En conclusión, las estructuras macromoleculares de superficie tienen características únicas que se vinculan con E. coli genotipo, lo que sugiere que los efectos genómicos sobre las morfologías celulares se pueden identificar rápidamente mediante el mapeo mecánico de AFM.
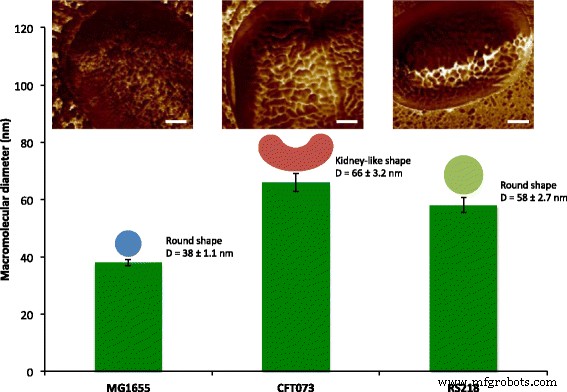
Cuantificación de macromoléculas de superficie de E. coli células que utilizan mapeo mecánico AFM. Macromoléculas de superficie de la superficie celular de tres E. coli Los genotipos MG1655, CFT073 y RS218 se caracterizaron con tamaños que iban de 38 a 66 nm y con formas redondas o parecidas a riñones. Las imágenes de la topografía se colorearon con mapeo de adherencia con las barras de escala =200 nm.
Antecedentes
Debido a la resolución inferior de la microscopía óptica y al entorno de trabajo restringido de la microscopía electrónica, los investigadores en el campo microbiano rara vez consideran usar la apariencia de células bacterianas, sino que adoptan métodos analíticos moleculares o químicos para la identificación de fragmentos genómicos, expresión de proteínas, etc. No es sorprendente que esos métodos tengan una serie de inconvenientes, entre ellos, que requieren mucho trabajo y tiempo y, por lo tanto, se necesitan enfoques más sencillos, eficientes y flexibles. Inventado en 1982 por Binnig et al., El microscopio de fuerza atómica (AFM) está diseñado para usar una sonda a nanoescala monitoreada por un rayo láser para la observación de la superficie de una muestra con resolución nanoscópica o incluso atómica [1]. Al tomar imágenes de la morfología a través de una punta de sonda física, esta técnica supera el límite de resolución y las restricciones ambientales de la microscopía óptica y electrónica y tiene una serie de ventajas, como la preparación simple de la muestra y entornos de trabajo flexibles en condiciones de aire o fluidos ambientales [2, 3]. Aún así, con respecto a los estudios microbianos, las aplicaciones actuales de AFM implican principalmente la obtención de imágenes cualitativas de las morfologías estáticas o dinámicas de las células bacterianas o la expresión de flagelos y pili [4, 5, 6], mientras que pocos estudios se han centrado en la ultraestructura superficial de células microbianas y el análisis cuantitativo de propiedades celulares.
En esta investigación, se seleccionó a AFM para el estudio de superficie de Escherichia coli ( E. coli ) células, y las formas y dimensiones de la bacteria individual se observaron mediante la topografía AFM y las imágenes de fase. Además, se descubrió que el mapeo mecánico simultáneo revela información biomecánica adicional sobre los componentes de la superficie, donde se pueden detectar pequeñas diferencias en las características adhesivas entre las macromoléculas y la matriz circundante durante cada contacto físico entre la punta y la muestra. Aplicando técnicas tan avanzadas a los campos microbianos, hemos examinado tres E. coli genotipos que contenían una cepa de laboratorio y dos cepas patógenas humanas para la identificación de macromoléculas de superficie. Los resultados mostraron que la técnica podría ofrecer una resolución de imagen superior a la escala de la punta AFM al detectar la distribución mecánica de la muestra. En conclusión, sugerimos que tal desarrollo en la ciencia de superficies no solo proporcionaría a los investigadores que trabajan en campos microbianos detalles de la apariencia celular, sino que también contribuiría a nuestro conocimiento de las características de la superficie de otros sistemas biológicos o nanomateriales.
Métodos
Muestras microbianas
Los tres E. coli Las cepas probadas en esta investigación fueron aisladas clínicamente y proporcionadas por el laboratorio del Prof. Ching-Hao Teng en el Instituto de Medicina Molecular de la Universidad Nacional Cheng Kung. MG1655 es la cepa de laboratorio intestinal y de tipo salvaje de E. coli K-12, y las otras dos cepas son patógenos humanos:CFT073, la principal causa de infecciones del tracto urinario, y RS218, asociada con meningitis neonatal en lactantes [7].
Sustrato funcionalizado
Se aplicaron dos pasos de modificaciones de superficie para la unión covalente entre la superficie sólida y las células microbianas. Primero, se utilizó una solución de 3-aminopropiltrietoxisilano (APTES, Sigma-Aldrich Co. LLC, EE. UU.) Para formar un NH 2 inicial -capa funcionalizada en la superficie, donde el recubrimiento APTES estable contribuyó a los enlaces Si-O estables; Si proporcionado por APTES y O de la superficie oxidada. Al poseer dos funciones COOH, el glutaraldehído se combina con el NH 2 función de APTES con un COOH y funciona con el NH 2 en la superficie bacteriana con el otro COOH.
Los sustratos limpios se sumergieron en solución de APTES, con una mezcla de APTES al 5% en etanol, durante una hora y se enjuagaron con etanol y ddH 2 O. Los portaobjetos se secaron usando una corriente de nitrógeno y luego se colocaron en una solución de glutaraldehído al 2% en PBS durante la noche y se lavaron con PBS.
Preparación de la muestra
Colonias individuales de E. coli Las cepas se seleccionaron de las placas de agar de caldo de lisogenia (LB) y se incubaron en caldo LB. Después de los tiempos de cultivo de 12 h, la solución bacteriana se diluyó luego 1:100 en caldo LB fresco preequilibrado. Después de otras 12 h para el cultivo microbiano, la solución bacteriana se sometió a centrifugación a 1500 × g (4000 rpm) durante 3 min y se resuspendió en caldo LB, repitiendo este proceso dos veces. Se dejaron caer doscientos microlitros de la solución bacteriana sobre el sustrato funcionalizado y se dejó reposar durante 30 min. Luego, la muestra se sumergió en agua destilada dos veces para eliminar las células sueltas y se fotografió inmediatamente bajo AFM en aire ambiente.
Caracterización de AFM
Se seleccionaron un instrumento AFM (Bruker Nano, Santa Barbara, CA, EE. UU.) Y una sonda de nitruro de silicio con la constante de resorte calibrada de 0,7 N / my un radio de punta de 10 nm para el examen de superficie de muestras microbianas. La frecuencia de exploración de AFM y los píxeles de línea fueron de 0,5 Hz y 256 líneas, respectivamente, para el tamaño de exploración de 10 μm para la primera detección de topografía, y luego los parámetros se establecieron como 0,3 Hz y 512 líneas para el tamaño de exploración de 2 μm para la observación detallada. El modo nanomecánico cuantitativo PeakForce (QNM) se utilizó para el mapeo nanomecánico, donde las propiedades adhesivas de la superficie se calcularon a partir de la fuerza de atracción máxima entre las curvas fuerza de extracción-distancia.
Nuestro estudio anterior sobre Streptococcus mutans mostró la evolución mecánica en la superficie bacteriana durante 2 h, monitoreada por mapeo mecánico continuo de AFM, y se verificó que las muestras microbianas permanecían vivas dentro de ese período [8, 9, 10]. Para garantizar la viabilidad de E. coli muestras utilizadas en este trabajo, se realizaron los estudios previos sobre las muestras bacterianas, y el cambio continuo en la adherencia de la superficie durante 4 h implicó que las células permanezcan vivas durante al menos 4 h después de la preparación de la muestra (que se muestra en el archivo adicional 1). En consecuencia, las muestras microbianas analizadas en este trabajo fueron medidas por AFM dentro de las 2 h posteriores a la preparación de la muestra. Todos los E. coli Los genotipos se cultivaron individualmente y en diferentes momentos, y las mediciones de AFM se realizaron inmediatamente después de la preparación de la muestra. En otras palabras, las muestras bacterianas no se alinearon esperando el examen, por lo que los efectos del tiempo de espera en las diferencias entre E. coli las tensiones se redujeron al mínimo. Los datos cuantitativos para cada E. coli Se obtuvieron cepas de las mediciones tomadas dentro de las 2 h en este estudio.
Análisis estadístico
Prism (GraphPad Software, EE.UU.) se utilizó para el análisis estadístico en este trabajo. Las longitudes celulares y los tamaños macromoleculares se presentaron como valores medios junto con el error estándar de la media (SEM). Las múltiples comparaciones entre E. coli Los genotipos se procesaron utilizando un análisis de varianza ordinario unidireccional (ANOVA). El nivel de confianza del 95% ( p <0.05), y los asteriscos indican el grado de diferencia significativa que se encontró. El número de muestra n de cada cepa era> 40.
Resultados y discusión
Ultraestructura de superficie por mapeo múltiple
Al escanear con AFM a una escala de observación de 10 μm en una muestra bacteriana, varios E. coli Se pudieron ver las células MG1655, y la forma celular en tres dimensiones se pudo observar a partir de la imagen topográfica (Fig. 1a); los contornos claros de las células mostradas en dos dimensiones se obtuvieron mediante la imagen del error de deflexión (Fig. 1b), y se pudieron encontrar varios túbulos además de las células bacterianas. Los filamentos en forma de onda (Fig. 1c) eran consistentes con la aparición de flagelos microbianos reportados en otro trabajo, que confirmó tal hallazgo como flagelos, y también se pudieron ver los pili más cortos y con forma de cabello [11]. Al disminuir el área de observación de la sonda para el estudio detallado de células microbianas individuales, la topografía mostró una ligera diferencia en la dirección vertical entre las superficies celulares, como se muestra en la Fig.1d, y la imagen del error de deflexión pareció proporcionar más información morfológica mientras el ruido ambiental recolectado fue demasiado para investigar la ultraestructura de la superficie celular (Fig. 1e). Cuando se midieron las propiedades biomecánicas simultáneas de la muestra durante el contacto entre la punta y el objeto probado, se descubrió que la superficie bacteriana en realidad estaba compuesta por una gran cantidad de macromoléculas con forma y tamaño específicos, como lo reveló el mapeo de la fuerza adhesiva ( Fig. 1f); por lo tanto, la resolución morfológica en las imágenes topográficas y de error de deflexión se mejoró aún más con información biomecánica.
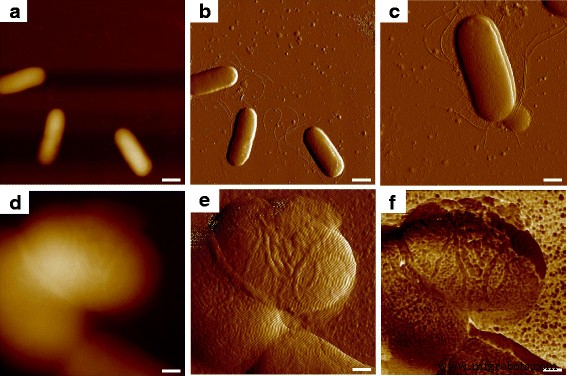
Ultraestructura superficial de E. coli MG1655 con mapeo múltiple AFM. un y b fueron las imágenes de error topográfico y de deflexión, y c fue la imagen detallada del error de deflexión de la célula bacteriana con la expresión de flagelos y pili. Luego se enfocó una sola celda, donde d y e fueron las imágenes de error topográfico y de deflexión, y f fue el correspondiente mapeo de adherencia. Las barras de escala =1 μm en a y b , 500 nm en c y 200 nm en d - f
En la Fig. 2a, la expresión de flagelos por E. coli Evidentemente, se pudo ver MG1655, donde el tamaño de los filamentos era similar a los de la Fig. 1b, con propiedades de adhesión relativamente más bajas que las del sustrato. Además, se descubrió que la superficie bacteriana estaba compuesta por componentes circulares que se caracterizaban por ser menos adhesivos en comparación con la matriz circundante. Esta observación es similar a nuestros hallazgos anteriores sobre las capas de tejido de la piel de los ratones, y los granos recurrentes y comparables se consideraron macromoléculas, cuya estructura es más densa y consistente que las observadas en la región intermolecular, de modo que las diferencias en el rendimiento de la adhesión podrían ser fácilmente detectado [12]. La capa más externa de la envoltura celular en las bacterias Gram-negativas es una capa de proteínas de autoensamblaje, como se ilustra en la Fig. 2b, que se conoce como proteína de la capa superficial (capa S) [13]. Las estructuras de la capa S se midieron tradicionalmente mediante microscopía electrónica, mientras que los requisitos de los entornos de vacío y el revestimiento conductor perdieron la información nativa y en tiempo real sobre las proteínas. Aunque algunas investigaciones extrajeron las proteínas de la capa S y las reensamblaron en un sustrato de mica para el escaneo AFM, faltaron los resultados para el desempeño in situ y en tiempo real de la estructura de la capa S [14, 15]. Sobre la base de la arquitectura celular y algunas imágenes previas de la superficie microbiana por microscopía electrónica, consideramos las macromoléculas observadas como proteínas de la capa S [16].
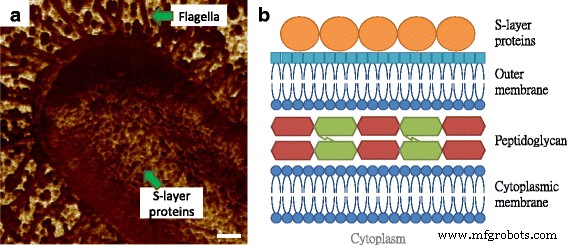
Ilustración de estructuras verticales y superficiales de E. coli células. un Las macromoléculas de superficie en E. coli Imagen de celda MG1655 mediante mapeo de adhesión AFM. b La arquitectura molecular de la envoltura celular en microbios gramnegativos, que consta de membrana citoplasmática, peptidoglicano, membrana externa y capa S. La barra de escala =200 nm
Al comparar las mediciones de AFM y microscopía electrónica de transmisión (TEM), la primera tiene varias ventajas sobre la última, como una preparación de muestras más simple, requisitos experimentales menos restringidos y aplicaciones de imágenes más amigables con la biología. La selección de TEM en el campo biológico generalmente se selecciona debido a su capacidad para ver los orgánulos intercelulares y las imágenes de ultra resolución (normalmente nanométricas o subnanométricas) que se pueden obtener. El mapeo de adhesión de AFM en el trabajo actual mejoró la resolución de la característica y presentó las disposiciones de las macromoléculas de superficie de una manera que no dependía del tamaño de la punta, sino de la estructura intrínseca de la muestra en sí. Además, este enfoque permite la resolución a nanoescala de la superficie microbiana en un rango de decenas de micrómetros cuadrados de área.
Diferencias de manipulación genómica en las características morfológicas
Después de observar la ultraestructura de la superficie de E. coli Células MG1655, un tema de interés es cómo se organizan las macromoléculas en otras cepas. Los patógenos humanos E. coli Así, CFT073 y RS218 fueron examinados por AFM con los mismos parámetros experimentales, y no hubo diferencias significativas en las formas y dimensiones celulares entre estos tres genotipos en el tamaño de característica de 10 μm, como se ve en la Fig. 3a-c. Se utilizó el mapeo de la fuerza adhesiva para identificar las proteínas de la capa S, y se detectaron estructuras diferentes con varias formas y tamaños entre las diferentes E. coli cepas, como se muestra en la Fig. 3d-f. Para facilitar la comparación, las imágenes detalladas de mapas de adherencia de E. coli las cepas se muestran en la Fig. 3g – i. Las macromoléculas de superficie se caracterizaron por tener una forma redonda en las células MG1655 y RS218, aunque con diámetros moleculares diferentes, que fueron 38 ± 1,1 nm ( n =80) para MG1655 y 58 ± 2,7 nm ( n =46) para RS218. Por otro lado, las células CFT073 poseían una forma única de proteínas de la capa S, que eran parecidas a un riñón con una diferencia de longitud entre dos puntos finales de 66 ± 3,2 nm ( n =44). Después de analizar los tamaños de las proteínas de la capa S de estos tres genotipos para realizar comparaciones múltiples, los resultados demostraron las diferencias significativas entre estas cepas (Fig. 4).
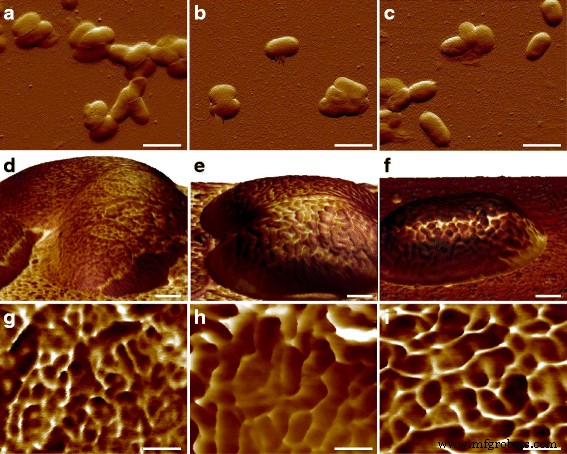
Características morfológicas de E. coli genotipos. La fila superior muestra las imágenes de error de deflexión de a MG1655, b CFT073 y c RS218. La fila del medio mostraba las topografías 3D coloreadas con mapas de adherencia en d MG1655, e CFT073 y f Células RS218. La fila inferior era el mapa de adherencia detallado en g MG1655, h CFT073 y i RS218. En el mapeo de adherencia, el color más oscuro se refiere a menores rendimientos de adherencia y viceversa. Las barras de escala eran de 2 μm para a - c , 200 nm para d - f y 100 nm por g - yo
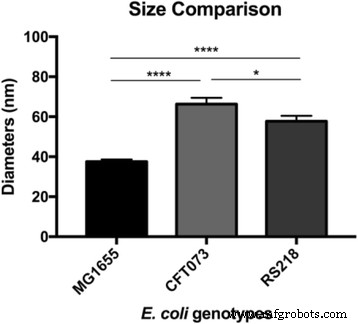
Tamaños moleculares de E. coli genotipos. Los diámetros de las proteínas de superficie se detectaron mediante AFM y se procesaron mediante ANOVA unidireccional para las comparaciones múltiples. **** p <0.001 y * p <0.05
Se informa que las capas S microbianas desempeñan papeles importantes en muchas funciones, que contienen células protectoras de entornos severos, ataques de fagocitosis y bacterias depredadoras. Además, las capas S también sirven como adhesina que permite la colonización eficaz [17]. Las estructuras de la capa S han sido bien estudiadas por TEM y se han categorizado en varios tipos de celosía con el espacio entre centro a centro en el rango de 4-35 nm [16]. La variación entre nuestros resultados de AFM y los informes de TEM de la literatura se pensó como las diferentes metodologías de imágenes, donde TEM proporciona la morfología 2D de la estructura de la capa S y AFM captura la topografía 3D que incluye las múltiples influencias contribuidas por el radián celular y la rugosidad y la geometría de la sonda AFM.
Originalmente se pensó que los diferentes tipos de estructuras de la capa S podían usar sus diversas características taxonómicas para distinguir entre especies bacterianas, aunque luego se descubrió que incluso para una sola especie, las tinciones microbianas podrían poseer diferentes redes de proteínas [13, 16, 18, 19]. Si bien algunos estudios investigaron el papel de la capa S en la formación de filamentos, los tipos de proteínas en las membranas celulares y las diversidades genómicas en tamaño entre E. coli genotipos, las diferencias en las proteínas de la capa S rara vez se han observado [20, 21, 22]. Los resultados del estudio actual revelaron las diferencias en las características morfológicas entre E. coli MG1655, CFT073 y RS218 y sugieren que las apariencias de las macromoléculas de superficie probablemente eran específicas del individuo E. coli genotipo.
Conclusiones
En este trabajo, se detectó información nanoestructural genómica específica sobre la superficie bacteriana mediante mapeo mecánico AFM, que distinguió las diferencias adhesivas entre las macromoléculas y la matriz circundante. Las macromoléculas de superficie de las células microbianas se consideraron como las proteínas de la capa superficial, de acuerdo con la arquitectura molecular de los microbios Gram-negativos. Se encontró que las disposiciones y tamaños de esas macromoléculas eran específicas de las E. coli genotipos con distintas formas y tamaños, y el análisis estadístico demuestra que estas diferencias son significativas. En conclusión, consideramos que la estructura de la capa S bacteriana depende del genoma y puede ser el método potencial para el diagnóstico rápido de enfermedades asociadas a microbios o cepas microbianas. Para implementar la aplicación práctica de la característica de la capa S, se requiere el examen de más genotipos bacterianos para el catálogo del complemento. Actualmente estamos estableciendo la base de datos que conecta las características morfológicas bacterianas y el desempeño fisiológico / patológico y creemos que será un progreso prometedor para la aplicación práctica del examen AFM.
Abreviaturas
- AFM:
-
Microscopio de fuerza atómica
- ANOVA:
-
Análisis de varianza unidireccional
- APTES:
-
3-aminopropiltrietoxisilano
- LB:
-
Caldo de lisogenia
- QNM:
-
Nanomecánica cuantitativa
- Capa S:
-
Capa de superficie
- TEM:
-
Microscopía electrónica de transmisión
Nanomateriales
- Los científicos de IBM inventan un termómetro para nanoescala
- Pioneros de AFM reconocidos con el Premio Kavli
- El próximo brote del gran petróleo está en la nanoescala
- Los no conductores conducen la corriente a nanoescala
- Síntesis biogénica, caracterización y evaluación del potencial antibacteriano de nanopartículas de óxido de cobre contra Escherichia coli
- Efecto superficial sobre el transporte de petróleo en nanocanales:un estudio de dinámica molecular
- Dispersión en ángulos pequeños de fractales gordos a nanoescala
- Dependencia de la resonancia de plasmón de superficie localizada del dímero de nanoprisma Ag truncado desalineado
- Fabricación de alto rendimiento de nanofibras de calidad utilizando un electrohilado de superficie libre modificado
- Fabricación de pozos a nanoescala con alto rendimiento en película delgada de polímero utilizando litografía de arado dinámico basada en puntas AFM
- Defectos en la superficie del nanofósforo de MgAl2O4 dopado con Ti



