Estabilidad y variabilidad de autoensamblaje de las proteínas de la cáscara del microcompartimento bacteriano en respuesta al cambio ambiental
Resumen
Los microcompartimentos bacterianos (BMC) son orgánulos proteicos autoensamblados que están muy extendidos entre el reino procariota. Al segmentar las enzimas y vías metabólicas clave utilizando una capa poliédrica, las BMC desempeñan un papel esencial en la asimilación de carbono, la patogénesis y la ecología microbiana. La capa de BMC se compone de múltiples homólogos de proteínas que se autoensamblan para formar la arquitectura definida. Existe un gran interés en la ingeniería de BMC para desarrollar nuevos nanobiorreactores y andamios moleculares. Aquí, presentamos la caracterización cuantitativa de la dinámica de formación y autoensamblaje de las proteínas de la cáscara de BMC en diferentes condiciones de pH y sal utilizando microscopía de fuerza atómica de alta velocidad (HS-AFM). Demostramos que la concentración de sal de 400 mM tiende a dar como resultado parches de concha de una sola capa más grandes formados por hexámeros de concha, y se observó una tasa dinámica más alta de autoensamblaje de hexámeros a pH neutro. También visualizamos la variabilidad de las proteínas de la cáscara desde ensamblajes hexaméricos hasta matrices similares a fibras. Este estudio avanza nuestro conocimiento sobre la estabilidad y variabilidad de los autoensamblajes de proteínas BMC en respuesta a cambios microambientales, lo que informará el diseño racional y la construcción de estructuras de BMC sintéticas con la capacidad de remodelar su autoensamblaje y robustez estructural. También ofrece una potente caja de herramientas para evaluar cuantitativamente el autoensamblaje y la formación de nanoestructuras basadas en BMC en aplicaciones biotecnológicas.
Introducción
Los microcompartimentos bacterianos (BMC) son orgánulos proteicos, que se asemejan estructuralmente a las cápsides virales, que dividen el citoplasma de las bacterias [1]. Están muy extendidos entre los filos bacterianos [2] y permiten que las bacterias compartimenten vías metabólicas clave en ausencia de orgánulos unidos a la membrana que se encuentran en los eucariotas [3, 4]. Las BMC están formadas por una capa de proteína semipermeable que encapsula un núcleo de enzima luminal. La cáscara se compone de tres tipos de componentes de proteínas estructurales, que incluyen BMC-H (que contiene un dominio Pfam00936), BMC-T (que contiene dos dominios Pfam00936) y BMC-P (con un dominio Pfam03319) [5, 6, 7, 8,9]. Los componentes principales del caparazón son BMC-H, que aparecen como hexámeros con superficies convexas y cóncavas y enlosan las facetas del caparazón con su lado cóncavo hacia afuera [10] (Fig. 1). BMC-P forman pentámeros que se proponen cubrir los vértices de la forma icosaédrica, y BMC-T forman pseudohexámeros que se encuentran en las facetas de la concha, presumiblemente responsables de la permeabilidad de la concha.
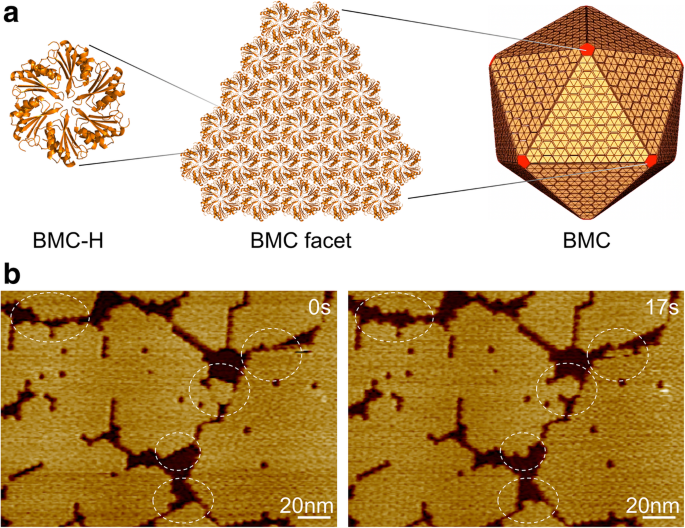
Microcompartimento bacteriano, organización de la cáscara y autoensamblaje. un Cientos de copias de homólogos de proteína de caparazón de BMC se autoensamblan para formar un orgánulo proteico icosaédrico. Las proteínas BMC-H, en amarillo, forman las facetas y las proteínas BMC-P, en rojo, ocupan los vértices. b Topografías AFM de facetas de concha compuestas por hexámeros Hoch_5815 BMC-H. Se observaron eventos dinámicos (círculos) en segundos usando HS-AFM
Las interacciones proteína-proteína específicas aseguran el autoensamblaje de las proteínas BMC para formar arquitecturas altamente definidas para cumplir con su funcionalidad metabólica. Se supone que las interacciones laterales entre las proteínas de la cáscara son el factor principal para determinar las propiedades de autoensamblaje de la cáscara icosaédrica [10]. Se ha observado que los homólogos de BMC-H pueden formar varias formas, incluidas láminas bidimensionales [11, 12], nanotubos [13,14,15,16,17] y estructuras de filamentos [15, 18,19,20] .
Sobre la base del autoensamblaje, la permeabilidad selectiva y las propiedades de encapsulación enzimática de los orgánulos naturales, las BMC se han considerado un sistema ideal con un gran potencial en bioingeniería, incluida la construcción bioinspirada de biorreactores a nanoescala que encierran enzimas metabólicas y la generación de nuevos andamios moleculares con nuevas funciones [21,22,23,24,25,26]. Sin embargo, quedan algunos problemas clave por abordar en la bioingeniería de BMC, por ejemplo, qué tan estables son las estructuras de BMC y cómo manipular y evaluar de manera efectiva el autoensamblaje y la formación de agregados de proteínas de BMC. Las investigaciones de las estructuras y el ensamblaje de las cáscaras de BMC y las BMC completas se han llevado a cabo mediante cristalografía de rayos X, microscopía electrónica (EM), microscopía de fluorescencia y dispersión dinámica de luz (DSL) [10, 11, 16, 22, 27,28 , 29,30,31]. Recientemente, hemos aprovechado el AFM de alta velocidad (HS-AFM) para realizar la primera visualización del proceso de autoensamblaje dinámico de las proteínas BMC-H [12].
En este trabajo, usamos HS-AFM para monitorear la dinámica estructural de los parches de BMC-H en diferentes condiciones iónicas y de pH, lo que brinda información sobre la modulación del ensamblaje de proteínas de la capa de BMC y ofrece una poderosa herramienta para la evaluación cuantitativa, en la resolución molecular , sobre la estabilidad y variabilidad del autoensamblaje de la proteína del caparazón de BMC.
Métodos
Preparación de la muestra
La proteína BMC-H purificada (Hoch_5815) de Haliangium ocraceum fue proporcionada amablemente por el Dr. Kerfeld (Laboratorio Nacional Lawrence Berkeley). Para el intercambio de tampones, muestras almacenadas en ~ 80 mg mL −1 en tampón Tris (Tris-HCl 50 mM, pH 7,8, KCl 100 mM, MgCl 2 10 mM ) se diluyeron a 0,5 mg mL −1 utilizando el búfer deseado antes de la proyección de imagen de AFM (archivo adicional 1:Figura S1). El tampón de control es Tris-HCl 50 mM (pH 7,8) y MgCl 2 10 mM .
Microscopía de fuerza atómica
Se usaron tampones deseados para la absorción de la muestra en mica y formación de imágenes AFM. Después de 5 min de absorción en la mica, Hoch_5815 se enjuagaron con el tampón deseado para eliminar las proteínas inmovilizadas y luego se tomaron imágenes con AFM (archivo adicional 1:Figura S1). Las imágenes HS-AFM se capturaron a 30 o 40 Hz en solución en modo CA utilizando un AFM JPK NanoWizard ULTRA speed equipado con un escáner ULTRA Speed 2.8 μm y sondas Ultra-Short Cantilever USC-0.3 MHz (NanoWorld). Se aplicaron fuerzas de carga mínimas de ~ 100 picoNewton durante las imágenes de AFM para reducir la alteración del ensamblaje de proteínas [12, 32, 33, 34, 35, 36].
Procesamiento y análisis de imágenes
El análisis de imágenes se realizó inicialmente utilizando JPK SPM Data Processing (JPK). El análisis de imágenes HS-AFM se realizó utilizando una macro personalizada en Image SXM (http://www.ImageSXM.org.uk), como se describió anteriormente [12]. Para analizar los tamaños de los parches Hoch_5815, se aplanaron imágenes de 512 × 512 píxeles capturadas a una velocidad de escaneo de 30 Hz para eliminar cualquier inclinación XY y umbral Z, seguido de conversión binaria para mostrar proteína versus no proteína. Se utilizó el análisis de partículas para calcular el área de superficie de las proteínas en estas imágenes binarias. Los parches se definieron como objetos separados por> 3 píxeles (~ 2 nm), con el fin de identificar parches individuales frente a parches adyacentes. Las pruebas iniciales mostraron que si se establece una mayor cantidad de píxeles, los parches adyacentes podrían contarse como un único parche continuo, mientras que si se usa un número de píxeles más pequeño, los espacios entre los hexámeros individuales en los parches podrían contarse erróneamente como el límite entre parches. Para analizar la dinámica de las proteínas, se analizaron series de imágenes de 256 × 256 píxeles capturadas a una velocidad de escaneo de 40 Hz, lo que dio una resolución temporal de aproximadamente 6,4 s por fotograma. Las imágenes binarias se restaron de la imagen anterior en la serie para mostrar imágenes AFM diferentes. Se empleó el análisis de partículas de las imágenes de diferencia para contar el área de proteínas ensambladas y desensambladas. La ecuación utilizada para calcular la tasa dinámica se muestra a continuación:
$$ \ mathrm {Tasa} \ \ mathrm {de} \ \ mathrm {dinámico} \ \ mathrm {eventos} \ (R) =\ frac {\ mathrm {Número} \ \ mathrm {de} \ \ mathrm {hexámeros} \ \ mathrm {agregado} \ \ mathrm {o} \ \ mathrm {eliminado} \ \ mathrm {in} \ \ mathrm {a} \ \ mathrm {s} \ mathrm {eries} \ \ mathrm {de} \ \ mathrm {marco} \ mathrm {s} \ kern0.5em (N)} {\ mathrm {Total} \ \ mathrm {superficie} \ \ mathrm {área} \ \ mathrm {de} \ \ mathrm {proteína} \ \ mathrm { en} \ \ mathrm {marco} \ kern0.5em (A) \ times \ mathrm {tiempo} \ kern0.5em (T)}, $$donde N representa la suma de píxeles blancos y negros en una imagen de diferencia de umbral dividida por el número de píxeles correspondientes a un único hexámero a esa escala (archivo adicional 1:Figura S3, Figura S5). Los datos se presentan como media ± desviación estándar (DE). El análisis estadístico se realizó mediante ANOVA multivariante o ANOVA bidireccional según lo especificado.
Resultados
Usamos las proteínas BMC-H (Hoch_5815) de una myxobacterium Haliangium ocraceum , que se expresaron en Escherichia coli y caracterizado como hexámeros con una simetría de seis veces [12]. Los hexámeros Hoch_5815 podrían autoensamblarse para formar láminas de una sola capa en la segunda escala de tiempo, que representan los componentes estructurales básicos de la arquitectura icosaédrica de BMC (Fig. 1a). Las imágenes HS-AFM nos permiten visualizar el ensamblaje dinámico y la flexibilidad organizativa de los fragmentos de hoja (Fig. 1b) y estimar cuantitativamente el tamaño del parche y la velocidad dinámica de las proteínas BMC-H utilizando el análisis de imágenes desarrollado (consulte la sección "Métodos").
Respuesta a la variación del pH
Medimos los cambios en el tamaño del parche como una indicación de la capacidad general de Hoch_5815 para autoensamblarse. El tamaño del parche aumenta con el aumento del pH de 3 a 10 (Fig. 2a; Archivo adicional 1:Figura S2, Tabla S1), lo que sugiere que un pH alto es más favorable para el autoensamblaje de proteínas Hoch_5815 que las condiciones de pH bajo. Esto es algo distinto de los comportamientos de ensamblaje de las proteínas RmmH, que resultaron ser insolubles a pH 6, forman matrices ordenadas de nanotubos a pH 8 y eran propensas a desensamblarse a pH 10 [13]. Además, observamos un alto grado de variabilidad estructural de los autoensamblajes HOCH_5815 (como lo indica una gran DE en la Fig. 2a, archivo adicional 1:Figura S2).
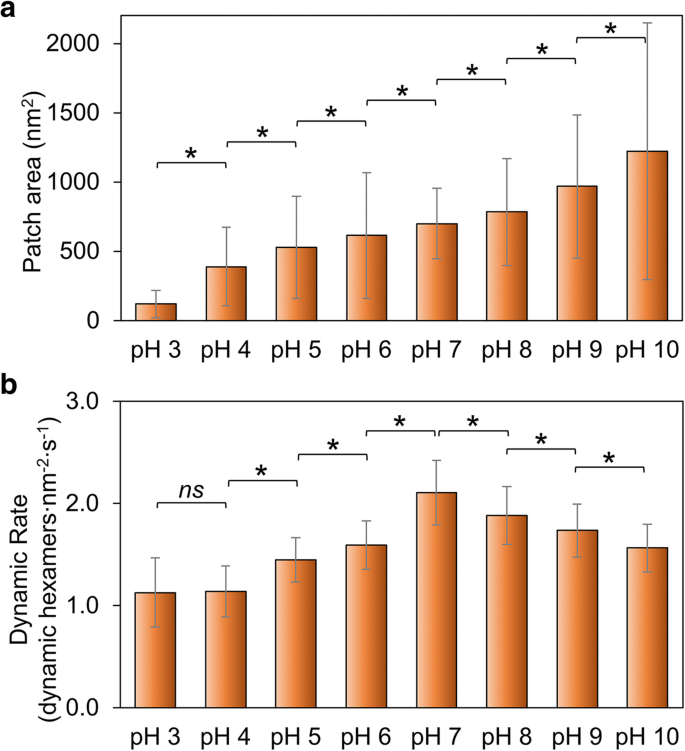
Efectos del pH ambiental en el autoensamblaje de Hoch_5815. un Las áreas superficiales medias de parches individuales de Hoch_5815 determinadas por AFM ( n =50) (Archivo adicional 1:Figura S2). b Las tasas medias de eventos dinámicos determinadas por HS-AFM ( n =50). * p <0.05, ns no significativo (ANOVA multivariado)
Las imágenes de AFM sobre el autoensamblaje de proteínas Hoch_5815 en láminas de caparazón han revelado que la formación de láminas de caparazón se atribuye a una combinación de ensamblaje y desmontaje de hexámeros [12]. Además, examinamos las tasas de dinámica de autoensamblaje Hoch_5815 y eventos dinámicos en diferentes pH (archivo adicional 1:Tabla S2) para explorar la estabilidad de las interacciones proteína-proteína Hoch_5815. La tasa de dinámica de autoensamblaje es la más alta a pH 7 y disminuye tanto en condiciones ácidas como alcalinas (Fig. 2b; Archivo adicional 1:Figura S3). En particular, disminuye rápidamente en condiciones ácidas, en particular de pH 7 a pH 6 y parece relativamente constante entre pH 4 y pH 3, como se muestra en la Fig. 2b.
Es probable que el pH tenga un gran impacto en las propiedades electrostáticas de los residuos de aminoácidos ubicados en la interfaz hexámero-hexámero. La dinámica disminuida y un tamaño más pequeño de parches de concha observados en condiciones ácidas ilustran que Hoch_5815 tiene una capacidad de autoensamblaje reducida. La dinámica reducida y un tamaño más grande de parches de concha observados en las condiciones alcalinas sugieren interacciones hexámero-hexámero estables, mientras que la dinámica aumentada de los hexámeros Hoch_5815 implica interacciones hexámero-hexámero flexibles en la condición de pH neutro.
Respuesta a la variación de las concentraciones de sal
También verificamos si la concentración de sal del tampón tiene impactos en el ensamblaje de Hoch_5815. En concentraciones bajas (100-200 mM) de MgCl 2 , CaCl 2 y KCl, las proteínas Hoch_5815 forman parches relativamente más pequeños que los ensamblados en concentraciones más altas (300-500 mM) (Fig. 3a; Archivo adicional 1:Figura S4). A 500 mM, observamos hojas Hoch_5815 de doble o multicapa (archivo adicional 1:Figura S4). Estas observaciones son consistentes con el hallazgo anterior de que una mayor fuerza iónica podría facilitar la formación de cristales 2D más extensos y bien ordenados por CcmK, las proteínas de la cubierta de los carboxisomas para la asimilación de carbono [37]. Sin embargo, los nanotubos altamente ordenados formados por RmmH se desmontaron cuando la concentración de NaCl se incrementó de 50 a 500 mM [13], lo que indica los mecanismos potencialmente diferentes que median la formación de láminas planas y formas tubulares por los hexámeros de caparazón.
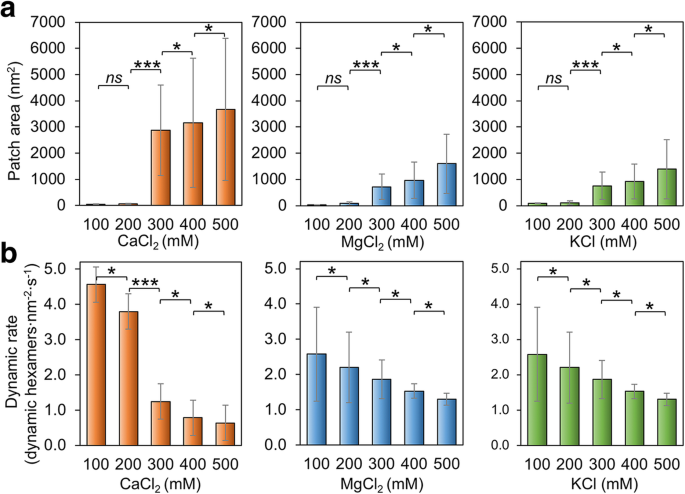
Efectos de la concentración de sal en el autoensamblaje de Hoch_5815. un Las áreas de parche medias medidas por AFM en un rango de CaCl 2 de 100 a 500 mM , MgCl 2 y KCl ( n =50). El aumento de la concentración de sal dio como resultado un aumento del tamaño de los parches. Se observaron cambios significativos en el área del parche entre 200 y 300 mM (*** p <0,001, * p <0.05, ns ANOVA bidireccional no significativo). b Las tasas medias de eventos dinámicos determinadas a partir de series de imágenes AFM de alta velocidad en un rango de CaCl 2 de 100 a 500 mM , MgCl 2 y KCl ( n =50). Cada cambio de 100 mM en la concentración de sal provocó un cambio significativo en la tasa de eventos dinámicos (*** p <0,001, * p <0.05, ns ANOVA bidireccional no significativo)
Además, las variaciones del autoensamblaje Hoch_5815 causadas por los cambios en MgCl 2 y las concentraciones de KCl son relativamente similares. Por el contrario, el cambio en el tamaño del parche es más pronunciado (hasta un aumento de 3000 veces) cuando el CaCl 2 la concentración se eleva de 200 a 300 mM (Fig. 3a), lo que sugiere la mayor sensibilidad del autoensamblaje de Hoch_5815 al CaCl 2 que a MgCl 2 o KCl.
La tasa dinámica de autoensamblaje de Hoch_5815 también se ve afectada por cambios en la concentración de sal tampón. El aumento de MgCl 2 , CaCl 2 , o las concentraciones de KCl podrían resultar en la disminución de la tasa dinámica Hoch_5815 (Fig. 3b; Archivo adicional 1:Figura S5). Dado el aumento en el tamaño del parche observado bajo concentraciones de sal más altas (Fig. 3a), parece que las interacciones laterales entre los hexámeros Hoch_5815 son más estables bajo altas concentraciones de sal. Cambios en CaCl 2 La concentración tuvo una respuesta más pronunciada y hubo un cambio significativo en la tasa de eventos dinámicos entre 200 y 300 mM (Fig. 3b), mientras que las respuestas a los cambios en MgCl 2 y KCl son relativamente similares, de acuerdo con los cambios en el tamaño del parche (Fig. 3a). Curiosamente, las proporciones más altas de eventos de montaje frente a eventos de desmontaje se observaron bajo 400 mM de MgCl 2 , CaCl 2 , o KCl (archivo adicional 1:Tabla S2). Esto llevó a la formación de conjuntos Hoch_5815 de una sola capa grandes y estables con sal de 400 mM (Archivo adicional 1:Figura S4). Los ensamblajes de doble capa observados a 500 mM también son estables y exhiben bajas tasas de movimiento de hexámeros.
Flexibilidad del ensamblaje de proteínas BMC-H
Al reducir la fuerza de escaneo a 100 pN, minimizamos los efectos del escaneo de la punta AFM en el ensamblaje de proteínas BMC y obtuvimos imágenes AFM de resolución molecular de hexámeros individuales (Fig. 4). Tanto los eventos de ensamblaje como los de desensamblaje se pueden ver en la misma vista, verificando la naturaleza dinámica de los ensamblajes de shell BMC en lugar de los artefactos de escaneo de punta [12]. Las imágenes de HS-AFM también revelaron la variabilidad de las agregaciones de proteínas Hoch_5815. Al obtener imágenes de las muestras a pH 7,5 en presencia de solo 10 mM de MgCl 2 , sorprendentemente, ocasionalmente observamos la formación de estructuras fibrosas junto con el desmontaje de los hexámeros Hoch_5815 en la segunda escala de tiempo (Fig. 4a). Estas estructuras similares a fibras podrían empaquetarse densamente en paralelo, de forma similar a los haces de nanotubos ensamblados por hexámeros de caparazón [13,14,15,16]. Sin embargo, el espacio entre dos fibras es 3,72 ± 0,31 nm ( n =30) y su altura promedio es de 2,46 ± 0,22 nm ( n =30), menor que la de las láminas de concha formadas por hexámeros Hoch_5815 (3,45 ± 0,16 nm, n =25) (Fig. 4b – f). Estas estructuras de fibra son bastante flexibles y dinámicas durante la generación de imágenes y podrían mostrar arquitecturas rectas o en espiral con diferentes tamaños. Dada la apariencia concomitante de las estructuras de fibra con el desmontaje de los hexámeros Hoch_5815 y su altura reducida en comparación con las hojas de hexámeros de una sola capa, especulamos que estas estructuras similares a fibras están formadas por los péptidos individuales Hoch_5815 desmontados de los hexámeros (Fig. 4g). . Es probable que la absorción del sustrato en condiciones de tampón específicas (como baja fuerza iónica) pueda conducir a la unión de los lados de la hélice alfa de los péptidos Hoch_5815 a la superficie del sustrato y a la unión lineal de los péptidos con péptidos vecinos, aunque se supone que las interacciones intra-hexámeros son probablemente fuertes [5]. El mecanismo detallado que subyace a la variabilidad de la agregación de proteínas de la capa queda por dilucidar más.
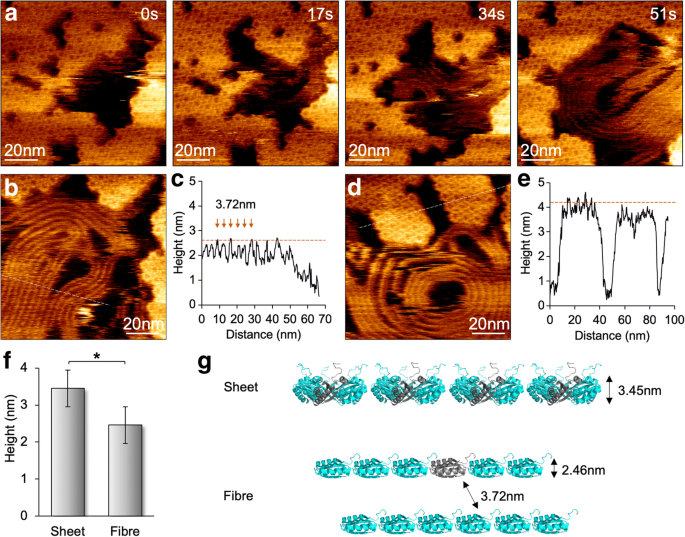
Formación y dinámica de estructuras fibrosas junto con los conjuntos de láminas de concha bajo HS-AFM. un Aparición de estructuras similares a fibras durante el desmontaje de láminas de carcasa compuestas por hexámeros Hoch_5815, como se muestra en la serie de imágenes AFM. b Topografía AFM de estructuras de fibras. c Análisis de sección transversal (línea de guiones en el panel b ) revela un espaciado de 3,72 ± 0,31 nm ( n =30) entre estructuras de fibras adyacentes, y la altura media es 2,46 ± 0,22 nm ( n =30). d Topografía AFM de parches de concha compuestos por hexámeros Hoch_5815. e Análisis de sección transversal (línea de guiones en el panel d ) revela que la altura media de los hexámeros Hoch_5815 es 3,45 ± 0,16 nm ( n =25). f Las estructuras fibrosas presentan una altura reducida en comparación con las láminas planas formadas por hexámeros Hoch_5815 (* p <0,05, ANOVA bidireccional). g Organización y formación propuestas de la estructura similar a una fibra, que representa una cadena de monómeros Hoch_5815
Discusión
Las BMC comprenden cientos de proteínas que se autoensamblan para formar estructuras de orden superior. La capa de BMC, que consta de numerosos homólogos de proteínas, es un sistema ideal para estudiar el autoensamblaje y las interacciones de las proteínas. Como técnica poderosa para analizar la organización de la biomembrana, el ensamblaje de proteínas y las interacciones físicas que son altamente relevantes para las funciones fisiológicas de los sistemas biológicos [32, 35, 38, 39], la AFM se ha aprovechado para visualizar la organización y la dinámica de autoensamblaje de Proteínas de la cáscara de BMC y las arquitecturas y características mecánicas de las estructuras de BMC [12, 30, 31, 40,41,42]. Este trabajo representa, hasta donde sabemos, la primera determinación cuantitativa de la dinámica de autoensamblaje de las proteínas de la cáscara de BMC en la formación de láminas bidimensionales en respuesta a cambios ambientales utilizando AFM. Los resultados destacan la variabilidad inherente y la dependencia ambiental del autoensamblaje de la proteína BMC-H. En comparación con EM y DSL, AFM exhibe un gran potencial para monitorear las acciones dinámicas del autoensamblaje de proteínas BMC en tiempo real con detalles moleculares.
Las interacciones proteína-proteína son de gran importancia en la formación y configuración de la capa de BMC [10]. La concentración de proteínas también se ha documentado como un factor crítico para impulsar la formación de la cáscara [41, 43]. Además, los estudios de solubilidad in vitro han demostrado que el pH y la fuerza iónica en solución podrían influir en la estabilidad estructural de las BMC [17, 27], así como en los comportamientos de ensamblaje de las proteínas de la cubierta de las BMC en la formación de láminas bidimensionales [37, 41]. ], nanotubos [13, 17] y nanocajas [28], que recuerdan su impacto en el ensamblaje de la cápside del virus [44, 45]. También encontramos precipitación de proteínas y no se formaron parches cuando el pH> 10 y <3 o la concentración de sal <10 mM o> 600 mM (datos no publicados). Aquí, mostramos además que la tendencia de ensamblaje y la dinámica dependen del pH y la concentración de sal. Aunque las proteínas de la cáscara pueden autoensamblarse en un amplio rango de pH, el entorno de pH neutro parece ser capaz de mejorar la dinámica de ensamblaje (Fig. 2b). Se encontró que los cationes con una concentración de ≥ 300 mM promueven la formación de láminas bidimensionales; Los cationes 400 mM parecen ser deseables para la formación de láminas de una sola capa grandes y estables (Fig. 3). Estas condiciones se alinean con las condiciones citosólicas de las células bacterianas y son fisiológicamente relevantes. Por ejemplo, en la mayoría de las condiciones fisiológicamente relevantes, el pH de E. coli el citosol es aproximadamente de 7,4 a 7,8 [46] y la concentración de iones es de aproximadamente 100 a 400 mM, que es vital para las interacciones de proteínas, la unión proteína-ligando, la señalización, el mantenimiento de los potenciales electrostáticos de la membrana y el gradiente de proteínas a través de las membranas [47, 48]. Aunque queda por investigar más a fondo cómo las interacciones entre las muestras y el sustrato de mica afectan el autoensamblaje de las proteínas BMC, las imágenes de AFM nos brindan la oportunidad de analizar cuantitativamente los cambios dinámicos del autoensamblaje de las proteínas BMC en respuesta a las variaciones ambientales.
La dinámica de ensamblaje dependiente del medio ambiente de las proteínas BMC en la formación de fragmentos de capa que se describen aquí podría representar sus comportamientos en la formación de todo el BMC. De hecho, las estructuras 3D BMC parecen ser los orgánulos mantenidos dinámicamente diseñados en la naturaleza. Los BMC presentan una notable flexibilidad y heterogeneidad estructural; la suavidad mecánica de las estructuras de caparazón de BMC determinadas por nanoindentación de AFM [30] y la dinámica de desequilibrio del ensamblaje de BMC revelada por simulaciones computacionales [49] destacaron las diferencias entre BMC y ensamblajes de virus robustos. Asimismo, se ha dilucidado que la biosíntesis de carboxisomas se correlaciona con la luz y los acompañantes [50, 51]. Muy recientemente, se ha indicado que CcmK3 y CcmK4 pueden formar heterohexámeros y casquete en la capa carboxisoma de una manera dependiente del pH, posiblemente proporcionando un medio para regular la permeabilidad de la capa carboxisoma y el CO 2 asimilación en el microambiente altamente dinámico [52]. El mecanismo exacto subyacente a cómo las condiciones ambientales en solución afectan el ensamblaje termodinámico de las proteínas BMC aún no se ha investigado, por ejemplo, utilizando una combinación de estudios experimentales y simulaciones computacionales.
Dado el autoensamblaje de estructuras de BMC, existe un interés significativo en la ingeniería de BMC y el diseño de nuevos nanobiorreactores, andamios moleculares y biomateriales basados en BMC en aplicaciones de biotecnología, por ejemplo, mejora del metabolismo celular, encapsulación de enzimas, administración molecular y terapia. . El conocimiento avanzado sobre la resiliencia estructural y la variabilidad de las BMC en respuesta a los cambios ambientales no solo informará las estrategias para producir nanoestructuras robustas basadas en BMC en huéspedes heterólogos, es decir, E. coli o plantas [31, 53, 54], sino que también allanan el camino para modular la formación de nanomateriales 2D, así como la apertura y cierre de las jaulas proteicas basadas en cáscaras de BMC, facilitando así la regulación funcional y la entrega molecular dirigida. Previamente, hemos demostrado la viabilidad de utilizar un enfoque de modificación genética para manipular los contactos específicos en las interfaces de las proteínas de la cáscara y sus comportamientos de autoensamblaje [12]. Este estudio refuerza nuestra caja de herramientas para evaluar y manipular el autoensamblaje del shell BMC en diferentes entornos.
Conclusiones
En resumen, aprovechamos HS-AFM para llevar a cabo las investigaciones cuantitativas del autoensamblaje de la proteína del caparazón de BMC en diferentes condiciones de pH y sal. Se demostró que la formación de parches de una sola capa más grandes de hexámeros de caparazón se promueve a una concentración de sal de 400 mM, y el pH neutro dio como resultado una mayor velocidad dinámica de autoensamblaje de hexámeros. También se visualizó la transición organizativa de las proteínas de la cáscara de conjuntos hexámeros a matrices similares a fibras. Este estudio ilustró que las condiciones ambientales juegan un papel importante en la determinación de la organización y el autoensamblaje de las proteínas del caparazón de BMC.
Abreviaturas
- BMC:
-
Microcompartimento bacteriano
- BMC-H:
-
Hexámero de microcompartimento bacteriano
- BMC-P:
-
Pentámero de microcompartimento bacteriano
- BMC-T:
-
Trímero de microcompartimento bacteriano
- DSL:
-
Dispersión de luz dinámica
- E. coli :
-
Escherichia coli
- EM:
-
Microscopía electrónica
- HS-AFM:
-
Microscopía de fuerza atómica de alta velocidad
Nanomateriales
- Evaluación de IoT y el impacto de 5G
- Innovación en redes privadas:configure y cambie el ancho de banda y la duración sobre la marcha
- Presentación de la estructura atómica y electrónica de las nanofibras de carbono de copa apilada
- Estudio de los primeros principios sobre la estabilidad y la imagen STM del borofeno
- Síntesis fácil de óxido de estaño mesoporoso similar a un agujero de gusano a través del autoensamblaje inducido por evaporación y las propiedades mejoradas de detección de gas
- Prueba de las propiedades estructurales, electrónicas y magnéticas de Ag n V (n =1–12) Clusters
- Fabricación y caracterización de nanoclips de ZnO mediante el proceso mediado por poliol
- Robots, cobots y automatización:¿la pandemia está impulsando una ola de cambios en la fabricación?
- Cómo el repunte de la fabricación cambiará el reclutamiento y la retención de empleados
- El impacto ambiental de la fabricación desde la revolución industrial hasta la automatización y todo lo demás
- ¿La automatización y los robots cambiarán el mundo?



