Control de la translocación del ADN a través de nanoporos de estado sólido
Resumen
En comparación con el estado de los bio-nanoporos, todavía hay varios desafíos que deben superarse antes de que los nanoporos de estado sólido puedan aplicarse en la secuenciación comercial de ADN. La baja resolución espacial y temporal son los dos principales desafíos. Debido a las restricciones en la longitud de los nanoporos y las propiedades superficiales de los nanoporos de estado sólido, todavía hay espacio para mejorar la resolución espacial. Mientras tanto, la translocación del ADN es demasiado rápida bajo una fuerza eléctrica, lo que da como resultado la adquisición de pocos puntos de datos válidos. Por tanto, la resolución temporal de los nanoporos en estado sólido podría mejorarse si la velocidad de translocación del ADN está bien controlada. En esta mini revisión, resumimos brevemente los métodos para mejorar la resolución espacial y nos concentramos en métodos controlables para promover la resolución de la detección de nanoporos. Además, proporcionamos una perspectiva sobre el desarrollo de la secuenciación de ADN por nanoporos.
Introducción
En las últimas décadas, se ha avanzado mucho en la aplicación de la secuenciación del ADN para leer las secuencias de bases en el genoma [1, 2]. Para desarrollar medicamentos personalizados, los investigadores han estado buscando un método de secuenciación de ADN más rápido y económico, en el que los medicamentos objetivo y los tratamientos médicos se puedan aplicar a un individuo específicamente [3, 4]. Debido a que la tecnología de nanoporos se utilizó en la detección de ADN [5], se consideró un método eficaz para la secuenciación de próxima generación [6, 7]. La tecnología Nanopore es una plataforma prometedora para identificar detectores de molécula única altamente sensibles para ADN [5] o ARN [8]. En el esquema de detección básico, una cámara electroquímica se separa en dos depósitos (compartimentos cis y trans) mediante una fina membrana con un nanoporo [9], que conecta la solución conductora y el analito en la cámara electroquímica. Al aplicar un voltaje a través de la membrana, los iones de electrolito fluyen a través del nanoporo de estado sólido y forman una corriente de poro, que se mide utilizando una configuración de abrazadera de parche con componentes electrónicos ultrasensibles asociados. Cuando una molécula o complejo molecular pasa a través del nanoporo, el analito puede excluir algunos iones del volumen definido por el nanoporo, que pueden detectarse monitoreando cambios breves en la corriente. Tanto del tiempo de residencia (tiempo de permanencia) dentro del nanoporo como de la firma de amplitud actual, se puede obtener información sobre las moléculas. La resolución espacial de la secuenciación de nanoporos está determinada por las dimensiones del nanoporo, lo que sugiere que se puede usar como un sensor único para pequeños objetos moleculares que dan como resultado una firma de corriente detectable. Además, los sensores de nanoporos son fáciles de integrar en dispositivos de laboratorio en un chip altamente portátiles y miniaturizar [10].
Se han logrado avances significativos en la secuenciación del ADN mediante nanoporos, como los nanoporos de estado sólido [2, 11] y los nanoporos de proteínas [2, 7]. Se ha logrado la secuenciación del ADN mediante nanoporos de proteínas [7]. Sin embargo, los sistemas de nanoporos de proteínas tienen límites para el estudio de moléculas biológicas. Hay menos limitaciones con los nanoporos de estado sólido en comparación con los nanoporos biológicos / proteicos. Estas dificultades pueden superarse mediante nanoporos en estado sólido. En comparación con los nanoporos de proteínas, son funcionales en rangos más amplios de temperaturas, voltajes y condiciones de solvente y se pueden ajustar en diámetro con precisión subnanométrica. Son prometedores para su aplicación en tecnología de próxima generación para secuenciación de ADN.
Se han fabricado muchos nanoporos de estado sólido de diferentes materiales y estructuras para sensores de ADN. Sin embargo, la secuenciación del ADN no se logra mediante nanoporos en estado sólido. Para los sensores de nanoporos de estado sólido, existe la necesidad de superar dos obstáculos principales, en cuanto a su resolución espacial y resolución temporal, antes de su aplicación comercial a la secuenciación de ADN. La dificultad con respecto a la resolución espacial es que el nanoporo de estado sólido puede distinguir el pequeño espacio entre dos nucleótidos vecinos para lograr una resolución de base única. El obstáculo de la resolución temporal es que la translocación del ADN es demasiado rápida bajo una fuerza eléctrica, lo que da como resultado la adquisición de muy pocos puntos de datos válidos mediante pinzas de parche u otros sistemas de adquisición de señales existentes. Esta mini revisión presenta una descripción general de los diversos métodos para mejorar la resolución espacial y la resolución temporal de la detección de ADN de nanoporos en estado sólido. Esta mini revisión también se centra más en los métodos para ralentizar la translocación del ADN a través de nanoporos de estado sólido.
Resolución espacial
En 2001, Li et al. [12]. Se han demostrado varios nanoporos de estado sólido para la detección molecular de ADN, como óxido de silicio [13], silicio [14], Al 2 O 3 [15] y HfO 2 [dieciséis]. Si bien los nanoporos de estado sólido pueden, en última instancia, ser resistentes a las condiciones químicas y mecánicas, tienen algunas limitaciones, como una baja resolución espacial. Debido al grosor de los materiales, docenas de bases pueden atravesar nanoporos de estado sólido a la vez. En la actualidad, el nanoporo de nitruro de silicio más delgado es de 3 nm, lo que aún no distingue los cuatro tipos de base [17].
Curiosamente, el grosor de una sola capa de material bidimensional (2D) es de aproximadamente 3,0-11,0 Å, que es comparable al espaciado entre dos nucleótidos vecinos a lo largo de ssDNA (3,2-5,2 Å) [18]. Membranas bidimensionales, como grafeno (3,4 Å [19]), MoS 2 (6,5 Å [18]), WS 2 Se ha demostrado que (7 Å [20]) y h-BN (11 Å [21]) detectan translocaciones de ADN [21, 22, 23] debido a su alta relación señal-ruido y resolución espacial. Está claro por su resolución espacial que esos materiales pueden usarse para la detección de ADN. Además, las técnicas de crecimiento reproducibles y los procedimientos de transferencia a gran escala hacen posible la fabricación de poros subnanométricos en membranas 2D a gran escala.
El grafeno es una hoja atómicamente delgada de átomos de carbono dispuestos en una red bidimensional en forma de panal [24]. Los investigadores han demostrado que se pueden detectar y caracterizar moléculas de ADN individuales en solución con nanoporos de grafeno [22, 25]. Sin embargo, existen fuertes interacciones hidrofóbicas entre los nucleótidos del ADN y el grafeno, y el ADN se obstruirá y adherirá severamente a los nanoporos del grafeno, lo que impactaría notablemente en la velocidad de translocación [26]. Modificado con los grupos hidrófilos [26] o recubierto, un material hidrófilo [25] sobre grafeno podría mejorar la hidrofilia del grafeno y evitar que el ADN se adhiera a su superficie. Desafortunadamente, ya sea la modificación con grupos hidrófilos o el recubrimiento con materiales hidrófilos aumentará el espesor de la película suspendida, lo que conducirá al aumento del espesor de los nanoporos, disminuyendo así la resolución espacial del grafeno.
Dicalcogenuro de metal de transición en capas es otro material 2D, que incluye MoS 2 [18, 27] y WS 2 [20]. Se detectó una alta relación señal / ruido (SNR> 10) y una mejora de cinco veces la señal de corriente iónica cuando el ADN se translocó a través del MoS 2 membrana de nanoporos [18]. Mientras tanto, MoS 2 es hidrófilo, por lo que no se necesita ningún tratamiento de superficie especial para evitar la interacción hidrófoba entre el ADN y su superficie. El otro material, WS 2 tiene una banda prohibida directa de 2,1 eV [28], y su emisión de fotoluminiscencia (PL) es más fuerte que la del bien estudiado MoS 2 [29]. Danda y col. [20] fabricó un WS 2 nanopore y demostró el logro de un tamaño de nanoporo controlado atómicamente utilizando pulsos de luz cortos, que pueden tener efectos positivos en los nanoporos de estado sólido para la detección de ADN.
Además, Liu et al. [21] informó sobre el primer experimento de translocación de ADN a través de nanoporos de h-BN. Al igual que el grafeno, el h-BN tiene poca hidrofilicidad, lo que hará que el ADN bloquee los nanoporos. Posteriormente, Zhou et al. [23] mejoró con éxito la hidrofilia de los nanoporos de h-BN aprovechando la antioxidación y la integridad del material de h-BN después del tratamiento con ozono UV. La naturaleza aislante de h-BN puede exhibir una durabilidad y propiedades aislantes más notables en una solución de alta fuerza iónica que en el grafeno. Es un candidato competitivo para lograr una resolución de base única en estructuras de nanoporos superdelgadas.
El uso de materiales bidimensionales puede aumentar potencialmente la resolución espacial del dispositivo para lograr una resolución de un solo nucleótido. Aunque se han informado experimentos de detección de ADN para varios materiales bidimensionales, nadie ha informado del logro de la secuenciación de ADN de nanoporos en estado sólido. La resolución temporal de la secuenciación de nanoporos también es un desafío.
Resolución temporal
La velocidad de translocación del ADN a través de un nanoporo de estado sólido es muy rápida, de hasta 0,01-1 μs / base [30], lo que lleva a muy pocos datos efectivos recopilados por los parches de sujeción comerciales. Como tal, no es posible distinguir todas las bases en función de la señal de corriente de bloqueo. Las velocidades de translocación del ADN de nanoporos de estado sólido de materiales 2D, como el grafeno, MoS 2 , WS 2 , y h-BN, se muestran en la Fig. 1. Idealmente, la velocidad de translocación del ADN en un nanoporo debe ser de 1 a 100 pb / ms para permitir un registro satisfactorio de la señal de cada nucleótido [32].
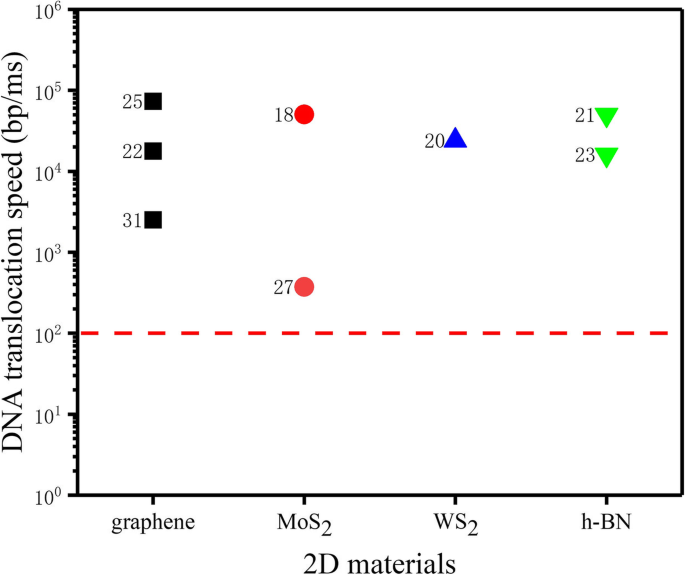
Velocidad de translocación del ADN de nanoporos de estado sólido 2D [18, 20,21,22,23, 25, 27, 31]. La línea roja punteada indica una tasa de translocación de ADN de 100 pb / ms
En este contexto, ralentizar la velocidad de translocación del ADN es un objetivo importante perseguido por muchos investigadores. Se han desarrollado varios métodos para ralentizar la translocación del ADN y mejorar la resolución temporal de la detección de nanoporos en estado sólido. El método habitual consiste en cambiar la influencia de factores experimentales como la temperatura [33], la viscosidad del electrolito [27], el voltaje de activación [34], la concentración de iones [35] y la densidad de carga superficial de un nanoporo [36] cambiando el ADN translocación a través de él. Wanunu y col. [33] se concentró en ralentizar la translocación del dsDNA a través de nanoporos de estado sólido cambiando la temperatura, el voltaje y la longitud del ADN. Además, Feng et al. [27] mostró que un sistema de gradiente de viscosidad, basado en líquidos iónicos a temperatura ambiente, se puede utilizar para controlar la dinámica de la translocación del ADN a través de MoS 2 nanoporos, y demostró que la alta viscosidad de los líquidos iónicos a temperatura ambiente proporciona velocidades óptimas de translocación de un solo nucleótido de 373 bp / ms.
Aunque muchos enfoques tienen el potencial de reducir la velocidad de translocación del ADN y facilitar la detección de corriente iónica, todavía no pueden cumplir con los requisitos para la secuenciación del ADN. Por tanto, es necesario desarrollar métodos más radicales para controlar el paso del ADN a través de los nanoporos. Aquí, discutimos los métodos para controlar la estructura de nanoporos y el movimiento cuantitativo del ADN para mejorar la resolución temporal.
Sistema de dos nanoporos
Los investigadores han utilizado sistemas de dos nanoporos para manipular las translocaciones de moléculas de ADN, que pueden detectar de forma controlable la misma molécula muchas veces. Dos nanoporos apilados, separados por un compartimento de cavidad del tamaño de un micrómetro [37] (Fig. 2a), pueden atrapar moléculas de ADN durante un cierto período de tiempo y liberarlas de forma controlada. La dinámica de las moléculas de ADN se puede deducir de las señales de los dos poros mediante un análisis de correlación, que proporciona una prueba eléctrica directa de la translocación. Además, debido a las barreras de entropía del sistema de nanoporos de dos capas, el movimiento browniano de las moléculas de ADN puede limitarse, lo que puede mejorar la precisión de la secuenciación del ADN con nanoporos. En comparación con las técnicas de detección de una sola molécula, las moléculas de ADN se pueden medir varias veces en sistemas de nanoporos de dos capas colocando en serie varios poros en lugar de pasarlos de un lado a otro a través de un solo poro [39].
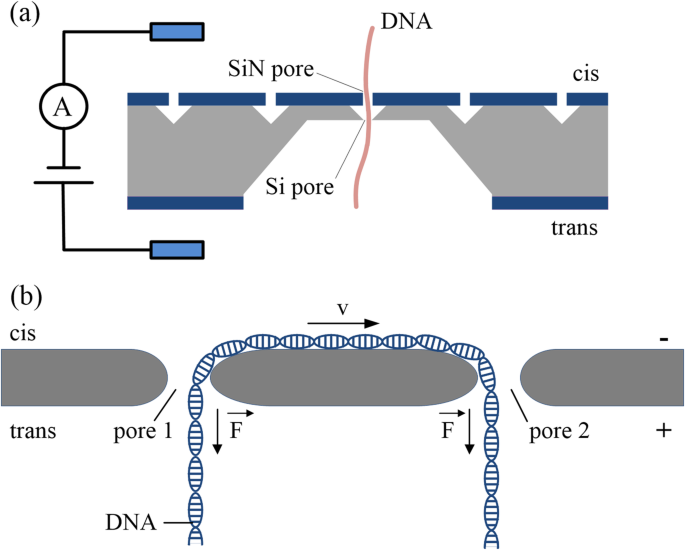
un Esquema del sistema de nanoporos de dos capas utilizado para la detección de ADN de nanoporos [37]. b Esquema del sistema de nanoporos dobles utilizado para la detección de ADN de nanoporos [38]
Los sistemas de nanoporos dobles proporcionan otro método para controlar el transporte molecular y unir moléculas de manera eficiente entre dos poros; son un enfoque mecanicista sin etiquetas para la manipulación del ADN [40]. En un sistema de nanoporos dobles, hay dos nanoporos adyacentes y direccionables de forma independiente dentro de la misma membrana de estado sólido. Durante el paso electroforético de una molécula de ADN a través de uno de los nanoporos, se puede capturar una sola molécula de ADN en ambos poros, lo que lleva a un "tira y afloja" entre los dos nanoporos [38] (Fig. 2b). Por lo tanto, se aplican fuerzas a los diferentes extremos de una molécula de ADN, lo que ralentiza e incluso detiene por completo su movimiento. El sistema de doble nanoporo abre un nuevo camino hacia la captura mecánica de ADN en nanoporos de estado sólido, y es una técnica prometedora para medir una amplia gama de biomoléculas con las ventajas de no tener etiquetas y tener una alta señal. relación ruido y bajo costo. Puede confinar y atrapar de manera eficiente las moléculas de ADN para ralentizar la translocación del ADN y también se puede utilizar para estudiar la física de este tira y afloja a nanoescala del ADN [41].
Nanopore de trampa óptica
Los nanoporos de trampa óptica permiten que las pinzas ópticas atrapen partículas de menos de decenas de nanómetros de tamaño. Hace posible la captura óptica de proteínas [42], fragmentos de ADN [43] y otras biomoléculas [44], así como pequeños virus [45]. La teoría básica detrás de los nanoporos de trampas ópticas es una trampa óptica de acción inversa autoinducida [46]. El rayo láser enfocado en la región de la matriz de nanoporos formará un campo de luz local de alta densidad de potencia en el borde de la capa de metal en el agujero. A medida que una partícula se mueve entre el campo de luz local, puede provocar un gran cambio en la transmisión óptica local, que a su vez producirá una gran fuerza óptica y dieléctrica sobre la partícula. Se utiliza una estructura de doble nanoagujero para romper el cuello de botella de tamaño de las partículas capturadas. Muhammad y col. [47] demostró el uso potencial de nanoporos trampa óptica con nanopartículas de sílice y Au de 20 nm. La forma de mancuerna del nanoagujero doble se molió en la película de Au y se perforó un nanoporo de 25 nm a través del Si x suspendido N y membrana en el medio del doble nanoagujero, como se muestra en la Fig. 3a. La fuerza electroforética que impulsa las nanopartículas a través del nanoporo, al pasar por el borde del agujero, la fuerza plasmónica de acción inversa autoinducida existente entre las puntas del nanoagujero doble se opuso a la fuerza electroforética, reduciendo la velocidad de las nanopartículas. Los resultados mostraron que el atrapamiento óptico extendió el tiempo de translocación electroforética en cuatro órdenes de magnitud. Kim y col. [43] realizó la captura óptica de ADN plasmídico y ADN lambda mediante el uso de estructuras nanoplásmicas de nanoporos individuales. La tecnología tiene aplicaciones potenciales para la detección de ADN y se pueden configurar múltiples campos de luz local para realizar una detección en paralelo. Sin embargo, la oscilación de la corriente de iones de alta frecuencia puede afectar los resultados de detección de corriente del ADN. Esto puede deberse a la competencia entre las fuerzas de retroacción electroforéticas y autoinducidas que hacen que las nanopartículas floten hacia arriba y hacia abajo a través de la boca del nanoporo.
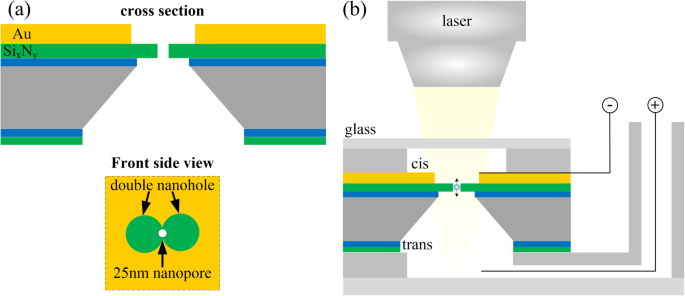
un Esquema del chip de nanoporos de trampa óptica [47]. b Esquema del sistema de retroacción autoinducido
Pinzas ópticas
Las pinzas ópticas se pueden utilizar para controlar la translocación molecular a través de nanoporos y se han utilizado comúnmente en los últimos años. En 2006, Keyser et al. [48] demostró por primera vez un tira y afloja molecular entre las fuerzas electroforéticas y mecánicas mediante la aplicación de pinzas ópticas para controlar la translocación del ADN a través de SiN x nanoporos. Este sistema actúa como un simple resorte Hooke, y la fuerza de tensión en el cordón se puede calcular según la ley de Hooke: F ot =−k trampa Z , donde F ot es la fuerza óptica, k trampa es la rigidez de la trampa a lo largo de la dirección de desplazamiento, y Z es la deformación lineal del cordón [48]. El método de pinzas ópticas, que atrapa una perla de poliestireno atada al ADN en el cruce de un rayo láser enfocado, puede manipular la perla de poliestireno atada al ADN en tres dimensiones y tiene un rango de pico-newton de sensibilidad a la fuerza, como se muestra en la Fig. 4a . Para atrapar el ADN translocado dentro del nanoporo, la fuerza de tensión se ajustó para equilibrar la fuerza del campo eléctrico ( F el ) en el ADN. Por lo tanto, la tensión se puede utilizar tanto para reducir la velocidad de translocación del ADN como para extraer la molécula de ADN de un nanoporo [52]. Este sistema permite un muestreo espacial simultáneo y mediciones de fuerza de alta resolución de ácidos nucleicos y proteínas, lo que ha logrado un progreso significativo en la secuenciación del ADN. Sin embargo, la técnica de las pinzas ópticas adolece de varias dificultades fundamentales. Primero, es difícil escalar hasta una gran cantidad de nanoporos. En segundo lugar, el calentamiento causado por el láser en las pinzas ópticas impacta fuertemente en la corriente iónica a través de un nanoporo y el nivel de ruido, lo que requiere que la perla atrapada ópticamente esté a varios micrómetros de distancia del nanoporo [53].
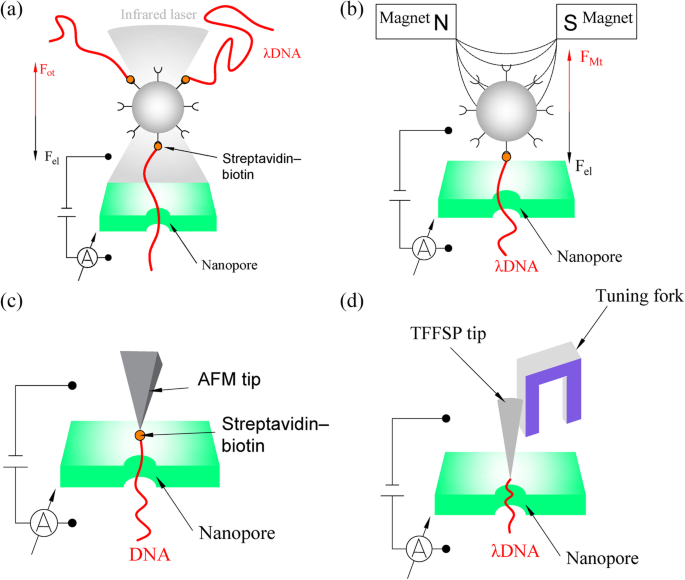
un Esquema de pinzas ópticas utilizadas para la detección de ADN por nanoporos [48]. Cuando el sistema de pinzas ópticas está en equilibrio, la fuerza óptica (F ot ) es igual a la fuerza del campo eléctrico (F el ). b Esquema de pinzas magnéticas utilizadas para la detección de ADN por nanoporos [49]. Cuando el sistema de pinzas magnéticas está en equilibrio, la fuerza magnética (F Mt ) es igual a la fuerza del campo eléctrico (F el ). c Esquema de AFM utilizado para la detección de ADN de nanoporos [50]. d Esquema de TFFS utilizado para la detección de ADN de nanoporos [51]
Pinzas magnéticas
Las pinzas magnéticas proporcionan otra forma de controlar la translocación del ADN mediante la fuerza de tensión, y se ha demostrado que la técnica de las pinzas magnéticas es eficaz para ralentizar la translocación del ADN [49]. En este sistema, como se muestra en la Fig. 4b, las moléculas de ADN se pueden unir a una perla magnética del tamaño de un micrómetro utilizando una fuerte interacción oro-tiol [54] o estreptavidina-biotina [49]. Luego, el extremo libre del ADN se puede capturar en el nanoporo mediante un campo eléctrico aplicado. Posteriormente, se pueden usar dos imanes con un pequeño espacio para crear un gradiente de campo magnético. Esta técnica puede equilibrar la fuerza eléctrica sobre el ADN atrapado para reducir las velocidades de translocación e incluso la electroforesis inversa. En comparación con las pinzas ópticas, las pinzas magnéticas son un candidato prometedor para la espectroscopia de fuerza masivamente paralela. En este sistema, cientos de perlas y, por lo tanto, moléculas de ADN se pueden controlar simultáneamente dentro de cientos de nanoporos, que es fácilmente escalable a muchos nanoporos direccionables. Esto puede acelerar el proceso analítico en órdenes de magnitud. Sin embargo, en comparación con las pinzas ópticas, una desventaja obvia del enfoque de las pinzas magnéticas es la falta de control tridimensional de las moléculas [55].
Sonda de detección de fuerza
Aunque se ha logrado un progreso significativo en el control de la velocidad de translocación del ADN mediante pinzas ópticas y pinzas magnéticas, los métodos de atrapamiento de perlas tienen un problema con el movimiento browniano que dificulta el control del movimiento de la perla con una resolución de menos de 10 nm [51] . Para superar esto, se ha utilizado AFM para controlar la velocidad de translocación del ADN [50], y también puede medir simultáneamente la fuerza y la corriente de bloqueo. En un estudio que utilizó este sistema, como se muestra en la Fig. 4c, el ADN se ató a la punta de una sonda AFM y luego se fijó en el soporte de la sonda. Controlando el movimiento de la sonda, la translocación del ADN podría controlarse para reducir su velocidad e incluso invertir la electroforesis. Además, al retraer la punta a una altura por encima de la superficie correspondiente a la longitud de la molécula, se pueden repetir las mediciones. Con la ayuda de AFM, la detección de ADN ha avanzado en la práctica y la teoría porque la resolución de detección puede mejorarse en gran medida mediante las señales combinadas de la corriente de bloqueo y las mediciones de fuerza de AFM [56]. Sin embargo, todavía existe un obstáculo en la aplicación de técnicas de nanoporos en la detección de ADN, a saber, la ocurrencia intermitente de fluctuaciones regulares en la fuerza (y la corriente) cada 0.35-0.72 nm cuando una molécula de ADN se desliza de una manera relativamente sin fricción a través de un nanoporo. Estas fluctuaciones se atribuyen a los nucleótidos individuales que se trasladan a través del nanoporo en un movimiento similar a un torniquete [50].
Los estudios han demostrado que un diapasón, que puede utilizarse como sensor de detección de fuerza, puede controlar el ADN para que pase a través de un nanoporo a una velocidad subnanométrica [51, 57]. En un estudio que utilizó este aparato integrado, como se muestra en la Fig. 4d, se unió una molécula de ADN a la punta de una sonda que estaba pegada a una de las puntas de un diapasón. Se utilizó un sistema de nanoposicionamiento, que posee una precisión subnanométrica, para sujetar el diapasón [51]. La posición de la punta de la sonda puede detectarse mediante el sensor de fuerza de retroalimentación basado en el diapasón y controlarse manipulando el sistema de posicionamiento nanométrico. Esta velocidad de movimiento es 10 veces más lenta que la del ADN manipulado con pinzas ópticas y 1000 veces más lenta que el ADN que pasa libremente a través de nanoporos de estado sólido [57]. En comparación con el AFM convencional, un diapasón puede proporcionar un movimiento de exploración más rápido y poseer una alta sensibilidad a la fuerza cuando se sumerge en el líquido. Al incorporar TFFS con un nanoporo, la corriente iónica a través de un nanoporo, la posición de la punta y la amplitud vibratoria de la punta se pueden medir simultáneamente durante el paso de una molécula de ADN a través del nanoporo.
Sonda Si
Todos los métodos anteriores, a saber, pinzas magnéticas, pinzas ópticas, AFM y TFFS, necesitan escanear la posición del nanoporo. Tienen que ubicar el nanoporo dentro de la longitud efectiva del ADN para asegurarse de que el ADN pueda pasar a través del nanoporo. El direccionamiento de nanoporos es una parte importante de estos métodos, pero es difícil [32] conectar el ADN a una gran superficie de una sonda de silicio (Fig. 5), más grande que el área del chip, lo que significaba que podían insertar fácilmente el ADN inmovilizado en un nanoporo sin escanear la ubicación del nanoporo en la membrana. Se ha demostrado la viabilidad de utilizar una sonda de Si con ADN inmovilizado y un controlador de posición para controlar el movimiento del ADN dentro y fuera de los nanoporos. La dificultad de este método es que la sonda de Si se sumerge en la solución y se conecta al ADN mediante el acoplamiento de péptidos. La densidad del ADN en la superficie de la sonda es difícil de controlar, por lo que es probable que ocurran varios ADN que atraviesan un nanoporo al mismo tiempo, lo que afecta la corriente de detección.
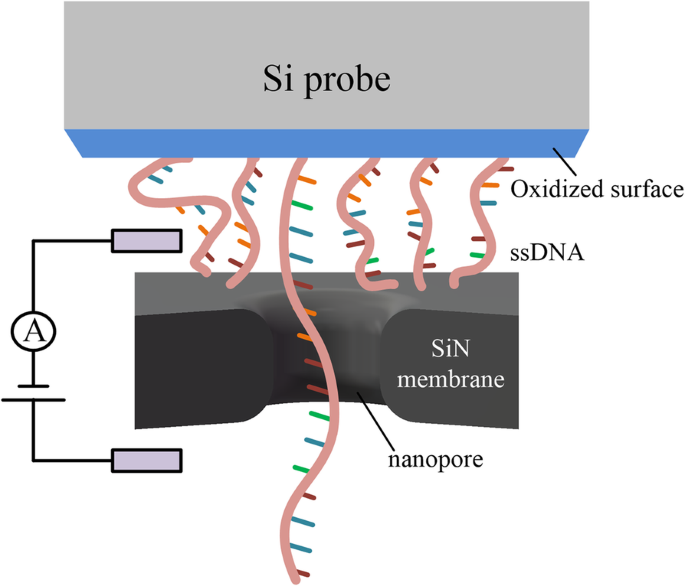
Esquema del sistema de sonda de Si inmovilizado con ADN utilizado para la detección de ADN con nanoporos [32]
Pinzas ópticas, pinzas magnéticas, microscopía de fuerza atómica (AFM) y detección de fuerza basada en diapasón (TFFS) pueden detectar las fuerzas reales y la posición de la molécula en el nanoporo, que promete controlar el paso del ADN a través de los nanoporos con la velocidad adecuada. La dificultad de abordar los nanoporos se evita mediante el uso de una sonda de Si. Además, el uso de un sistema de dos nanoporos es un método factible para controlar y ralentizar el paso del ADN a través de los nanoporos. Además, un nanoporo de trampa óptica tiene potencial para la detección de ADN en el futuro. Aquí, resumimos la velocidad de translocación del ADN de un nanoporo de estado sólido integrado con algunos métodos de control de ADN, como un sistema de dos nanoporos, un nanoporo de trampa óptica, pinzas ópticas, pinzas magnéticas, AFM, TFFS y sonda de Si (Tabla 1) .
Conclusión
Materiales 2D monocapa, como grafeno, MoS 2 , WS 2 , y h-BN, son posiblemente los materiales más delgados que se pueden conseguir, ya que son tan gruesos como el espacio entre los nucleótidos. En comparación con las membranas de nanoporos de estado sólido tradicionales, las membranas 2D monocapa son ideales para dispositivos de nanoporos, ya que exhiben una alta relación señal / ruido de corriente iónica y regiones de detección relativamente grandes. Son potencialmente elegibles para realizar la secuenciación de ADN mediante la combinación con pinzas ópticas, pinzas magnéticas, AFM, TFFS, sonda de Si, sistema de dos nanoporos o nanoporos de trampa óptica. Sin embargo, con estas técnicas, han surgido varios desafíos, que deben resolverse antes de la comercialización de la secuenciación de ADN por nanoporos. El primero de ellos ocurre cuando las perlas o puntas de la sonda están cerca de un nanoporo, cuando es más difícil discriminar nucleótidos de ADN con señales de corriente iónica. Deben usarse asas moleculares u otras moléculas más largas para agregar la longitud de una hebra de ADN que pueda contrarrestar el efecto sobre la señal actual provocado por perlas o puntas. En segundo lugar, las matrices de nanoporos deben usarse para realizar una detección paralela y de alto rendimiento, pero la tecnología de detección paralela no está lo suficientemente madura en la actualidad. En tercer lugar, de acuerdo con los métodos de fabricación actuales, es difícil fabricar un sistema de dos nanoporos y un sistema de trampa óptica de nanoporos con alta precisión y reproducibilidad, lo cual es muy importante para la detección de ADN de nanoporos. Un haz de iones de helio puede ser la tecnología clave para resolver este problema [11, 58]. Por lo tanto, esperamos que la secuenciación de nanoporos de ADN continúe siendo un foco de investigación y pueda integrarse con más ideas nuevas y enfoques innovadores para lograr tasas de error bajas, registros paralelos rápidos y altos y longitudes de lectura largas de hasta 100 kilobases.
Disponibilidad de datos y materiales
Todos los datos generados o analizados durante este estudio se incluyen en este artículo publicado.
Abreviaturas
- 2D:
-
Dos dimensiones
- 3D:
-
Materiales tridimensionales
- TMD:
-
Dicalcogenuro de metal de transición en capas
- SNR:
-
Relación señal / ruido
- PL:
-
Fotoluminiscencia
- SIBA:
-
Acción de espalda autoinducida
- AFM:
-
Microscopía de fuerza atómica
- TFFS:
-
Detección de fuerza basada en diapasón
Nanomateriales
- Relés de estado sólido
- Síntesis de ADN
- Amazon Alexa Controlando un Chromecast
- Mejorar mediante la supervisión inalámbrica
- Lidera escuchando
- Eliminación de defectos mediante la confiabilidad del equipo
- Blog:Detección de genes a través de microarrays
- Los científicos de IBM miden la transferencia de calor a través de átomos individuales
- Protección de IoT mediante engaños
- Recubrimiento de orificios
- Sensor de dióxido de carbono de estado sólido



