Detección virtual guiada por farmacóforos para la reutilización de fármacos
La reutilización de fármacos es una estrategia establecida desde hace mucho tiempo para identificar nuevas aplicaciones de terapias aprobadas o en investigación que están más allá del alcance de la indicación médica original. 1 Desde el reposicionamiento fortuito de un medicamento diseñado para la hipertensión y la angina de pecho para convertirse en la infame "pequeña píldora azul" de Pfizer, comúnmente conocida como Viagra, la reutilización de medicamentos no ha sido tan prominente en el ojo público. La urgencia global de las terapias para tratar el COVID-19 ha traído esta estrategia farmacológica una vez más a la atención masiva.
El modelado de proteínas en 3D incluye varios métodos de modelado y simulación molecular para la reutilización de fármacos. Una publicación anterior de esta serie exploró el acoplamiento molecular como un método para identificar posibles inhibidores que se unen a la proteasa principal del SARS-CoV-2. ¿Puede una alternativa in silico ¿Un método como el modelado farmacóforo logra resultados comparables y proporciona evidencia adicional que respalda la selección de un candidato sobre otro?
Tomar una ruta alternativa
El modelado de farmacóforos proporciona una abstracción de las características moleculares que son necesarias para el reconocimiento de un ligando por una proteína diana. Su representación de interacciones y enlaces moleculares proporciona una perspectiva contrastante con los métodos de simulación clásicos.
Obtuvimos un conjunto de datos inicial de varias estructuras proteicas principales de proteasa del SARS-CoV-2 de Diamond Light Source. 2 Alineamos estas estructuras y luego usamos el protocolo "Interaction Pharmacophore Generation" disponible en BIOVIA Discovery Studio para generar farmacóforos que representan las interacciones sin enlace de cada complejo receptor-ligando. El número total de características en los farmacóforos varió desde dos características para las estructuras cristalinas 5R80 y 5R7Y hasta un modelo de nueve características para 6LU7. Fusionamos los farmacóforos de los complejos individuales en un solo modelo y editamos las características agrupadas de cerca. El modelo final incluyó 14 características que representan contactos intermoleculares entre la proteasa y posibles aglutinantes de moléculas pequeñas.
Posteriormente realizamos in silico mutagénesis de exploración de alanina en los residuos del sitio activo de todos los complejos para identificar qué residuos reducen la afinidad de unión (punto caliente) de ese complejo proteína-ligando cuando muta. De todos los complejos, identificamos ocho residuos (HIS41, MET49, ASN142, HIS163, MET165, PRO168, GLN189 y GLN192) como puntos calientes en al menos un complejo y tres residuos que eran puntos calientes en al menos cuatro complejos. En el modelo de farmacóforo de 14 características, seis características se correspondían con interacciones con uno de los ocho residuos de hotspot. Un segundo conjunto de datos de M PRO no covalente complejos de ligandos liberados por Diamond Light Source 2 reveló una serie de modos de unión diferentes, lo que nos llevó a separar las seis características farmacóforos clave en dos grupos para su uso en el siguiente paso de la detección virtual.
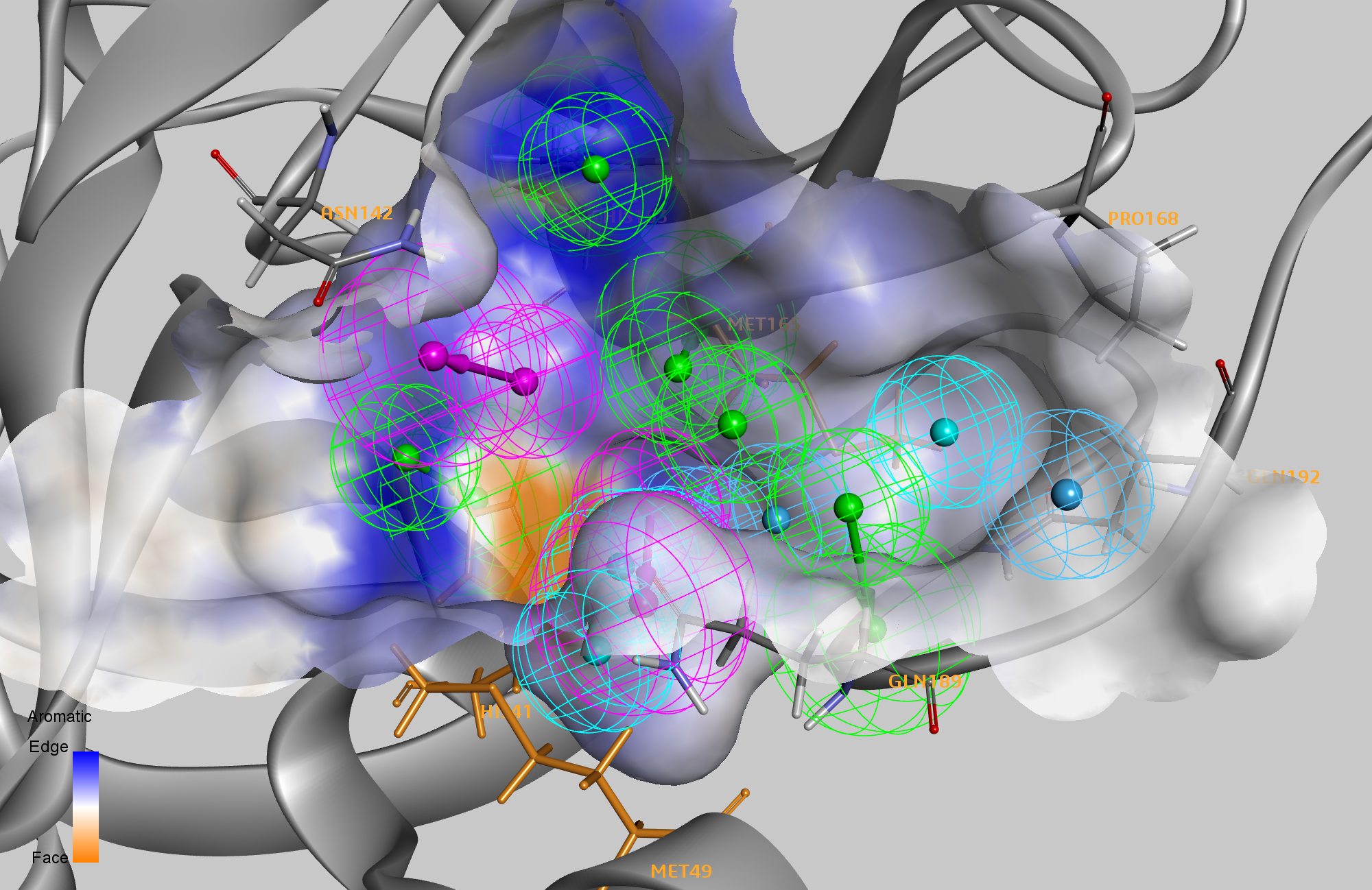
Ahora usamos este modelo de farmacóforo para realizar el cribado virtual de una biblioteca seleccionada que contiene 2650 medicamentos aprobados por la FDA que tienen múltiples confirmaciones de ligandos generadas previamente para una búsqueda rápida. Examinamos estos ligandos contra el farmacóforo de 14 características, con el requisito de que al menos una característica de cada uno de los dos grupos requeridos debía coincidir, así como al menos dos características de las ocho características restantes, para identificar un acierto. Esto es para permitir la exploración diversa de diferentes modos de unión de los ligandos, al tiempo que se asegura que solo se retengan las poses que pueden formar al menos dos interacciones con residuos significativos identificados en la mutagénesis de exploración de alanina. Básicamente, el algoritmo explora más de 12.000 posibles combinaciones de farmacóforos. Luego minimizamos los resultados de mejor ajuste para cada combinación de farmacóforos dentro de la proteína. Finalmente, determinamos las energías de unión de las poses optimizadas derivadas del farmacóforo usando CHARMm con un modelo de solvente implícito de Nacido Generalizado usando Volumen Molecular (GBMV). Este método es efectivamente un cálculo de acoplamiento molecular (como en nuestro blog anterior), pero aquí hemos utilizado nuestro modelo farmacóforo optimizado para restringir y filtrar los resultados acoplados.
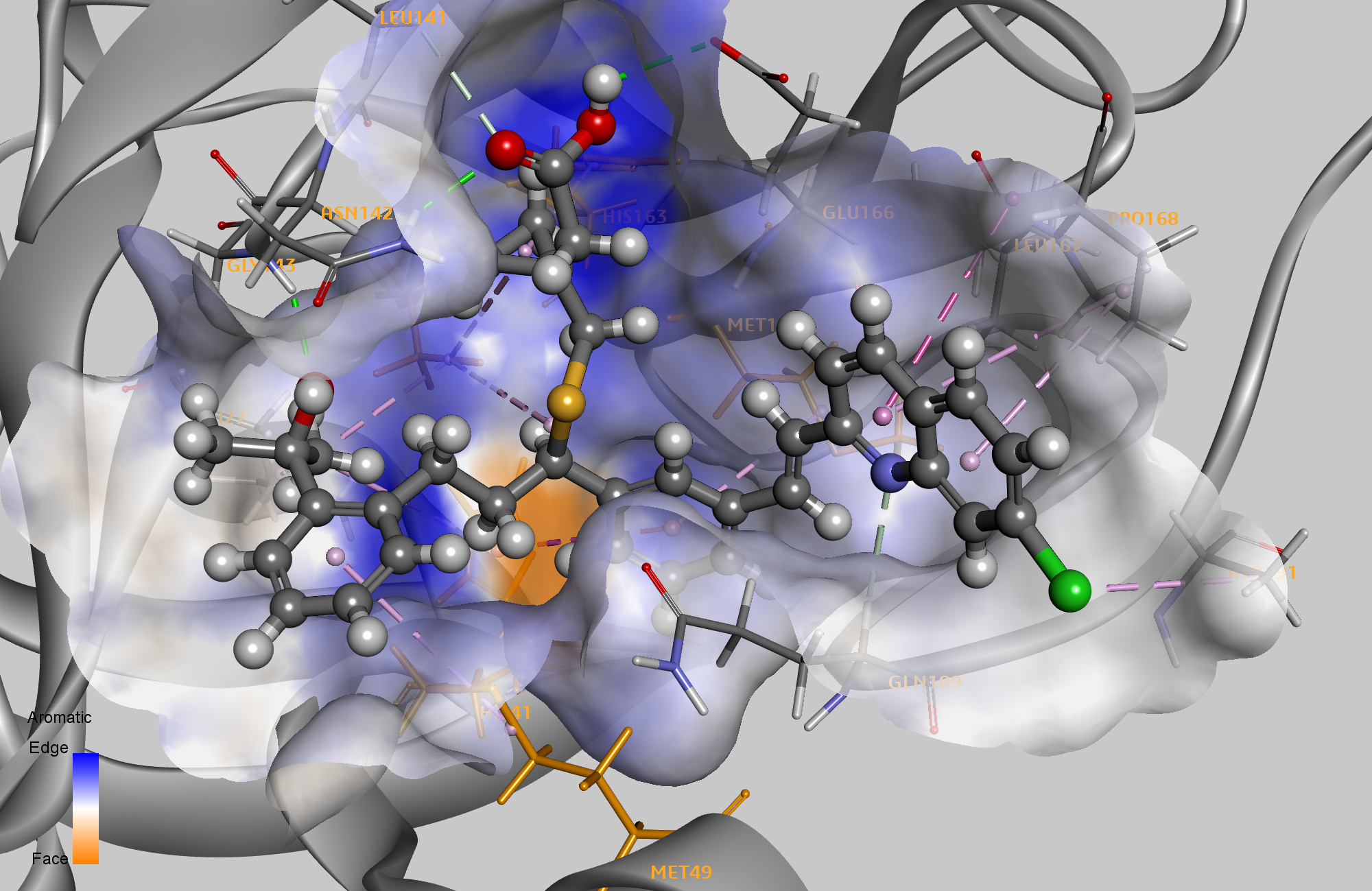
Calculamos las interacciones sin enlace de cada pose y las filtramos para enfocarnos solo en aquellas poses con interacciones con cuatro residuos clave:HIS41, MET49, CYS145 y MET165. HIS41, MET49 y MET165 fueron los tres residuos comunes a al menos cuatro complejos identificados anteriormente, y CYS145 es el segundo residuo en la díada catalítica significativa HIS41 / CYS145 que se encuentra en cada subunidad del homodímero de proteasa principal. Clasificamos los resultados con la energía libre de enlace calculada para identificar los diez candidatos más plausibles para una mayor exploración. El ritonavir fue el único ligando común identificado aquí y en el blog anterior. Ritonavir tuvo la séptima mejor energía libre de unión y anteriormente también ocupó el séptimo lugar en el estudio de acoplamiento. Actualmente, el ritonavir se encuentra en una serie de ensayos clínicos para COVID-19. 3
Video 1 :Pose con mejor puntuación para ritonavir en interacción con HIS41, MET49, SER144, CYS145, MET165, GLU166 y GLN189.
El ligando con mejor puntuación en el método presentado aquí fue Montelukast, un antagonista del receptor de cisteinil leucotrienos. Un artículo publicado recientemente ha planteado la hipótesis de su uso para limitar la progresión de la enfermedad en la infección por COVID-19. 4
Otros tres compuestos de máxima puntuación fueron los fármacos Telmisartan, Moexipril e hidroxicloroquina. El potencial de todos estos compuestos como medicamentos reutilizados para COVID-19 ha sido objeto de escrutinio recientemente, 5 siendo la hidroxicloroquina la que genera más controversias. Una diferencia adicional entre las diez listas de resultados principales de los dos métodos es la diversidad de los objetivos farmacológicos incluidos. Los diez primeros resultados de acoplamiento incluyen siete inhibidores de la proteasa del VHC o del VIH. La lista de resultados priorizados por farmacóforos incluye medicamentos con siete clases de objetivos diferentes.
Nuestro examen virtual derivado del farmacóforo produjo una lista de resultados priorizados que incluía varios medicamentos COVID-19 potenciales nuevos que no se identificaron en nuestro trabajo de acoplamiento anterior. Con este enfoque, demostramos la utilización de in silico mutagénesis por barrido de alanina como técnica útil para perfeccionar el modelo farmacóforo. También impusimos el requisito de que ciertas características estuvieran presentes para un ajuste durante el proceso de selección. Los resultados de estos dos estudios no son directamente comparables, no tanto por las diferencias en los algoritmos básicos, sino más bien por la implementación de diferentes estrategias de apoyo.
La breve comparación de resultados entre los dos métodos mostró poca superposición en las pistas en las que centrarse. Se podría decir que la similitud de ritonavir identificada por ambos métodos proporciona evidencia para respaldar la selección de este candidato para estudios adicionales. Estudios anteriores han demostrado que el uso de un consenso de funciones de puntuación de acoplamiento proteína-ligando mejora la identificación de posibles fármacos candidatos. 6 Por esta razón, los investigadores podrían utilizar los compuestos únicos con mejor puntuación de cada método para priorizar los compuestos para las pruebas experimentales.
En conclusión, el cribado virtual derivado del farmacóforo proporciona un método complementario y complementario para el acoplamiento, lo que contribuye a un consenso y una mayor confianza en la selección de candidatos para la validación experimental. Más allá de la urgencia de proporcionar un fármaco candidato para su reutilización, este método impulsado por farmacóforos también puede identificar ligandos que son más diversos para la posterior optimización de clientes potenciales.
Biologicos
- Parche de nicotina
- Inventario virtual e impresión 3D:la necesidad de seguridad
- C# para bucle
- C para bucle
- Nanofibras y filamentos para una mejor administración de fármacos
- Diseño de terapéutica generativa
- Reutilización de fármacos
- Tiny Pilot:Impresión 3D para máquinas virtuales basadas en kernel (KVM) sobre dispositivos IP
- Universal Robots lanza la conferencia virtual "más grande del mundo" para robots colaborativos
- Fabricación de PCB para 5G
- Los microrobots impresos en 3D son prometedores para la administración de medicamentos



