Reutilización de fármacos
Inhibición de la proteasa principal del SARS-CoV-2
El desarrollo de un nuevo fármaco es un proceso largo y costoso. Hoy en día, mientras el mundo se enfrenta a una pandemia, existe una necesidad urgente de identificar rápidamente los medicamentos que detengan la proliferación del virus. La reutilización de fármacos ofrece una alternativa atractiva a este largo proceso al intentar identificar si un fármaco que se sabe que es seguro en humanos podría utilizarse para tratar nuevas enfermedades.
Si bien el uso de dichos medicamentos reposicionados individualmente podría eventualmente no resultar en un beneficio clínico significativo, una combinación cuidadosa de medicamentos dirigidos a varias proteínas cruciales para la replicación y proliferación del virus podría ser muy efectiva, como fue el caso del VIH en la década de 1990. La pregunta urgente es ¿qué combinación sería más eficaz?
Aquí intentamos comprender la estructura del sitio activo de la proteasa del SARS-CoV-2, comparándolo con las estructuras existentes de la proteasa del SARS-CoV complejada con inhibidores micromolares, para que podamos comprender mejor las interacciones clave necesarias para crear un buen inhibidor. para la proteasa del SARS-CoV-2.
Luego, realizamos un experimento de detección virtual utilizando una biblioteca de medicamentos aprobados por la FDA para ver si se prevé que algunos de estos se unan a la proteasa. Examinamos cómo se predice que se unirán a la proteasa del SARS-CoV-2 y, por lo tanto, podrían usarse en una terapia de combinación.
Proteínas del SARS-CoV – 2
El genoma del SARS-CoV-2 de pacientes enfermos se aisló y secuenció rápidamente, proporcionando las secuencias de posibles objetivos proteicos. Estas proteínas comparten una gran similitud de secuencia con las proteínas del SARS-CoV e inicialmente los grupos de investigación comenzaron a construir modelos de homología. Ahora vemos que más y más de estas estructuras derivadas experimentalmente (rayos X y Cryo-EM) están disponibles.
Uno de los objetivos farmacológicos mejor caracterizados entre los coronavirus es la proteasa principal:Mpro, también llamada proteasa 3CL. 1 Junto con las proteasas similares a la papaína, esta enzima es esencial para procesar las poliproteínas que se traducen del ARN viral. 2 Escinde la cadena principal de aminoácidos en 11 sitios de escisión en la poliproteína grande 1ab (Fig. 1).
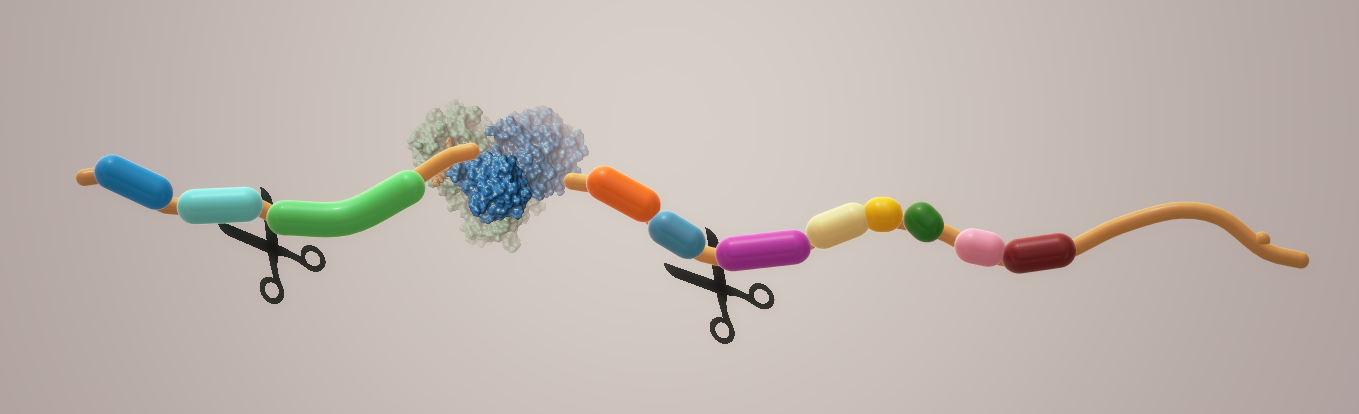
La inhibición de la actividad de esta enzima bloquearía la replicación viral. Dado que no se conocen proteasas humanas con una especificidad de escisión similar, es mucho menos probable que los inhibidores de este objetivo sean tóxicos y causen efectos secundarios.
Diseño de fármaco basado en estructura para reutilización de fármacos:proteasa principal
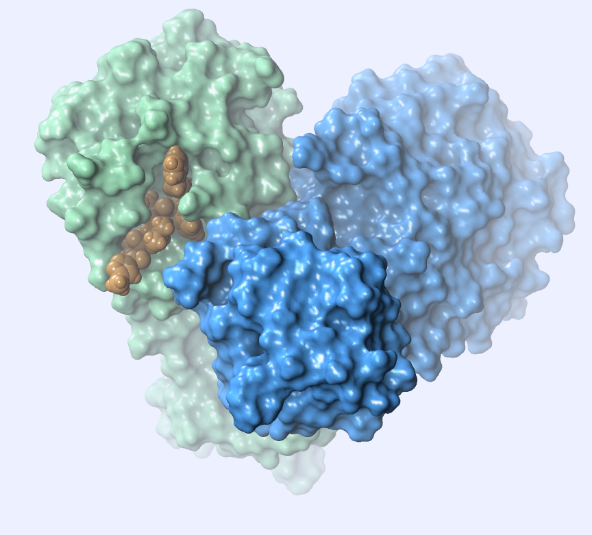
Esta proteasa principal es un homodímero (Fig. 2) y cada subunidad contiene una díada catalítica His41 / Cys145.
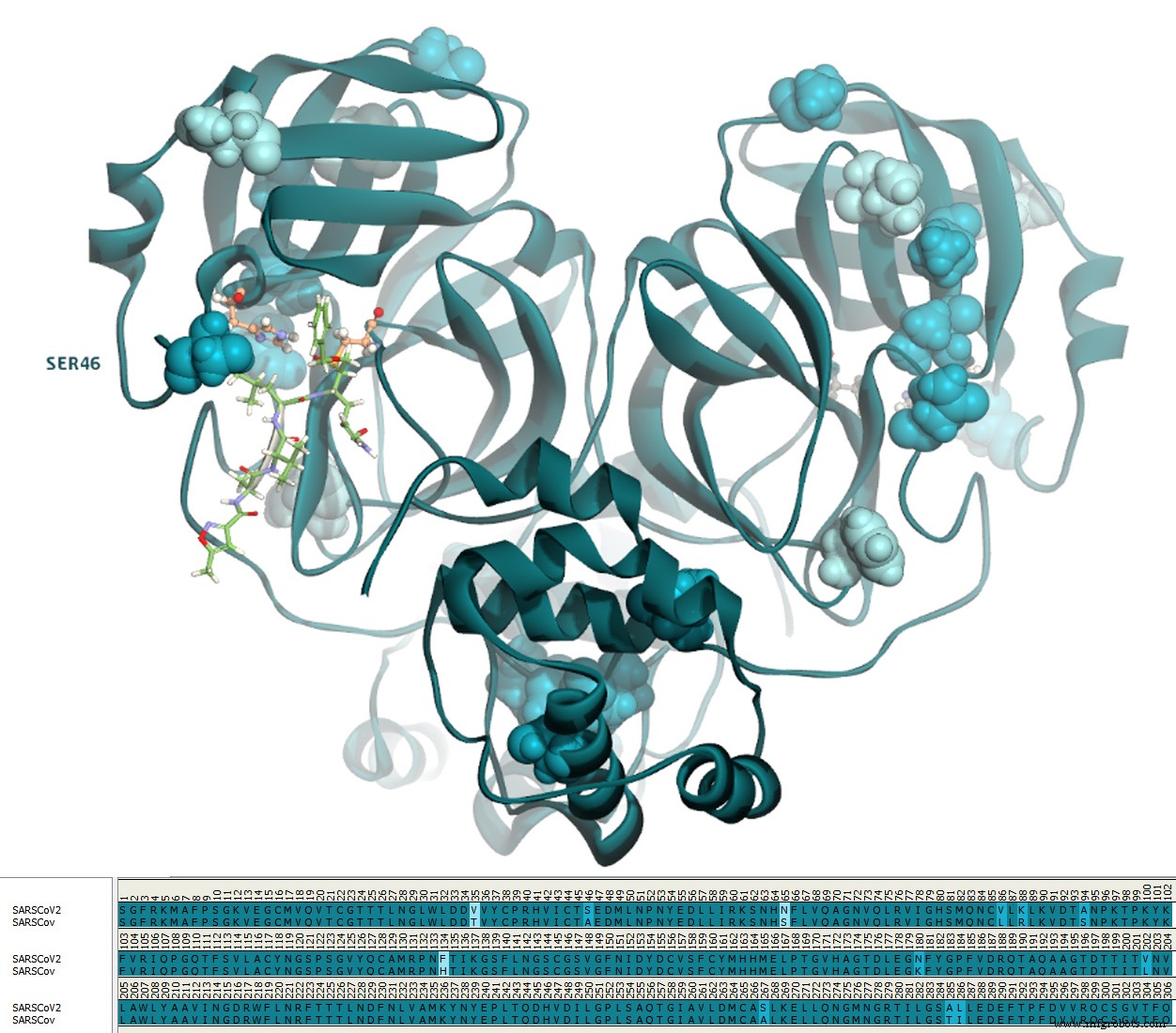
El SARS-CoV-2 comparte el 96,1% de identidad y el 99% de similitud con la proteasa del SARS-CoV. (Fig. 3) Solo hay una diferencia de aminoácidos en el sitio activo:el residuo 46 en el SARS-CoV-2 es una serina en lugar de una alanina en el SARS-CoV.
Hay muchas estructuras de SARS-CoV Mpro complejadas con ligandos disponibles en el PDB y los ligandos que tienen una afinidad de unión micromolar son todos ligandos covalentes.
Los sitios activos de la proteasa principal están muy conservados entre los coronavirus y suelen estar compuestos por cuatro sitios (S1 ′, S1, S2 y S4) 3 (Figura 4).
En el caso de las estructuras PDB 2ZU4 y 2GX4 para las cuales los ligandos tienen valores de Ki respectivamente de 0,038 µM y 0,053 µM, el tiol de la cisteína 145 en el sitio S1 'contacta con los inhibidores con un enlace covalente. En comparación con otros inhibidores con menor afinidad, esto parece ser importante para una mayor afinidad.
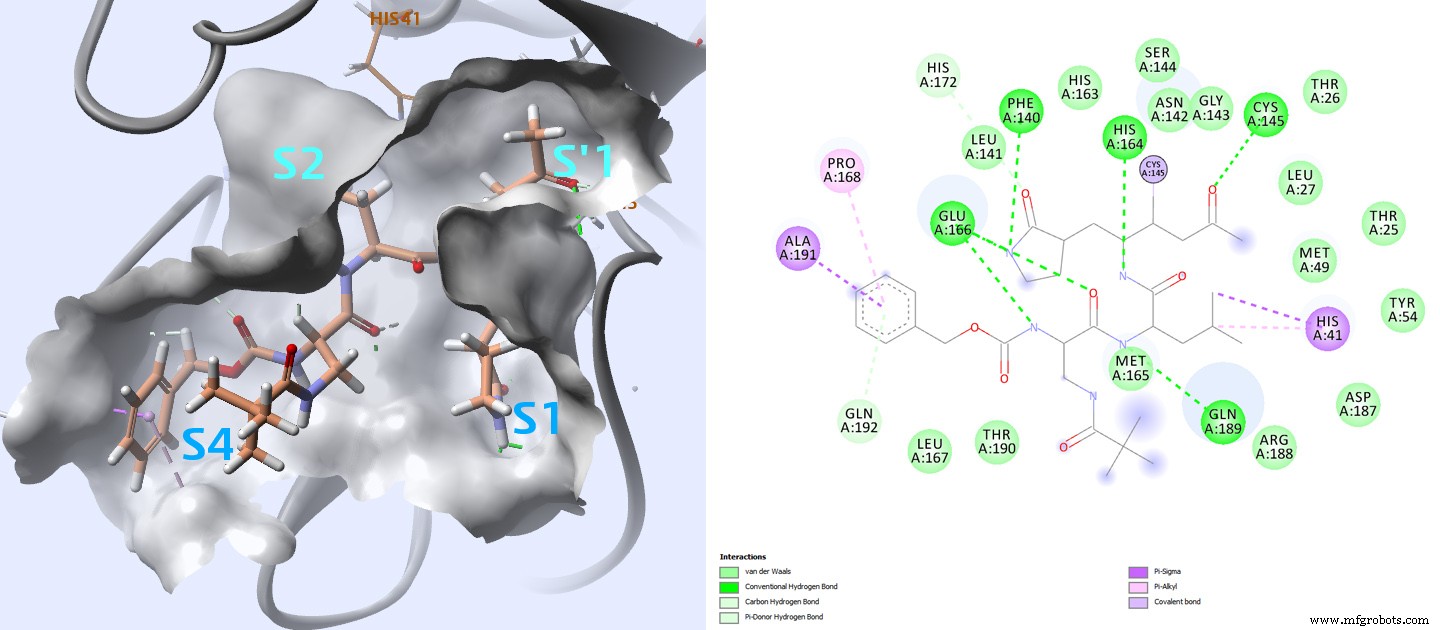
Un examen cuidadoso de cómo interactúan todos estos ligandos con esta proteasa puede proporcionar información sobre las interacciones clave que se deben monitorear al analizar los resultados del acoplamiento.
Proyección virtual
Aprovechando una estructura cristalina de alta resolución del dímero de proteasa del SARS-COV-2 en un complejo con un inhibidor N3 similar a un péptido unido covalentemente lanzado en febrero (6LU7) 4 , utilizamos esta estructura para realizar un experimento de detección virtual. El inhibidor N3 que se co-cristaliza se une en el bolsillo de unión al sustrato en una conformación extendida. (Figura 5)
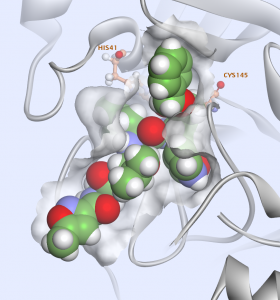
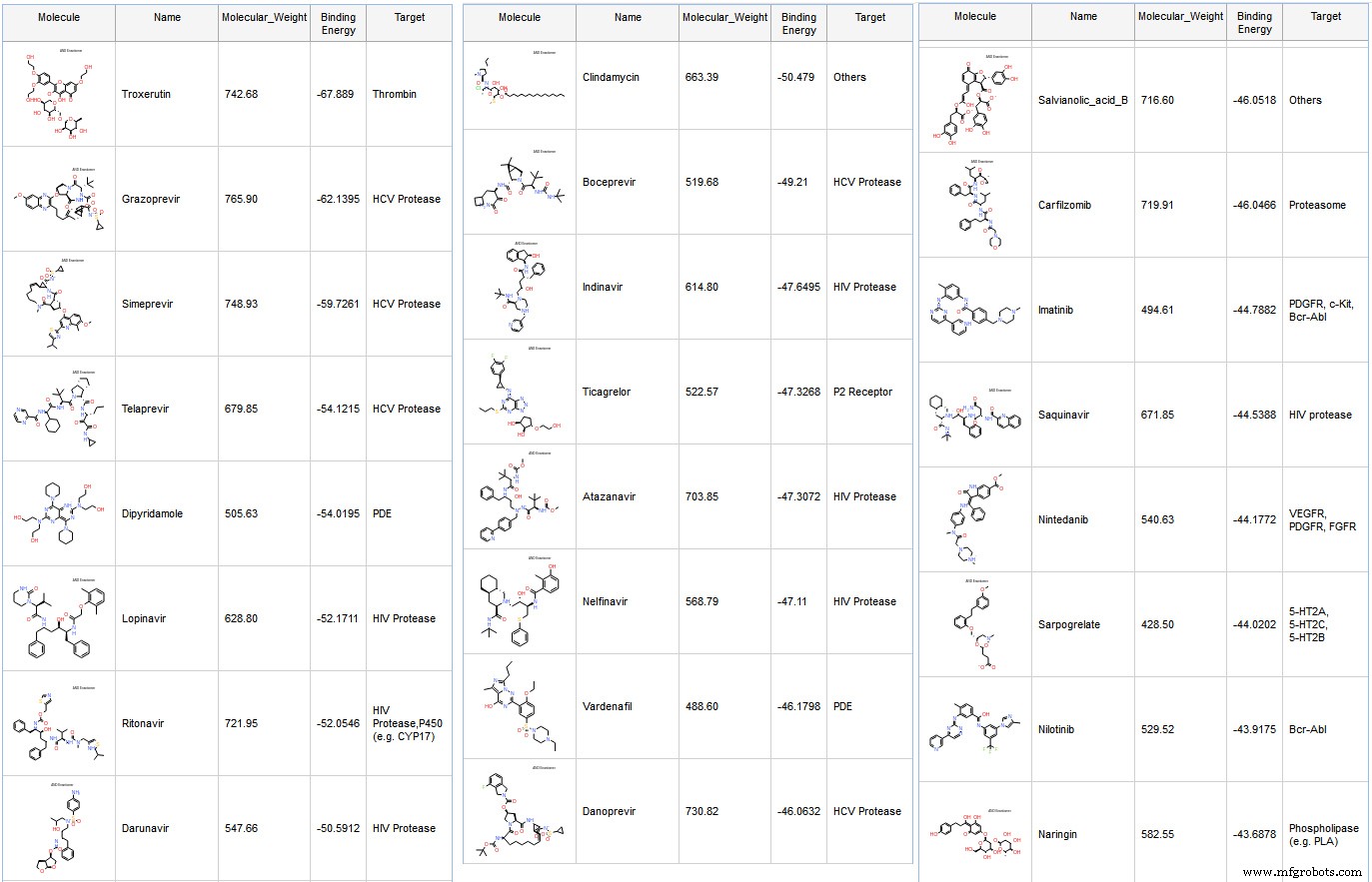
Comenzamos con una biblioteca que contiene medicamentos aprobados por la FDA con 2684 compuestos. Retenemos los compuestos con un peso molecular inferior a 800 kDa y realizamos el acoplamiento con GOLD del CCDC. Posteriormente, calculamos las energías libres vinculantes con disolvente implícito CHARMM y GBMV. Para cada pose, primero realizamos la minimización del ligando in situ con una esfera de 14 Å de diámetro para la flexibilidad del residuo y la entropía del ligando estimada al calcular la energía libre de unión. La mayoría de los compuestos acoplados son fármacos conocidos de proteasa del VHC y del VIH. (Figura 6)
Muchos compuestos hacen contactos en los 4 subsitios:S1, S'1, S2, S4 y algunos con HIS 41 y CYS 145, especialmente los inhibidores de la proteasa del VIH.
El compuesto mejor puntuado es la troxerutina, que es un flavonoide.
- Se ha demostrado que los flavonoides inhiben algunas proteasas 5 y recientemente IC 50 Los valores se calcularon a partir de las curvas inhibidoras dependientes de la dosis de herbacetina, rhoifolina y pectolinarina en el SARS-CoV. Los valores medidos fueron 33,17, 27,45 y 37,78 μM, respectivamente.
El dipiridamol (Fig.7), que es el quinto compuesto clasificado, se ha mencionado recientemente en una preimpresión Efectos terapéuticos del dipiridamol en pacientes con COVID-19 con disfunción de la coagulación, y se ha demostrado que suprime la replicación del VHC-19 a una CE50 de 100 nM in vitro.
Figura 7 :Representación 3D y mapa de interacción 2D de Dipiridamol en la proteasa principal. Existen interacciones con residuos catalíticos y con algunos de los residuos mencionados anteriormente para los inhibidores del SARS-CoV.
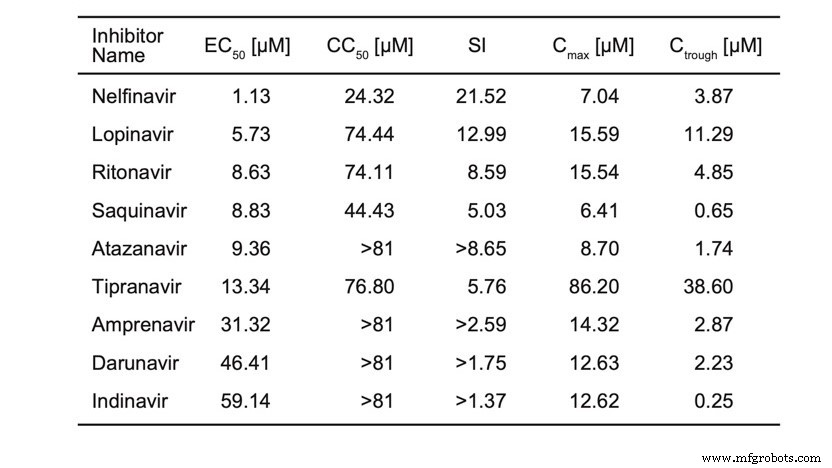
Una preimpresión reciente informó ensayos celulares de varios inhibidores de la proteasa del VIH:“El nelfinavir inhibe la replicación del coronavirus 2, síndrome respiratorio agudo severo, in vitro . ”
Estamos informando la tabla de la preimpresión (Tabla 1). Estos no son IC50, sino EC50, por lo que no dice qué tan bien se unen estas moléculas a la proteasa. Sin embargo, esto muestra que todos esos compuestos inhiben la replicación del SARS-CoV-2 y algunos son más activos que otros.
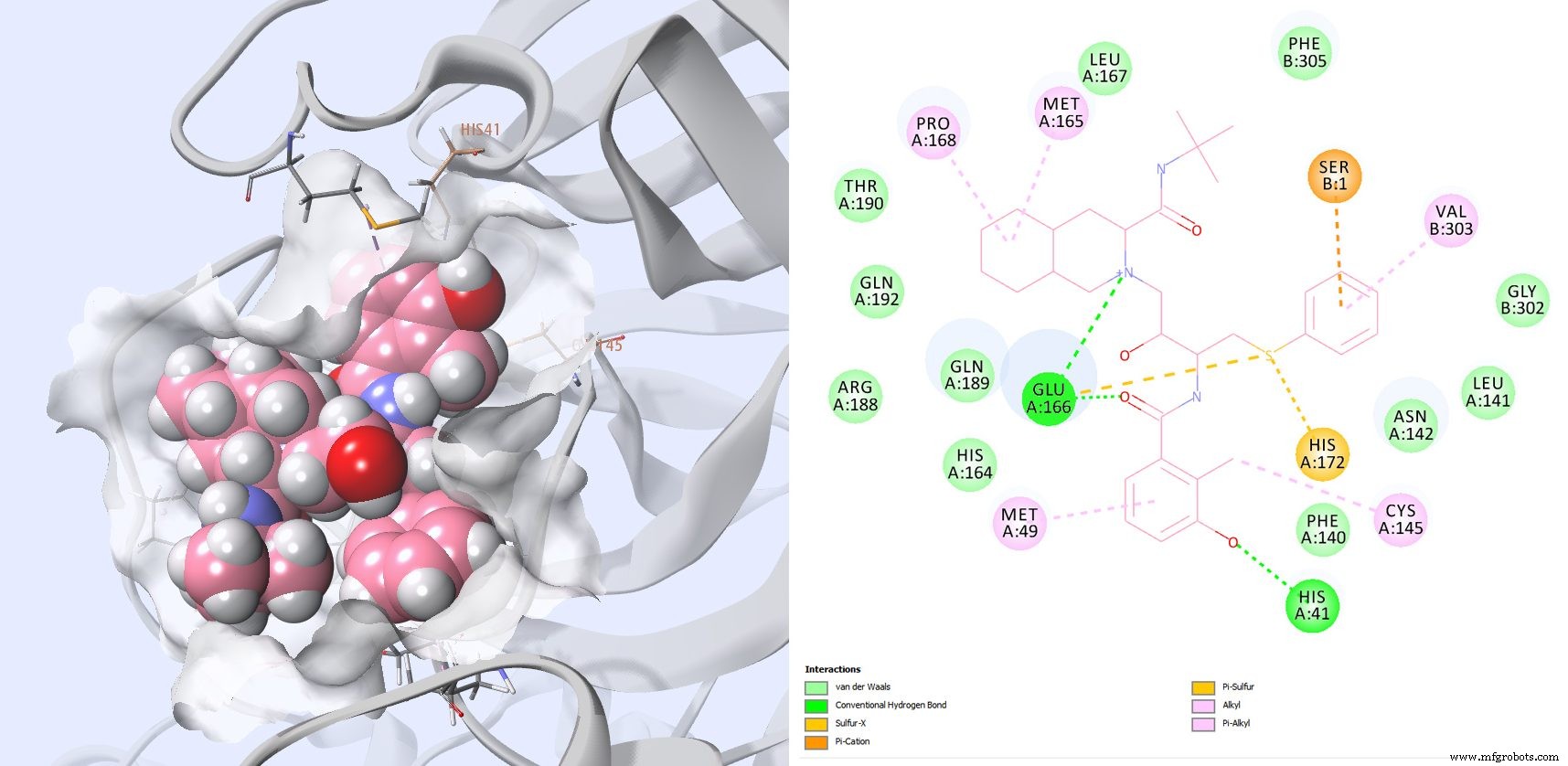
El ritonavir y el lopinavir interactúan con muchos residuos vistos previamente en otros compuestos que se dirigen a la proteasa del SARS-CoV (Fig. 8 y 9). Aunque se están realizando varios ensayos clínicos con una combinación de esas dos moléculas, lamentablemente no hay datos de IC50 disponibles para ritonavir o lopinavir en el SARS-CoV-2.
Figura 8 :Pose mejor puntuada para Ritonavir:y diagrama de interacción 2D que muestra que Ritonavir está interactuando con los dos residuos catalíticos:CYS145 y HIS41, pero también GLU166, PRO168 y GLN189 como se observa con otros inhibidores conocidos de SARS-CoV.
Figura 9 :Pose con mejor puntuación para Lopinavir:y diagrama de interacción 2D que muestra que Lopinavir está interactuando con los dos residuos catalíticos:CYS145 y HIS41, pero también GLU166, PRO168 y GLN189 como se observa con otros inhibidores conocidos de SARS-Co V.
El nelfinavir (Fig. 10) tiene una alta afinidad de unión prevista en nuestros cálculos y también se ha caracterizado contra COVID19 en una preimpresión reciente:"El nelfinavir es activo contra el SARS-CoV-2 en las células Vero E6".
Una segunda preimpresión muestra que Atazanavir es activo en las células infectadas con SARS-CoV-2:"Atazanavir inhibe la replicación del SARS-CoV-2 y la producción de citocinas proinflamatorias".
Una estructura PDB 6W63 que contiene un nuevo inhibidor para SARS-CoV-2 se publicó después de que comenzamos este trabajo (Fig. 11). No hay información publicada sobre la afinidad de unión de este compuesto al SARS-CoV-2 Mpro. Las cadenas laterales en el sitio de unión están casi en la misma orientación que para 6LU7. Acoplamos nuestros compuestos y los volvimos a calificar con enfoques MMGBSA como anteriormente y encontramos resultados muy similares.
Figura 11 :Estructura PDB 6W63:Proteasa principal SARS-CoV-2 en complejo con inhibidor X77.
Perspectivas
El sitio de unión de la proteasa del SARS-CoV-2 es muy grande, contiene cuatro subsitios y puede acomodar muchos ligandos diferentes con una afinidad de unión moderada. Como se explica aquí, la detección virtual es una herramienta útil para identificar posibles inhibidores y pudo darnos una explicación estructural de cómo los posibles inhibidores podrían estar interactuando con la proteasa.
Sin embargo, todas estas hipótesis requieren confirmación mediante evidencia experimental, como la medición de IC50, para construir modelos que sean más robustos.
Aquí no hemos realizado un acoplamiento covalente, pero como los compuestos más activos contra la proteasa del SARS-CoV eran covalentes, esto podría ser un requisito de inhibidores fuertes. Los inhibidores covalentes también muestran ventajas en comparación con los inhibidores reversibles, como tener una fuerte afinidad por el objetivo y una vida de acción prolongada en los pacientes.
Para que un fármaco sea activo en un paciente, debemos considerar otros aspectos farmacológicos como la farmacocinética. Será necesario evaluar la capacidad de estos fármacos para alcanzar las concentraciones plasmáticas y pulmonares objetivo después de la dosificación aprobada en humanos.
No estaba dentro del alcance de este trabajo comentar sobre otras investigaciones de diseño basadas en estructuras, ya que esto no sería oportuno para la epidemia actual. Nuestro propósito era evaluar terapias fácilmente utilizables para posibles fármacos candidatos contra COVID19.
Nos gustaría agradecer al Cambridge Crystallographic Data Center (CCDC) por el permiso para usar GOLD para la proyección virtual en este trabajo. GOLD ha demostrado tener éxito en la detección virtual, la optimización de clientes potenciales y la identificación del modo de unión correcto de moléculas activas. Una interfaz para GOLD está disponible dentro de Discovery Studio.
ENTRENAMIENTO DE PILOTO DE TUBERÍAS EN LÍNEA
El modelado y la simulación proponen nuevos conocimientos para el SARS-CoV-2
Biologicos
- Parche de nicotina
- China establecerá un vínculo de patente
- La cadena de suministro de medicamentos de EE. UU. en crisis:soluciones a la escasez
- COVID-19 ha revelado fallas fatales en la cadena de suministro de medicamentos de EE. UU.
- Cómo Blockchain podría ayudar a combatir las drogas falsificadas
- Aplicación de Blockchain y Machine Learning a las cadenas de suministro de fármacos
- Prueba todo en uno para el control de la COVID-19
- Un nuevo efecto fotónico podría acelerar el desarrollo de fármacos
- Smartwatch realiza un seguimiento de los niveles de medicación
- 5 Ws del sensor SARS-CoV-2 RapidPlex
- Los microrobots impresos en 3D son prometedores para la administración de medicamentos



